非小细胞肺癌(non-small cell lung cancer,NSCLC)是一种常见的肺癌类型,约占所有肺癌病例的85%[1]。在过去的几十年里,针对NSCLC的研究取得了显著进展,对其发病机制、诊断方法、治疗策略和预后因素都有了深入了解[2]。通过T细胞受体(T-cell receptor,TCR)组库分析可以了解NSCLC免疫应答和免疫监视的特征,从而揭示T细胞克隆多样性、免疫应答的活性以及肿瘤逃逸机制[3],对于指导免疫治疗策略和预测患者预后具有重要意义。TCR组库分析可以评估肿瘤组织中T细胞克隆的多样性[4],以反映免疫系统对抗肿瘤的能力;高克隆多样性通常与较好的预后结果相关[5]。同时,通过比较肿瘤组织和正常组织中的T细胞克隆组成,可以了解肿瘤免疫应答的活性程度,较高T细胞克隆丰度和活性可能意味着更强的抗肿瘤免疫应答[6]。此外,TCR组库分析可以识别免疫治疗相关的T细胞克隆群体或特定TCR序列,以协助发展个体化的免疫治疗策略[7],筛选免疫反应较强的TCR用于患者治疗。
目前,免疫组库测序技术可以较完整地获取患者TCR组库多样性等信息,以促进NSCLC免疫治疗发展[8]。免疫检查点抑制剂已成为NSCLC的主要治疗策略之一[9]。这些抑制剂通过阻断肿瘤细胞与免疫细胞之间的抑制信号,恢复T细胞对肿瘤的免疫攻击能力。ROBERT等[10]研究发现TCR组库多样性变化可预测免疫检查点疗法的有效性。此外,T细胞受体工程化T细胞(T cell receptor-engineered T cells,TCR-T)疗法[11]、肿瘤疫苗[12]等方法也逐步应用于NSCLC。ZHU等[13]的研究将小鼠中特定TCR基因重定向至人类T细胞,并通过体外实验观察到该基因导入对肝癌细胞的杀伤作用,为TCR-T疗法提供了理论基础。本研究组已开发了iTCep[14],可用于预测给定抗原肽的TCR结合特异性,为肿瘤疫苗研发提供参考。
目前,已存在一些免疫组库分析工具(如MiXCR[15]、Imonitor[16]等),但能实现从原始TCR组库测序数据到下游组库特征分析以及结果可视化完整流程的软件工具还很欠缺。为此,本研究拟构建一套系统的TCR组库分析流程以探究肿瘤相关特征和发掘潜在肿瘤标志物,涵盖从原始数据到质控、过滤、TCR序列的识别和提取等上游分析部分,以及组库克隆分布、克隆分型、V-J基因共享性、互补决定区3(CDR3)分布特征以及克隆追踪等下游分析流程,并将其用于分析NSCLC患者的肿瘤相关特征。通过对免疫组库的深入研究和分析,可以更好地理解免疫系统的工作原理,为免疫治疗和药物研发提供有价值的信息,也为后续封装成软件包并提供网页工具打好前期基础。
1 资料与方法
1.1 TCR序列原始数据准备
从NCBI BioProject数据库获取NSCLC(PRJNA544699)和甲状腺乳头状癌(papillary thyroid carcinoma,PTC;PRJNA664708)患者样本TCR测序数据集,用于流程方法测试及生物学特征案例分析和验证。其中,NSCLC数据集包括72例患者的220个肿瘤区域样本、64个匹配的非肿瘤肺组织样本和70个外周血单核细胞样本测序原始数据[17];共计包含704个样本,包括T细胞受体α链(T-cell receptor α chain,TRA)样本353例,T细胞受体β链(T-cell receptor β chain,TRB)样本351例。PTC数据集包括2例PTC患者肿瘤样本TCR测序数据[18]。
1.2 数据预处理
使用SRA Toolkit工具将SRA格式原始数据转化为fastq格式文件,作为流程起始输入文件。通过FastQC质量检查,使用Trimmomatic程序去除接头序列、低质量序列、重复序列和低质量克隆,对数据进行过滤以提高数据的准确性。
1.3 序列比对
基于MIXCR分析流程进行TCR序列比对。使用species参数设置物种为homo sapiens,使用receptor type参数设置数据类型为TCR,将质控后测序数据与MiXCR内置参考V、D、J和C基因片段数据库进行比对。
1.4 中间结果重注释
为方便下游分析,对前述分析结果进行重注释,筛选出cloneCount(clone数量)、cloneFraction(clone比例)、allVAlignments(V区基因比对)、allDAlignments(D区基因比对)、allJAlignments(J区基因比对)、nSeqCDR3(CDR3区核苷酸序列)、aaSeqCDR3(CDR3区氨基酸序列)、refPoints(序列比对参考位置)等结果。
1.5 克隆分布及克隆分型分析
使用Shannon-Wiener指数、Chao1指数以及Pielou均匀度计算TCR组库中的多样性(diversity)、丰富度(richness)与克隆性(clonality)。
免疫组库的多样性使用Shannon-Wiener 指数[19]进行评估,该指数是反映免疫组库多样性的经典指标。计算公式如下:
其中,s表示物种数目,Pi 表示该物种个体数占总个体数的比例。
免疫组库的丰富度使用Chao1指数[20]计算。Chao1指数是丰富度的非参数渐近估计量,计算中低频克隆性权重较高,能较大程度反映免疫组库克隆型丰富度。计算公式如下:
其中,Sobs 表示样本中观察到的克隆型数目,
克隆性指的是一组具有相同或相似免疫受体,即源自同一克隆的免疫细胞。克隆性是反映免疫组库中高克隆型扩增程度的指标,可用于评估免疫系统中的克隆扩增情况和免疫应答的多样性。克隆性可用Pielou均匀度指数[21]计算:
其中,Pi 表示该物种个体数占总个体数的比例。
为分析免疫组库克隆程度在不同组织间的表达差异,将肿瘤、癌旁和外周血组织根据克隆扩增数目分为低、中、高克隆型,克隆扩增数目区间分别为1~100、101~1 000和1 001及以上。此外,为分析不同年龄对免疫组库克隆分布的影响,将样本按年龄进行分组,其中64岁及以下为中年组(mid),65~79岁为老年组(old),80岁及以上为高龄老人组(eldest)。
克隆分布和克隆分型分析通过R语言(4.2.0版)计算,不同分组样本分布频率比较分析采用t检验, P<0.05表示差异有统计学意义。使用ggsignif包(0.6.4版)对P值进行布尔值转换,P≥0.05表示差异不显著,P<0.05表示差异显著,P<0.01表示差异非常显著,P<0.001表示差异极显著。使用ggplot2包(3.4.2版)进行数据可视化。
1.6 V-J基因共享性分析
V-J基因共享性即V、J基因各连接点的频率,可用于探索V、J基因间的关联性对其在不同组织间表达差异的影响。本研究选取Pi =0.001为频率阈值对V-J基因重要共享配对进行筛选,使用R语言(4.2.0版)进行数据处理,并使用circlize包(0.4.15版)进行可视化。在计算V-J基因共享性时,一般需考虑克隆扩增对其频率的影响。本研究为探究肿瘤低频克隆型对V-J基因共享性的影响,采用去克隆扩增权重的V-J基因共享性分析方法。此方法在进行频率计算前使用克隆分型替换克隆扩增数目,以去除克隆扩增权重的影响,能更好地反映低频克隆型的变化特征。
1.7 CDR3分布特征分析
正常情况下,机体免疫组库中T细胞表现为多克隆状态,其CDR3呈均匀的高斯分布。疾病状态下,由于抗原的持续刺激,会导致某一类型的T细胞偏好扩增,从而引起CDR3分布特征的变化。因此,本研究使用VDJtools对免疫组库中TCR可变区基因V以及氨基酸克隆型丰度分布进行分析,以探究克隆型偏态分布与NSCLC的关系。
1.8 克隆追踪
克隆追踪分析从输入的免疫组库中选择最丰富的克隆型,并对所有免疫组库进行批量追踪,可用于监测癌症免疫学中克隆型频率的变化。此外,以提供的核苷酸或氨基酸序列作为参数,可用于跟踪特定克隆型序列。使用immunarch包(0.67版)对数据进行克隆追踪分析,并使用ggplot2包(3.4.2版)进行数据可视化。
2 结果
2.1 克隆分布与克隆分型
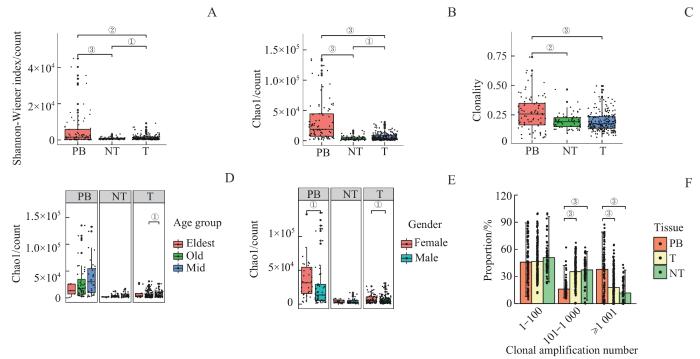
图1
图1
NSCLC克隆分布及克隆分型分析
Note: A. Shannon-Weiner index of sample TCRβ chain tumor, peripheral blood and paraneoplastic tissue. B. Abundance of TCRβ chain among different tissues. C. Clonality of TCRβ chain among different tissues. D. Difference in abundance of TCRβ chain among different age groups in different tissues, respectively. E. Differences in the abundance of TCRβ chains in different tissues by gender. F. Proportion of different tissues of TCRβ chains in different clonal typing. T—tumor; PB—peripheral blood; NT—normaol tissue (paraneoplastic tissue). ①P<0.05, ②P< 0.01, ③P<0.001.
Fig 1
NSCLC clonal distribution and clonal proportion analysis
克隆分型分析结果显示:低克隆分型中,3组之间无显著差异;中克隆分型中,外周血显著低于癌旁和肿瘤组织;高克隆分型中,外周血显著高于癌旁和肿瘤组织(图1F)。该结果提示NSCLC可能引起患者外周血细胞高克隆,激活血液中相关分子的生物学功能,从而形成全局性免疫反应。
2.2 V-J基因共享性
图2
图2
TRA 和 TRB V-J基因共享性
Note: A. V-J gene sharing in paraneoplastic tissue with the degree of sharing of linkage thickness response of patient 1. B. V-J gene sharing in peripheral blood of patient 1. C. V-J gene sharing in peripheral blood of patient 2.
Fig 2
V-J gene sharing of TRA and TRB
2.3 CDR3分布特征
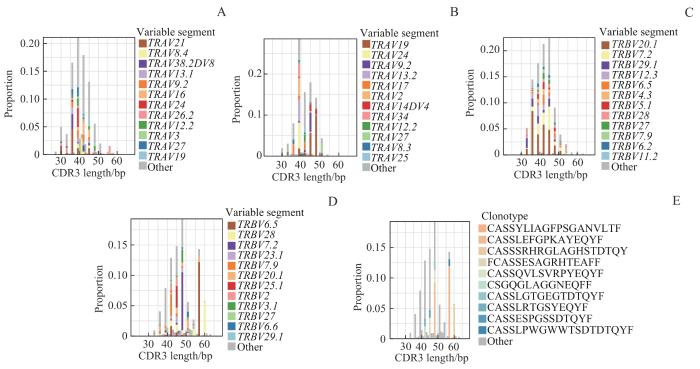
图3
图3
NSCLC患者 TRA 和 TRB V基因及高频克隆型CDR3分布特征
Note: A. CDR3 distribution characteristics of tumor TRA V gene of patient 1. B. CDR3 distribution characteristics of tumor TRA V gene of patient 2. C.CDR3 distribution characteristics of tumor TRB V gene of patient 2. D. CDR3 distribution characteristics of peripheral blood TRB V gene of patient 1. E. Distribution characteristics of high-frequency clonotype CDR3 in peripheral blood of patient 1.
Fig 3
Distribution characteristics of TRA and TRB V gene and high frequency clonotype CDR3 in NSCLC patients
2.4 NSCLC高频克隆型克隆追踪
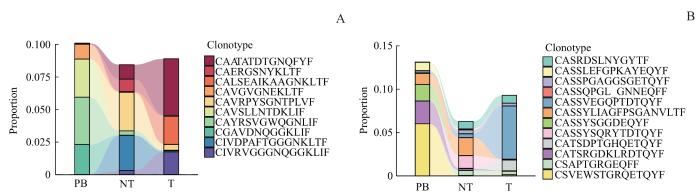
通过CDR3分布特征分析,发现了患者不同组织的高频扩增克隆型。为进一步探究这些高频克隆型在肿瘤发生过程中的动态变化,分别选取了TCRα链和TCRβ链中扩增程度最高的前10种与前12种克隆型进行克隆追踪分析。结果发现,CAATATDTGNQFYF、CIVRVGGGNQGGKLIF和CASSVEGQPTDTQYF在癌旁组织表达量较低,在肿瘤组织中表达量显著增加(图4),提示上述克隆型可作为潜在的NSCLC肿瘤相关抗原识别参考序列。CAVSLLNTDKLIF、CGAVDNQGGKLIF和CSVEWSTGRQETQYF在外周血中高表达,在癌旁和肿瘤组织中表达量显著降低;CAVRPYSGNTPLVF、CASSYLIAGFPSGANVLTF和CASSYSQRYTDTQYF在癌旁组织高表达,在肿瘤组织中的表达量显著降低(图4),提示NSCLC可能抑制上述克隆型的正常表达,进而影响相关生物学功能。CALSEAIKAAGNKLTF和CATSDPTGHQETQYF在癌旁组织几乎不表达,在肿瘤组织中表达量显著增加(图4),提示上述克隆型可作为潜在的NSCLC肿瘤新抗原识别参考序列。
图 4
图 4
NSCLC患者高频克隆型克隆追踪
Note: A.Dynamics of the top ten clonotypes with the highest degree of amplification in the TCRα chain among different tissues. B. Dynamics of the top twelve clonotypes with the highest degree of amplification in the TCRβ chain among different tissues.
Fig 4
High-frequency clonotype clone tracking in NSCLC patients
2.5 去克隆扩增权重V-J基因共享性分析
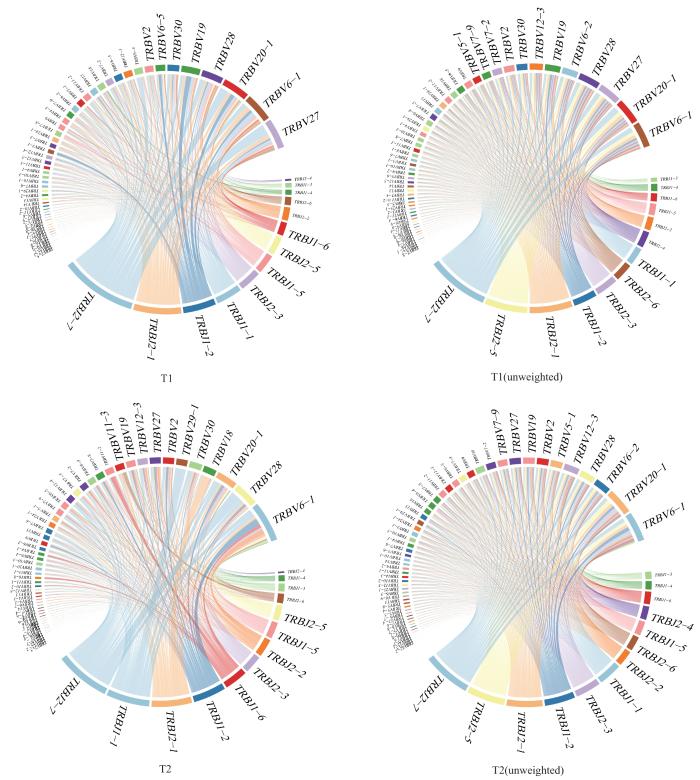
克隆分布结果显示,与外周血相比,患者肿瘤与癌旁组织在多样性、丰富度以及克隆性上均呈显著低表达,提示肿瘤中可能存在较多低频克隆突变。为进一步探索肿瘤相关低频克隆突变,选取2例PTC样本进行去除克隆扩增权重的V-J基因共享性分析。结果发现,患者1与患者2原本表达较低的TRBJ2-5基因在去除克隆扩增权重后表达频率显著增加(图5),提示该基因可能作为肿瘤免疫反应中潜在的低频关键基因。
图 5
图 5
PTC肿瘤组织克隆扩增与克隆型基因共享性对比
Note: T1 and T2 are V-J gene sharing when clonal amplification weights were not removed for patient 1 and patient 2, and T1 (unweighted) and T2 (unweighted) are V-J gene sharing when clonal amplification weights were removed for patient 1 and patient 2.
Fig 5
Comparison of clonal amplification and clonotype gene sharing in PTC
3 讨论
免疫组库的研究可以为免疫学、生物学、医学和药物研发等领域提供很多有价值的信息。免疫组库的分析可以揭示生物体在不同免疫状态下抗体的多样性和特征,包括抗体的结构、亲和力、特异性、功能等。这些信息可以帮助我们更好地理解免疫系统的工作原理,以及在疾病和免疫治疗中的应用。为此,本研究建立了一套系统免疫组库分析流程,包括从原始数据到质控、过滤、TCR及BCR序列识别与提取等上游分析部分到免疫组库多样性分析、基因扩增偏好、V-J基因共享性、CDR3长度分布等下游分析及数据可视化流程。
基于建立的免疫组库分析流程,本研究分析了一套NSCLC数据集,其中包含72例患者的220个肿瘤区域、64个匹配的非肿瘤肺组织样本和70个外周血单核细胞样本。结果发现,NSCLC肿瘤和癌旁组织中,TCR组库多样性、丰富度和克隆性降低。克隆分型分析中,外周血高克隆分型显著高于肿瘤和癌旁组织,提示NSCLC可能引起患者外周血细胞高克隆,激活血液中相关分子的生物学功能从而形成全局性的免疫反应。
为探究V-J基因在不同组织间表达的差异,本研究从NSCLC的TCRα链和TCRβ链分别挑选了4例患者分析其V-J基因共享性。结果发现TRAV21和TRAJ53基因,TRAV38-2DV8、TRAV13-2和TRAJ42基因高度共享,提示这些高共享性的V-J基因可能对NSCLC具有协同调控作用。在CDR3长度分布特征分析中,我们发现了TRAV21和TRBV6.5的非正态高克隆扩增,提示TRAV21和TRBV6.5的高克隆扩增可能是由NSCLC引起的,即上述基因可作为NSCLC的潜在肿瘤标志物。在高频克隆型CDR3特征分布分析中,我们提取了10个TRA高频克隆型和12个TRB高频克隆型用于克隆追踪分析,并在克隆追踪分析中发现了潜在的NSCLC肿瘤相关抗原和新抗原结合CDR3参考序列。最后,为探索肿瘤相关低频基因,本研究选取2例PTC样本分析去除克隆扩增权重后克隆型间的V-J基因共享性,结果发现,患者1与患者2原本表达较低的TRBJ2-5基因在去除克隆扩增权重后表达频率显著增加,提示该基因可作为肿瘤免疫反应中潜在的低频关键调控基因。此外,已有研究[25]发现乙型肝炎患者中同样存在TRBJ2-5基因的上调,进一步验证了本研究建立的TCR组库的可行性。
综上所述,本研究建立了一套系统的免疫组库分析流程,用于分析NSCLC的肿瘤相关特征,观察到NSCLC患者不同组织间克隆分布程度的差异和特定V-J基因的高共享性表达;随后,通过分析非正态克隆扩增的V基因和高频克隆型,发现了NSCLC潜在肿瘤标志物和抗原结合CDR3参考序列;最后,通过PTC患者去权VJ基因共享性分析发现了TRBJ2-5为潜在的低频关键调控基因。该免疫组库分析流程和肿瘤TCR组库特征分析方法还可以扩展到其他肿瘤和疾病。本研究组已建立流程预测病原体抗原蛋白序列中多肽疫苗候选表位[26]。本研究流程可从适应性免疫系统特异性识别抗原肽的角度,通过预测高频CDR3序列为抗原表位识别工作提供免疫学参考,从而进一步评估T细胞表位被免疫细胞捕获并引发免疫应答的潜力。总体上,本流程可应用于如下场景:比较不同肿瘤或疾病间的TCR克隆型频率,发现潜在的广谱或特异的肿瘤标志物;分析结合特定抗原如肿瘤新抗原的CDR3结合序列特征,建立TCR-抗原识别预测模型;与其他组学数据如RNA-seq、单细胞测序数据联合,挖掘组学中的免疫组库特征,进行免疫学机制研究。
作者贡献声明
谢鹭、赵卓明和鲁曼曼参与了研究设计与流程构建;谢鹭、赵卓明和刘振浩参与了论文写作和修改;张钰协助数据分析,赵卓明和许林锋参与了编程。所有作者均阅读并同意了最终稿件的提交。
AUTHOR's CONTRIBUTIONS
XIE Lu, ZHAO Zhuoming, and LU Manman participated in research design and process construction. XIE Lu, ZHAO Zhuoming, and LIU Zhenhao contributed to the writing and revision of the paper. ZHANG Yu assisted in data analysis. ZHAO Zhuoming and XU Linfeng were involved in programming. All the authors have read the last version of paper and consented for submission.
利益冲突声明
所有作者声明不存在利益冲突。
COMPETING INTERESTS
All authors disclose no relevant conflict of interests.
参考文献