癌睾丸抗原(cancer-testis antigen,CTA)是一类特异性表达于睾丸和肿瘤中的抗原,已应用于肿瘤治疗[1-2]。由于CTA具有局限表达于睾丸且在肿瘤中高表达特征,有研究者采用生物信息分析等方法鉴定癌睾丸基因(cancer-testis gene,CTG)[3-4]。这些技术不能确定基因表达的蛋白质是否具有免疫原性,故这些基因连同之前的CTA合称为CTG。在2009年,研究者分析了已鉴定的CTG,将其信息整理成数据库CTdatabase[3]。CTdatabase数据库中所有内容都经过了相关领域内专家的注释,包括基因名称、基因组位置等。此外,研究者根据CTG是否分布在X染色体上,将CTG分成2类:分布在X染色体上的CTG(CT-X)和分布在常染色体上的CTG(Non CT-X)[5-6]。
本研究采用生物信息学技术,研究CTG的分布特征和表达规律。通过多个数据库的联合分析,重新对CTG进行筛选;分析睾丸精子发生过程中的基因表达谱,鉴定出与CTG相关的GEP,并分析其在各种肿瘤类型中的活跃程度及其与患者预后的关联性。
1 材料与方法
1.1 CTG的筛选
1.1.1 GTEx转录组数据的获取
GTEx(Genotpye-Tissue Expression)项目致力于提供给研究者用于研究遗传变异与基因表达调控的资源。本研究中使用的是移除个体水平身份信息的基因表达数据,即正常组织的转录组数据(
1.1.2 HBM转录组数据的获取
Illumina Human Body Map(HBM)项目包含16种组织类型的转录组数据。为了与GTEx数据库的转录组数据相匹配,从HBM下载原始的测序短读测序片段(reads)数据(FASTQ格式存储),并用GTEx数据库的分析流程对HBM的数据进行比对和基因定量。HBM的FASTQ原始数据存储于EMBL-EBI数据库,访问号为E-MTAB-315。
1.1.3 TCGA转录组数据的获取
通过TCGAbiolink[8]检索TCGA数据库中33种癌症类型样本的转录组,并将用于gdc-client下载的索引保存至gdc_manifest.txt文件中。之后,用gdc-client下载33种癌症类型样本的转录组。本研究中使用的数据版本为v33.1,发布时间为2022年5月31日。
1.1.4 基因表达特异性分析
使用基因特异性度量(specific measure,SPM)衡量基因在睾丸组织中的特异表达情况[4]。具体定义如下:基因在不同组织中的表达矩阵表示为
其中,
基因特异性表达是指基因仅在某个组织中表达,而不在其他组织中表达。因此当基因
根据
1.1.5 筛选CTG
CTG的主要特征是在正常的睾丸组织中高表达,并且在肿瘤中也高表达。因此,可以分成两步筛选:先筛选睾丸特异表达基因(testis specific gene,TSG),再基于TSGs在肿瘤中的表达情况筛选出CTG。
使用正常转录组数据(GTEx数据库和HBM数据库)和肿瘤转录组数据(TCGA数据库)筛选CTG。首先,筛选TSG,具体步骤如下:①计算GTEx数据库中每个基因在不同组织类型中的平均表达量。②计算GTEx数据库中每个基因在睾丸组织中的特异性,即
1.2 GEP的鉴定
精子发生过程中的GEP是一组表达模式相似的基因构成的基因集[9]。因此,为了鉴定精子发生过程中CTG所包含的GEP,对CTG在睾丸单细胞的表达谱进行聚类分析。聚类结果可以反映CTG在精子发生过程中的表达模式。
1.2.1 睾丸单细胞转录组原始数据的获取
1.2.2 睾丸单细胞转录组测序分析
将下载的原始测序reads转换成FASTQ格式,并将文件名命名为cellranger可以接受的格式,最后使用cellranger对基因的表达水平进行定量。下游分析使用Python语言的scanpy库进行[12]。定量后的数据,经过质控、过滤、合并,使用scvi-tools进行去批次效应和过滤双细胞[13-14]。随后,使用leiden图聚类算法对细胞进行聚类[15]。leiden算法中影响聚类的参数主要是resolution。为了确定最优的聚类参数,在一系列resolution下使用leiden对细胞进行聚类,并用轮廓系数(silhouette score)评估聚类的效果。Silhouette score是一个经典的用于评估聚类结果的算法,数值越大说明聚类的结果越好。Silhouette score是每个样本单独计算的,由2个部分组成:
表1 睾丸细胞类型、缩写以及其对应的标志基因和细胞数
Tab 1
| Cell type | Abbreviation | Marker gene | Cell number/n |
|---|---|---|---|
| Spermatogonia stem cell | SSC | UTF1, DMRT1 | 5 822 |
| Differentiating spermatogonia | Differentiating SPG | DMRT1 | 2 006 |
| Early primary spermatocyte | Early primary SPC | DMC1 | 2 085 |
| Late primary spermatocyte | Late primary SPC | ZPBP, SPAG6 | 4 484 |
| Round spermatid | Round ST | ZPBP, SPAG6, ACR | 5 653 |
| Elongating spermatid | Elongating ST | TNP1 | 3 541 |
| Elongated spermatid | Elongated ST | TNP2 | 2 511 |
| Sertoli cell | SC | SOX9 | 1 368 |
| Peritubular cell | PC | MYH11 | 3 740 |
| Leydig cell | LC | DLK1 | 4 485 |
| Smooth muscle cell | SMC | MYH11, NOTCH3 | 672 |
| Epithelial cell | EC | VWF | 1 792 |
| Testis macrophage | TM | CD14 | 1 442 |
1.2.3 GEP鉴定过程
为了鉴定精子发生过程中CTG包含的GEP,首先查看所有CTG在睾丸单细胞转录组中的表达情况。由于CTG主要表达于生殖系细胞(见结果部分),随后选择生殖系细胞的表达谱用于鉴定CTG包含的GEP,具体步骤如下:①提取CTG在生殖系细胞中的表达矩阵。②使用leiden图聚类算法对CTG进行聚类。③类似于细胞聚类,选定一系列resolution,并用silhouette score评估聚类结果,选择最终的resolution。④使用最优的聚类参数对CTG进行聚类,得到表达模式相似的CTG组成的基因集。⑤使用热图展示CTG的表达情况,观察不同基因集表达模式的异同。
这些基因集包含不同的基因表达模式,每个基因集表示一个GEP,并且这些GEP之间互不重叠。每个GEP表示为
1.3 GEP的活跃程度及其与患者预后的关系
1.3.1 GEP活性分析
为了定量表示GEP在每个细胞(scRNA-seq)或者样本(Bulk RNA-seq)中的活跃程度,使用DecoupleR包中的wmean函数计算每个GEP的活跃程度[18],表示为
对一个细胞中所有基因,按照每个GEP对应的基因数重复抽样1 000次,并计算其权重平均值,得到该GEP权重平均值的零分布。零分布的均值和方差表示为
1.3.2 生存分析
生存分析用于探索GEP的活跃程度与患者预后的关系,使用R语言中的survival包实现。首先,通过DecoupleR包的wmean函数计算TCGA中每例患者GEP的活跃程度;再将患者分为GEP活跃程度高组和GEP活跃程度低组,比较2组生存曲线;同时,基于患者的 GEP 活跃程度与患者的生存情况,进行Cox回归分析。
2 结果
2.1 CTG的筛选
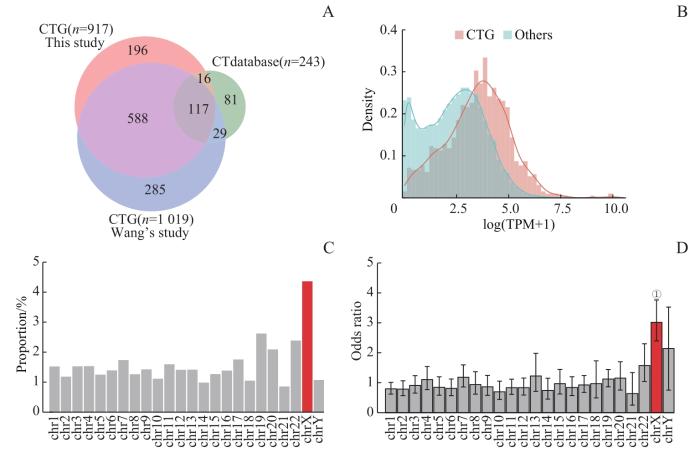
图1
图1
CTG的筛选
Note: A. Comparison between CTGs identified in this study and previous studies. B. Distribution of CTGs and non-CTGs in testis. C. The proportion of CTGs identified in different chromosomes. D. Odds ratio of CTGs on chromosomes. ①P=0.000, compared with other chromosomes.
Fig 1
Screening of CTGs
2.2 GEP的鉴定
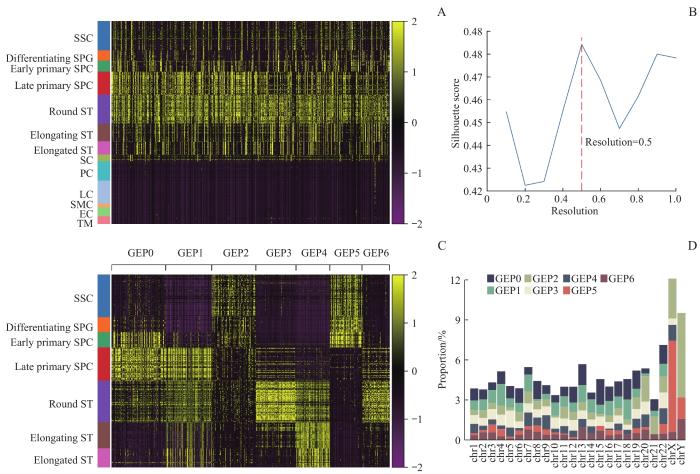
为了得到睾丸单细胞转录图谱,统一定量3套数据的原始测序数据,并进行质控和过滤。采用scvi-tools的整合算法将3组数据整合,共得到39 601个细胞。对整合后的睾丸组织单细胞转录组进行细胞聚类分析(图2)。结果显示,在一系列resolution参数中,当silhouette score取最大值时,对应的resolution为0.8(图2A)。基于此resolution值将细胞聚类为22个类。图2C、D分别展示了睾丸组织中体细胞和生殖系细胞的标志基因表达情况。基于此标注每个聚类类别的细胞类型,最终得到睾丸组织的单细胞转录组图谱(图2B)。共有13种细胞类型,每种细胞类型的全称、数量和标志基因详见表1。
图2
图2
睾丸单细胞转录组聚类分析和细胞类型标注
Note: A. Relationship between resolution parameters in cell clustering and the silhouette score. The red dotted line in the panel indicates the optimal resolution parameter in clustering. B. Single-cell transcriptome map of the testis. C. Expression of marker genes of somatic cell types (SOX9, CD14, VWF, DLK1, NOTCH3 and MYH11) in testis. D. Expression of marker genes of germline cell types (UTF1, DMRT1, DMC1, ZPBP, SPAG6, ACR, TNP1 and PRM2) in testis. The intensity of color in the C/D panels represents the level of gene expression after normalization.
Fig 2
Cluster analysis of testicular single-cell transcriptome and cell types labeling
图3
图3
鉴定CTG包含的精子发生过程中的GEP
Note:A. Expression of CTGs in the entire testicular single-cell transcriptome. B. The relationship between resolution and silhouette score in CTGs clustering. The red dotted line in the panel indicates the optimal position of resolution. C. Expression of CTGs in testicular germ line cells after clustering. D. The proportion of CTGs contained in each GEP on the chromosomes. The values in the heat map of A/B panels were converted to z-scores by the gene. The closer the color is to yellow, the higher the gene expression is; the closer the color is to purple, the lower the gene expression is.
Fig 3
Identification of GEP contained in CTGs during spermatogenesis
2.3 GEP的活跃程度及其与肿瘤患者生存的关系
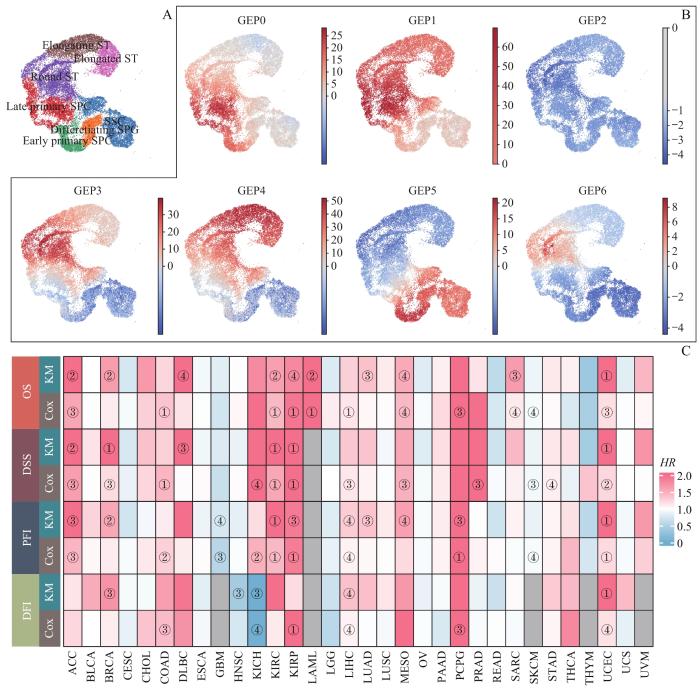
对7个GEP在每个细胞中的活跃程度进行定量计算,并用UMAP降维分析展示每个GEP在每个细胞中的活跃程度(图4A、B)。结果显示:GEP0主要活跃于Early primary SPC、Late primary SPC、Round ST中;GEP1几乎在每个细胞类型中都是活跃的,但在SSC、Differentiating SPG、Early primary SPC中的活跃程度较低;GEP2几乎在每个细胞类型中都是不活跃的;GEP3和GEP4主要活跃于Round ST、Elongating ST和Elongated ST中,GEP3在Round ST中最活跃,GEP4在Elongating ST中最活跃;GEP5主要活跃于SSC、Differentiating SPG、Early primary SPC;GEP6仅在Round ST中活跃。细胞类型缩写及含义见表1。
图4
图4
GEP的活跃情况以及GEP5与肿瘤患者生存的关系
Note:A. Single-cell map of testis germ line cells. B. The activity of GEP in testis germ line cells. The colors in the panel represent the degree of activity of each GEP. The closer the GEP is to red, the more active it is, and the closer the GEP is to blue, the more inhibited it is. C. Relationship between GEP5 activity and prognosis in different cancer types. OS—overall survival; PFI—progression free interval; DFI—disease free interval. The colors represent the hazard ratio (HR) of Kaplan-Meier (KM) survival analysis and Cox regression analysis. White means HR=1; pink means HR=2; light blue means HR=0; gray indicates missing data. ①P<0.001; ②P<0.01; ③P<0.05; ④P<0.10.
Fig 4
Activity of GEP and the relationship between GEP5 and the survival of cancer patients
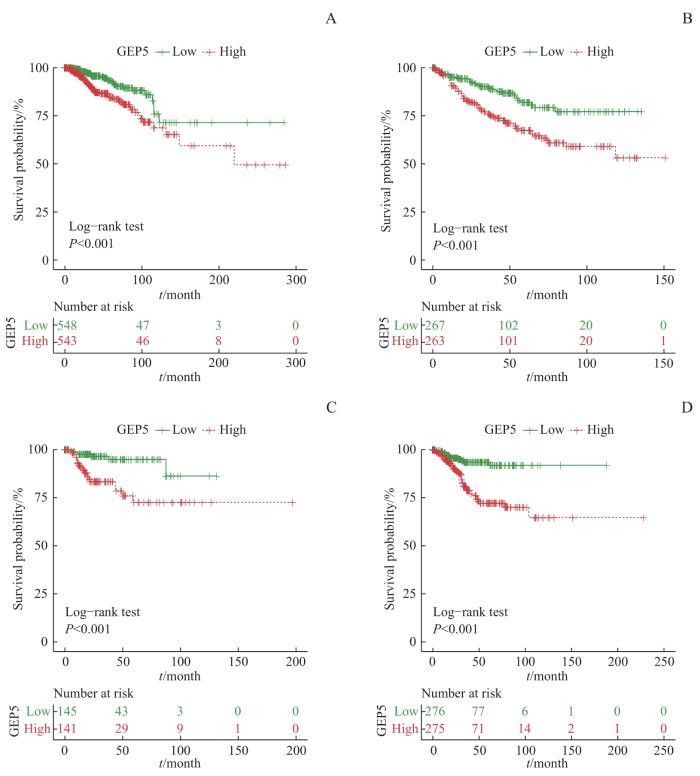
生存分析的结果汇总如图4C所示,整体来看,GEP5的活跃程度与患者的生存期有一定的关系。在大部分的肿瘤类型中,GEP5活跃程度高与患者的生存差有显著的关联,其中ACC(adrenocortical carcinoma)、BRCA(breast invasive carcinoma)、COAD(colon adenocarcinoma)、KIRC(kidney renal clear cell carcinoma)、KIRP(kidney renal papillary cell carcinoma)和UCEC(uterine corpus endometrial carcinoma)最显著(至少有4个风险比显著大于1)。BRCA、KIRC、KIRP、UCEC的疾病特异性生存(disease specific survival,DSS)曲线如图5所示,GEP5活跃程度高组的疾病特异性生存期与GEP5活跃程度低组比较,显著缩短。以上这些数据说明GEP5在多种肿瘤类型中的活跃程度与患者的生存期呈负相关。
图5
图5
GEP5在不同癌症类型中的活跃程度与DSS曲线
Note:A. DSS curve of BRCA.B. DSS curve of KIRC. C. DSS curve of KIRP. D. DSS curve of UCEC. The upper panel shows Kaplan-Meier survival curves; the lower panel presents the number of people at risk (disease) at different time points.
Fig 5
GEP5 activity in different cancer types and DSS curves
3 讨论
CTG具有局限表达于睾丸且在肿瘤中高表达的特征,可作为肿瘤的治疗靶点或诊断标志物。目前,很多这方面的研究聚焦于单基因分析,缺乏对基因间协同作用的研究。本研究利用生物信息学计算分析技术,探索CTG的协同表达规律。
本研究通过基因表达特异性分析,筛选出1 271个TSG,结合TCGA数据库进一步确认917个CTG。在睾丸组织中,CTG的表达水平整体上高于其他基因的表达水平。收录在CTdatabase而没有鉴定到的CTG有110个,CTdatabase根据CTG的表达特征将其分为睾丸限定的(testis-restricted)、睾丸/大脑限定的(testis/brain-restricted)、睾丸选择性的(testis-selective)3类[3]。我们推测,这110个CTG在本研究分析的睾丸组织表达特异性较差,故造成此差异。由于GTEx数据库和TCGA数据库更新,收录了更多的数据,本研究用这2个数据库的所有编码基因作为初始候选基因,而CTdatabase的初始候选基因是文献中记录的CTG[3]。因此,本研究筛选到的CTG远多于CTdatabase中收录的CTG。
为了鉴定精子发生过程中CTG包含的GEP,本研究重新整理了睾丸单细胞转录组数据,并标注了细胞类型。通过睾丸单细胞转录组数据的分析,发现CTG主要表达于生殖系细胞中。睾丸的体细胞类型有SC、LC、PC、SMC、EC、TM。除了SC之外,其他的细胞类型或多或少都会在其他成体组织或器官中存在,故特异表达于睾丸的CTG不会表达于这些细胞中。
根据睾丸生殖系细胞的基因表达谱,对CTG进行聚类,鉴定出7个精子发生过程中的GEP。每个GEP有其偏好表达的细胞类型和精子发生时期。对GEP的活跃程度定量计算后,发现GEP5活跃于SSC、Differentiating SPG、Early primary SPC细胞。此外,研究还发现GEP5中包含了大量的CT-X基因[5-6]。这说明在精子发生过程的前期,大部分的CT-X基因需要活跃的转录。X染色体上的基因受到减数分裂性染色体失活调控(meiotic sex chromosome inactivation,MSCI),其在减数分裂过程的前期活跃转录,在完成染色体联会后转录活性快速受到抑制,并形成XY体[19]。这或许意味着在精子发生过程中,GEP5的活跃程度受到MSCI的调控。
GEP5活跃的细胞类型是SSC、Differentiating SPG、Early primary SPC,即精原细胞增殖期和减数分裂早期。之前的研究结果表明,减数分裂早期相关的CTG对于肺腺癌的发生和发展有重要作用[4]。因此,本研究将GEP5在肿瘤中的活跃情况与患者的预后进行关联分析,以探究GEP5的活跃程度与患者预后之间的关系。结果显示,GEP5在ACC、BRCA、COAD、KIRC、KIRP和UCEC中的活跃程度高与患者的生存状况差有显著的关联。这说明GEP5在多种肿瘤类型中的活跃程度与患者的预后有关,具有成为多种肿瘤类型预后判断标志物的潜力。
综上所述,本研究通过多数据库联合分析,对CTG进行重新筛选和分类,并鉴定出7个GEP。其中,GEP5活跃于精子发生过程的前期,且富集CT-X基因。此外,在肿瘤中,GEP5的活跃程度越高,患者的生存状况越差,有望支持多种肿瘤类型的预后判断。
作者贡献声明
雷鸣设计并指导了整个课题的研究;侯宗良主要完成数据收集、数据分析及处理、文章撰写;杨琴和李少白主要负责数据分析指导、算法分析指导以及整篇论文的修改。所有作者均阅读并同意最终稿件的提交。
AUTHOR's CONTRIBUTIONS
LEI Ming designed and guided the whole research project. HOU Zongliang mainly completed the work of data collection, data analysis and processing and article writing. YANG Qin and LI Shaobai were mainly responsible for the guidance of data analysis and algorithm analysis, as well as the revision of the whole paper. All authors read the final manuscript and approved the submission.
利益冲突声明
所有作者声明不存在利益冲突。
COMPETING INTERESTS
All authors disclose no relevant conflict of interests.
参考文献