近年来,人工智能技术迅速发展,已成功应用于各个领域。在医学领域中,深度学习(deep learning)取得了巨大的研究进展,尤其是在消化内镜领域。卷积神经网络(convolutional neural network,CNN)的引入,已经实现了胃肠镜检查质量控制、胃早期癌识别、胃早期癌深度预测以及肠息肉监控等多项功能。Transformer技术在医学领域的热度和关注度正迅速超越CNN,成为该领域的新焦点。
1 材料与方法
1.1 数据集
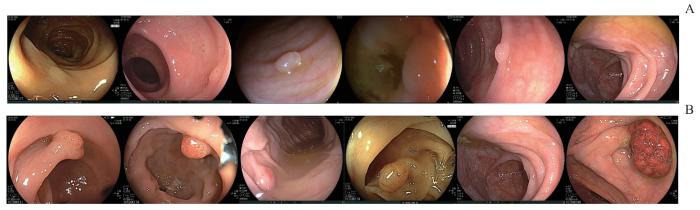
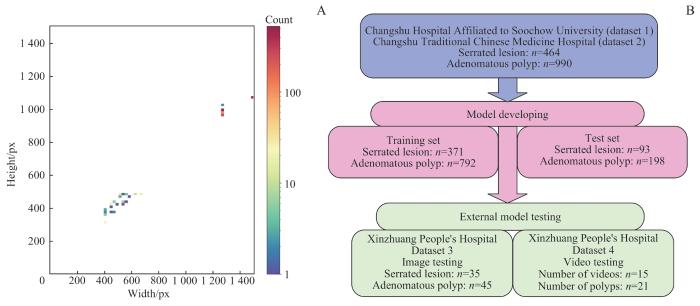
采用了4个数据集,时间跨度为2018年1月至2023年6月。数据集1和数据集2分别来自苏州大学附属常熟医院和常熟市中医院,包括1 454张肠息肉图像,用于模型的开发和测试。数据集3由常熟市辛庄人民医院提供,包含80张肠息肉图像,作为模型的外部静态图像测试集。数据集4也来自常熟市辛庄人民医院,包括15个肠镜视频,内含21个息肉,用于模型的外部视频测试。外部测试集仅用于评估模型性能,未参与训练或调参,以保证其独立性。结肠镜检查采用多个品牌的内窥镜系统,共13台电子内窥镜,包括开立(SonoScape HD-550)、奥林巴斯(OLYMPUS CV-V1)、宾得(Pentax EPK-i7000)和富士(ELUXEO 7000)。图像包括腺瘤性息肉和锯齿状病变图像(图1)。为了增强模型的泛化能力和鲁棒性,选择的内镜图像覆盖了不同清洁度的肠道情况。所有检出息肉均经切除并进行组织病理学检查;排除炎症性肠病、活动性结肠炎、凝血障碍、家族性息肉病和急诊结肠镜检查患者。
图1
图1
数据集中息肉图像示例
Note: A. Serrated lesions. B. Adenomatous polyps.
Fig 1
Examples of polyp images from the datasets
图2
图2
数据集图像分布情况
Note: A. Distribution of image sizes in the dataset. B. Distribution of images across various categories.
Fig 2
Distribution of images in the datasets
1.2 图像标注
所有图像和视频中展示的息肉均经病理学验证。如遇到活检和切除标本的病理结果不一致,优先采用切除标本的结果。两位具有10年以上工作经验的资深内镜医师根据病理结果对肠镜图像进行分类。将结肠息肉分为腺瘤性息肉(管状腺瘤、绒毛状腺瘤、绒毛管状腺瘤)和锯齿状病变(增生性息肉、无蒂锯齿状病变、锯齿状腺瘤),基于3个关键考量:首先,大多数结肠息肉可以归类为腺瘤性或锯齿状病变;其次,病理学上,在区分腺瘤性息肉和锯齿状病变方面表现出极高的一致性,而在区分增生性息肉和无蒂锯齿状病变方面则一致性较差;最后,本研究数据集中其他类型息肉的数量不足,无法用于开发一个稳健的“多类别”模型。为保证图像质量,采用定期维护的高分辨率内窥镜,按照肠道准备指南实行结肠镜检查前的肠道准备[13]。
使用Python3.7版本LabelMe(v5.3.1)图形图像标注工具[14]对结肠镜图像中的息肉进行了多边形(Polygons)标注,并根据病理学结果将其分为3类:正常肠道、腺瘤性息肉和锯齿状病变。为了适应深度学习模型的训练需求,开发一个专用的Python脚本,用以将LabelMe格式的JSON文件转换成整数掩膜格式。在这种格式中,每个类别被赋予一个唯一的整数标识:正常背景标为0,腺瘤性息肉标为1,锯齿状病变标为2。这样的转换使模型能够直接处理像素级的分类信息,显著提高了训练效率和图像分割的准确性。
制定规范化的图像标注程序,并对标注人员进行专业训练,以确保他们能够精准地识别息肉并严格按照既定程序操作。采用双重标注系统,允许不同的标注人员独立地标注同一图像,并对选定结果进行随机核对,以提高标注的一致性和精确率。定期邀请经验丰富的专家复审标注结果,并根据其反馈进行调整,以持续提高标注的品质。
1.3 图像预处理
本研究进行一系列系统的图像预处理和增强步骤。采用在线数据增强策略[15-16],所有数据增强操作都在模型训练过程中实时执行,不生成新的图像文件。这保证了在每个训练周期(epoch)中,模型都能够接触到略有差异的图像版本。这些操作包括随机上下和左右翻转、多角度旋转、仿射变换等,旨在增加训练数据量,防止模型过拟合,同时提升模型的泛化能力。在预处理阶段,使用RandomResize策略随机调整图像大小,此策略设定2 048×1 024为基准尺寸,并保持图像宽高比,通过应用0.5~2.0的缩放比例,动态调整图像尺寸,使其相对于基准尺寸缩小至50%或放大至200%。此外,还实施了50%概率的随机水平翻转,以及512×512尺寸的RandomCrop进行随机裁剪,以帮助模型从部分信息中识别目标。采用PhotoMetricDistortion进行光度失真处理,增强模型对于不同光照和颜色变化的适应性。
1.4 模型训练配置
优化深度学习分割任务,对4种预训练模型采用迁移学习策略[17]。这包括2个基于CNN的模型:Fast-SCNN,预训练于Cityscapes数据集;DeepLabV3plus,预训练于ADE20K数据集。此外,还使用了基于Transformer架构的Segformer模型,以及融合了 CNN和Transformer架构的KNet模型,两者均在 ADE20K数据集上进行了预训练。通过迁移学习策略,在收集并标注的息肉分割数据集上对这些预训练模型进行微调。这一策略不仅能够充分利用在大规模数据集上获得的先进特征,还大幅降低了从头开始训练所需的数据量和计算资源。
在训练过程中,采用随机梯度下降(stochastic gradient descent,SGD)作为优化器,并配合预设的多项式衰减(PolyLR)学习率调度器优化学习率,从而提高模型训练的效率。模型训练共设定为20 000次迭代,每批次处理5个样本,以保证学习过程的有效性。以Fast-SCNN的迁移学习过程为例,全面更新了Fast-SCNN骨干网络、2个辅助头和 DepthwiseSeparableFCNHead解码头的参数。利用 OpenMMLab(resnet101_v1c)上的预训练权重,这种策略不仅限于网络的特定层,而是涵盖了整个网络结构。通过SGD优化器和多项式衰减学习率调度,在40 000次迭代期间,学习率逐步降低以适应新数据。
1.5 模型性能评估
为全面评估结肠息肉图像分割模型的性能,采用多元化的评估指标:整体准确率(aAcc),用于反映模型在所有预测中的整体准确性;平均交并比(mIoU),作为图像分割领域的核心指标,评估预测与实际区域的重叠度;平均准确率(mAcc)和平均Dice系数(mDice),提供类别级别的性能评估和衡量预测与真实区域的重叠程度;平均F分数(mFscore),评价查准率(Precision)与查全率(Recall)的平衡;平均精确率(mPrecision)与平均召回率(mRecall),关注正类别的识别与覆盖能力;此外,通过混淆矩阵定量分析,实现对模型在像素级分类精度上的全面评估。
1.6 实验平台
利用PyTorch和其他Python库,构建、训练深度学习模型并处理图像。数据的整理、分析和可视化通过Pandas(1.3.4)、NumPy(1.21.4)、Matplotlib (3.5.0)和Plotly(5.4.0)实现。模型优化使用PyTorch(1.10.0+cu113),而模型的保存和加载则依赖于H5py(3.6.0)。另外,使用Matplotlib、tqdm和opencv-python进行数据可视化和图像处理。
利用OpenMMLab(
2 结果
2.1 模型训练和预测结果
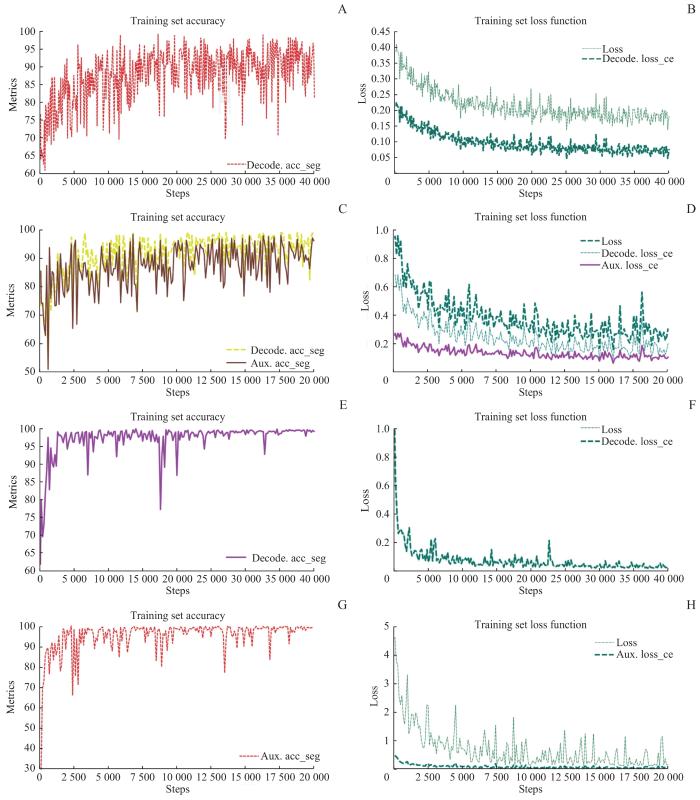
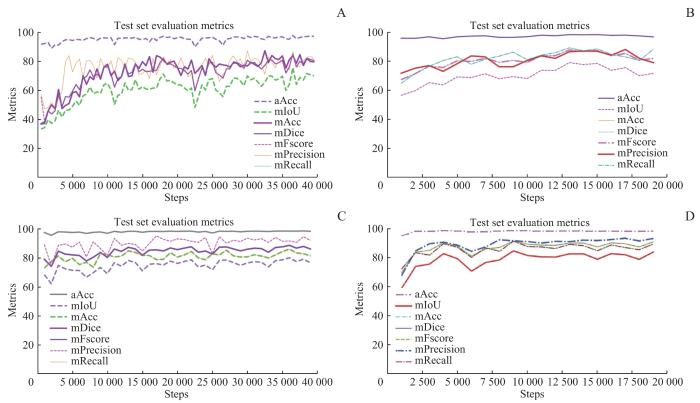
图3A、 C、 E和G展示了Fast-SCNN、 DeepLabV3plus、Segformer和KNet这4个深度学习分割模型在训练过程中准确率的持续提升,表明模型能够稳定地识别训练数据中的关键特征,从而实现对数据的有效拟合。图3B、D、F和H展现了这4个模型在损失函数上的下降趋势。随着训练的深入,每个模型的损失函数都呈现出明显的下降趋势,并逐步稳定化,这显示模型正在有效地向收敛状态进展。图4显示4个模型在测试集上的多项性能指标(aAcc、mIoU、mAcc、mDice、mFscore、mPrecision、mRecall)随训练过程的变化。虽然这些模型在特定指标的变化趋势上存在差异,但最终都显示出了随着训练进程的稳定性,没有出现明显的性能反弹或波动。
图3
图3
4种分割模型在训练集上的性能指标随训练进程的变化
Note: A/B.Accuracy (A) and loss function (B) fluctuation of the Fast-SCNN model. C/D.Variations in accuracy (C) and loss function (D) in the DeepLabV3plus model. E/F. Segformer model's accuracy (E) and loss function (F) changes. G/H. KNet model's accuracy evolution (G) and loss function (H)alterations. All figures encapsulate the decoder segmentation accuracy (decode.acc_seg) and auxiliary classifier segmentation accuracy (aux.acc_seg).
Fig 3
Changes in performance metrics for four segmentation models in the training set throughout the training process
图4
图4
4种分割模型在测试集上的性能指标随训练进程的变化
Note: A. Fast-SCNN model. B. DeepLabV3plus model. C. Segformer model. D. KNet model.
Fig 4
Changes in performance metrics for four segmentation models in the test set throughout the training process
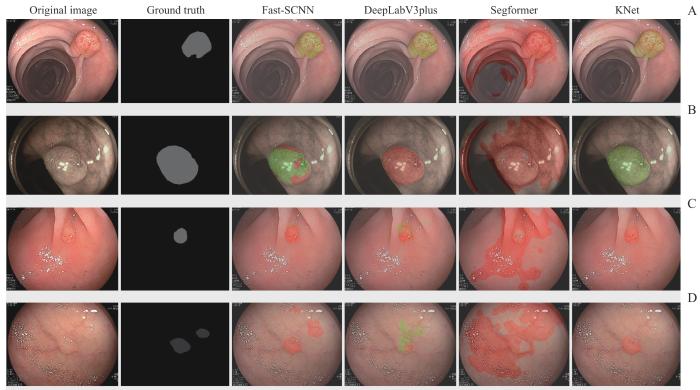
图5显示4种不同架构的息肉语义分割模型(Fast-SCNN、DeepLabV3plus、Segformer、KNet)在外部测试集上的预测效果,通过4张随机选取的结直肠息肉静态图像进行展示。该图包含6列,依次展示原始息肉图像、真实标注(ground truth,GT),以及上述4种分割模型的预测图。在模型预测图中,腺瘤性息肉的识别区域以绿色标识,而锯齿状病变的识别区域以红色标识,背景则未着色。KNet正确预测了所有息肉类型,并且KNet对图5A、B、C的预测区域与GT的一致性较高。然而,在图5D中,KNet漏诊了一枚微小锯齿状息肉。Fast-SCNN模型也准确预测了所有息肉类型,并成功识别了KNet漏诊的那枚微小锯齿状息肉。尽管Fast-SCNN的预测结果在主要区域的颜色标记大体准确,但在图5B和5D中存在一定比例的颜色标记误差。Segformer在随机选取的4张外部测试集图像上展现出较差的分割性能,表明模型的泛化能力有限。DeepLabV3plus在预测区域与GT的一致性方面表现较好,但在图5B和5D中错误地分类了息肉类型。
图5
图5
不同模型在外部测试集上的预测结果对比
Note: A/B. Adenomatous polyps. C/D. Serrated lesions. Green represents the predicted areas for adenomatous polyps, and red represents the predicted areas for serrated lesions.
Fig 5
Comparison of prediction results from different models in an external test set
2.2 模型性能评价
2.2.1 模型在测试集上的平均性能指标
对比不同架构深度学习模型在291张测试图像上的平均分割性能指标。Fast-SCNN作为CNN类模型之一,具有97.42%的aAcc,但其在mIoU 和 mAcc上的表现稍显逊色,分别为75.32%和83.80%。而DeepLabV3plus模型在所有性能指标上均显著提升,尤其是在mIoU和mAcc上,分别达到了78.63%和88.78%。Transformer架构的Segformer模型,在维持较高aAcc的同时呈现出均衡的性能,其mIoU和mAcc分别为80.17%和84.71%,并在 mPrecision上达到了92.97%,但mRecall略低(84.71%)。然而,在所有模型中,融合了CNN和Transformer优势的混合架构(Hybrid)模型KNet表现最为突出。KNet在几乎所有评价指标上都展现出了高性能:aAcc达到了98.59%,mIoU和mAcc分别为84.59%和91.24%,证明了其在识别息肉区域上的高准确性;KNet的mDice和mFscore均超过91%,显示了其在分割精确率和一致性上的优越表现。同时,该模型在mPrecision和mRecall上也保持了高水平,分别为91.46%和91.24%。
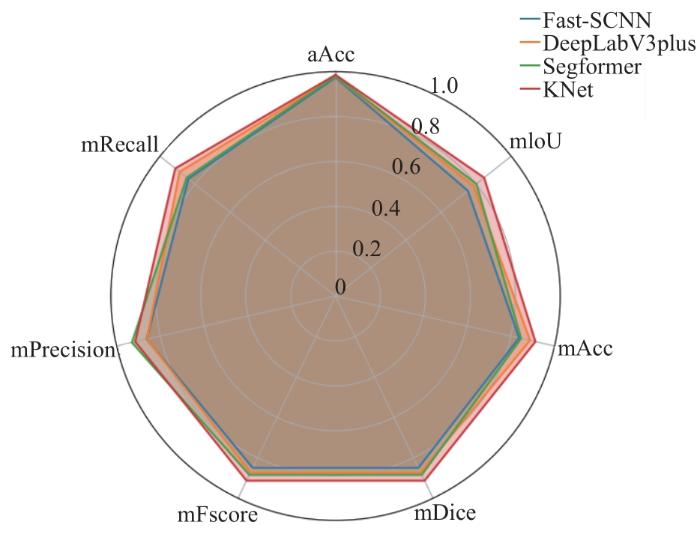
在图6所示的雷达图中,KNet模型(命名为KNet-ColoSeg)展现了显著的性能优势。KNet-ColoSeg在图表上的指标几乎完全超越了其他比较模型,形成了一种明显的包裹现象。KNet-ColoSeg在aAcc、mPrecision和mRecall等核心指标上表现尤为优异。
图6
图6
测试集上4种结肠息肉分割模型的平均分割性能指标对比
Fig 6
Comparative metrics of the average performance of four colon polyp segmentation models in the test set
2.2.2 模型在测试集不同类别息肉上的性能指标
对4个已构建的结直肠息肉语义分割模型进行测试集上的性能评估(表1)。评估结果显示,在背景类别的分割中,这些模型呈现出优异的性能,平均IoU达到98.28%,Acc为99.27%,Dice系数和F分数均为99.13%,Precision为98.995%,Recall为99.27%。在腺瘤性息肉类别中,所有模型的平均IoU为74.74%,Acc为83.68%,Dice系数为85.48%,F分数为85.48%,Precision为87.40%,Recall为83.68%。而在处理锯齿状病变类别时,所有模型的平均IoU为66.01%,Acc为78.45%,Dice系数为79.40%,F分数为79.40%,Precision为81.18%,Recall为78.45%。
表1 4种模型在测试集上的分类预测性能指标(%)
Tab 1
| Model | Category | IoU | Acc | Dice | Fscore | Precision | Recall |
|---|---|---|---|---|---|---|---|
| Fast-SCNN | Background | 97.74 | 99.02 | 98.86 | 98.86 | 98.70 | 99.02 |
| Serrated lesion | 59.08 | 71.13 | 74.27 | 74.27 | 77.70 | 71.13 | |
| Adenomatous polyp | 69.15 | 81.24 | 81.76 | 81.76 | 82.30 | 81.24 | |
| DeepLabV3plus | Background | 98.26 | 98.96 | 99.12 | 99.12 | 99.28 | 98.96 |
| Serrated lesion | 63.63 | 83.38 | 77.78 | 77.78 | 72.88 | 83.38 | |
| Adenomatous polyp | 74.00 | 84.02 | 85.06 | 85.06 | 86.12 | 84.02 | |
| Segformer | Background | 98.20 | 99.6 | 99.09 | 99.09 | 98.60 | 99.60 |
| Serrated lesion | 67.22 | 71.95 | 80.39 | 80.39 | 91.09 | 71.95 | |
| Adenomatous polyp | 75.09 | 82.57 | 85.77 | 85.77 | 89.23 | 82.57 | |
| KNet | Background | 98.91 | 99.50 | 99.45 | 99.45 | 99.40 | 99.50 |
| Serrated lesion | 74.12 | 87.33 | 85.14 | 85.14 | 83.05 | 87.33 | |
| Adenomatous polyp | 80.73 | 86.88 | 89.34 | 89.34 | 91.94 | 86.88 |
KNet模型(命名为KNet-ColoSeg)具有最高的交并比(IoU),属性能最优的模型。KNet-ColoSeg在背景类别的分割中表现卓越,其IoU达到98.91%,Acc高达99.50%,同时Dice系数和F分数均保持在99.45%,Precision和Recall分别为99.40%和99.50%。在处理锯齿状病变时,该模型展现出了74.12%的IoU和87.33%的Acc,Dice系数和F分数均为85.14%,Precision为83.05%,Recall为87.33%。对于腺瘤性息肉类别,KNet-ColoSeg的IoU为80.73%,Acc为86.88%,Dice系数和F分数均达到89.34%,Precision高达91.94%,Recall为86.88%。
2.3 人工智能模型预测新的图像和视频
KNet-ColoSeg模型在外部测试集上的综合性能评估中,aAcc、mAcc和mIoU分别为97.72%、90.13%和81.53%。模型在处理背景类别时具有优势,达到了98.81%的IoU、99.49% 的Acc、99.4%的Dice系数和F分数,以及99.31%的Precision和99.49%的Recall。针对锯齿状病变类别,模型展现了63.53%的IoU、92.72%的Acc、77.70%的Dice系数和F分数、66.87%的Precision以及92.72%的Recall。在腺瘤性息肉类别中,模型实现了82.25%的IoU、84.87%的Acc、90.26%的Dice系数和F分数、96.38%的Precision和84.87%的Recall。
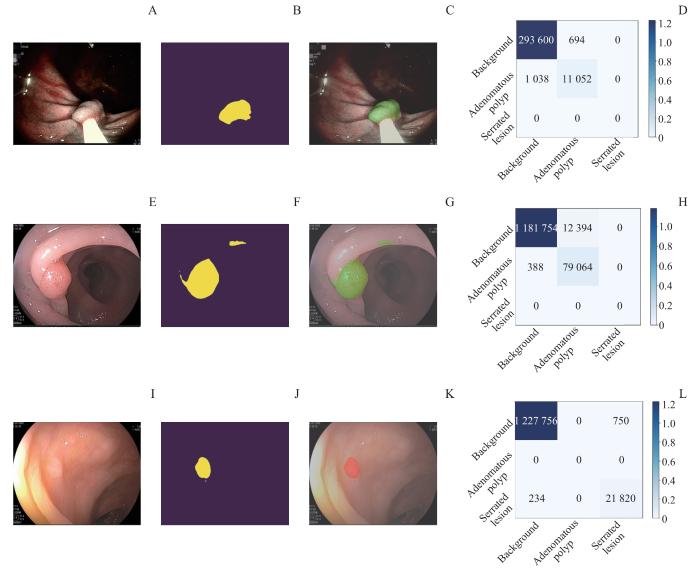
KNet-ColoSeg模型在外部测试集上的3张随机选取的结直肠息肉图像上的推理预测结果(图7),分别展示了原始息肉图像、模型预测输出、预测结果覆盖在原始图像上的视图,以及针对单张图像的混淆矩阵。从3张原始图像的分析中观察到,模型成功识别了所有息肉类别。尽管混淆矩阵揭示了少量像素的误分类,但总体来看,模型的预测与原始图像高度一致,显示了其在图像分割准确性方面的出色性能。
图7
图7
KNet-ColoSeg 模型在外部测试集上对新息肉图像的预测结果
Note: This figure consists of four columns, including the original polyp images (A, E and I), the model's prediction output (B, F and J), a view of the model's predictions superimposed on the original images (C, G and K), and the confusion matrix for the prediction of individual images (D, H and L).
Fig 7
Prediction results of KNet-ColoSeg model for new polyp images in an external test dataset
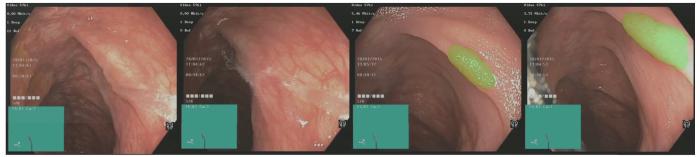
为了评估KNet-ColoSeg模型在实际应用中的性能,我们利用该AI模型对外部测试集(数据集4)的视频进行实时预测。图8展示了AI模型在实时定位和分割息肉的能力,并演示了其自动化判断息肉性质的过程。
图8
图8
KNet-ColoSeg模型对一例视频案例的实时预测过程
Note: The image sequence from left to right demonstrates a serrated lesion case successfully segmented by the AI model after irrigation with physiological saline.
Fig 8
KNet-ColoSeg model for real-time prediction of a video case
3 讨论
本研究从多个内镜中心收集结肠镜图像数据,创建了一个专门的数据集,用于训练和测试针对结直肠息肉分割的人工智能模型。该数据集包括锯齿状病变和腺瘤性息肉图像,旨在评估不同架构的结肠息肉语义分割模型。通过迁移学习技术,开发并比较了Fast-SCNN、DeepLabV3plus、Segformer和KNet深度学习模型。KNet模型结合了CNN和Transformer的优势,在测试集上表现最佳,其mIoU高达84.59%,将其命名为KNet-ColoSeg。在外部测试集上,KNet-ColoSeg展现出了81.53%的mIoU,证明了其在临床环境中的广泛适用性和鲁棒性。
结直肠癌的肿瘤发生假说,根据癌前病变的性质主要分为3种途径:腺瘤-癌变序列(adenoma-carcinoma sequence)[18]、锯齿状病变途径(serrated neoplasia pathway)[19]、de novo致癌[20]。从频度上看,作为结直肠癌的发展途径,前两者是结直肠癌发展的主要途径,但它们在临床病理学和分子异常方面被认为是互斥的[21-22]。2019年WHO指南[23]提出,使用“锯齿状病变”定义,将增生性息肉(hyperplastic polyps,HP)、传统锯齿状腺瘤(traditional serrated adenomas,TSA)和无蒂锯齿状病变(sessile serrated lesions,SSL)统称为锯齿状病变。在临床处理和病理诊断中,区分腺瘤性息肉和锯齿状病变至关重要。因为如果漏诊,该病可能通过不同途径恶化为癌症。因此,运用深度学习模型识别和区分这2种病变,具有重要的临床应用价值。
深度学习技术,特别是其卓越的特征提取和数据处理能力,为结肠镜下息肉的智能化识别和诊断提供了有效方法。随着深度神经网络技术的快速发展,其在息肉图像分割方面的应用也日益增多。ResUNet++网络,通过结合残差块、压缩激发块、空间金字塔池化和注意力机制,在CVC-612数据集上实现了79.55%的Dice系数和79.62%的mIoU[24];DeepLab在PASCAL VOC-2012息肉语义图像分割任务中达到了79.7%的mIOU[25]。尽管,两者在息肉分割性能上均取得了显著成果,但未能进一步区分息肉性质。在本研究中,通过汇集多个内镜中心的结肠镜图像,构建了一个包含锯齿状病变和腺瘤性息肉的专用数据集。在此数据集上训练的KNet-ColoSeg模型获得了91.31%的mDice系数和84.59%的mIoU,效果优于上述研究。
在结直肠息肉的研究中,多种深度学习模型架构已得到广泛应用[26-28]。特别是CNN,因其在处理图像等固定形状数据方面的优势而备受关注。此外,最初在自然语言处理领域确立新标准的自注意力机制的Transformer模型,也已成功扩展到计算机视觉领域[29]。这些技术的发展不仅提升了分析效率,还为结直肠息肉的识别和分类提供了新视角。本研究使用2种基于CNN的模型(Fast-SCNN和DeepLabV3plus)、1种基于Transformer架构的模型(Segformer)以及1种融合CNN和Transformer的混合架构模型(KNet),并通过在测试集上的比较,筛选出了最优模型。KNet在上述4种模型中表现出最佳性能,这得益于其统一、简洁且高效的框架,利用一组动态可学习的内核,专门生成特定实例或类别的掩码。这种内核更新策略允许KNet自适应地根据输入图像中的特征,有效区分不同的实例。结合了CNN的空间特征提取能力和Transformer的注意力机制,KNet在处理结直肠息肉图像分割任务时,通过二分匹配而无需非极大值抑制(NMS)的端到端训练和推断过程,显著提升了性能和效率[30]。
本研究通过汇集并构建一个涵盖锯齿状病变与腺瘤性息肉的结肠镜图像数据集,所开发的KNet-ColoSeg模型在结直肠息肉分割任务上显示出优越的性能,实现了对结直肠息肉图像像素级的分割。利用CNN与Transformer的结合,这一模型不仅在测试集上实现了91.31%的mDice系数和84.59%的mIoU,在外部测试集上也表现出81.53%的mIoU,展现了其对于临床诊断支持的潜在应用价值。
尽管本研究在结直肠息肉分割任务中提供了一些见解,仍需注意一些限制。特别是数据集中SSL的样本数量有限,限制了在构建能够识别多类别息肉的强健模型方面的进展。在我们的视频案例测试中发现,尽管模型能够在冲洗后识别SSL区域,但在确定息肉性质时偶有误分类情况发生。未来工作将着重于数据集的扩增,以便进一步提高模型的准确性和鲁棒性。
作者贡献声明
陈健负责研究设计及论文写作;王珍妮、王甘红、刘罗杰参与数据收集和整理;夏开建负责代码解释和报错解决;徐晓丹负责修订论文。所有作者均阅读并同意了最终稿件的提交。
AUTHOR's CONTRIBUTIONS
CHEN Jian was responsible for the design and writing of the paper. WANG Zhenni, WANG Ganhong, and LIU Luojie participated in data collection and organization. XIA Kaijian led the interpretation of codes and the resolution of errors. XU Xiaodan was responsible for revising the manuscript. All authors have read and agreed to the submission of the final manuscript.
利益冲突声明
所有作者声明不存在利益冲突。
COMPETING INTERESTS
All authors disclose no relevant conflict of interests.
参考文献