蛋白赖氨酸去甲基化酶5C(lysine demethylase 5C,KDM5C)是以H3K4me2、H3K4me3为底物的特异性的赖氨酸去甲基化酶,通过减少H3K4me3的水平调控靶基因的转录[5]。该基因位于X染色体上,编码的蛋白普遍表达在各种类型的细胞中,具有较低的细胞特异性。KDM5C基因突变见于多种肿瘤,包括前列腺癌[6]、结肠癌[7]、肾癌[8]、胃癌[9]等等。已有研究表明KDM5C突变可以导致异染色质非编码 RNA表达异常增加,引发基因组不稳定[10],产生更多的随机突变和染色体的缺失及异常的核型。此外,表观遗传的改变与肿瘤细胞的异常代谢息息相关。在肾透明细胞癌中,KMD5C的突变会改变癌细胞的糖代谢,导致糖原蓄积[8];在胆管癌中则会改变细胞的脂肪代谢[11]。本研究拟探索敲除KDM5C后肾透明细胞癌出现的新表型,并结合生物信息学分析探讨敲除KDM5C后促进肾透明细胞癌发生发展的潜在机制,分析其与患者生存预后的相关性,为临床具有该基因突变的患者改善治疗效果提供新的思路。
1 材料与方法
1.1 实验材料
1.1.1 细胞与试剂
肾透明细胞癌细胞786-O,Caki-1和人胚肾细胞293-T均购于中国科学院典型培养物保藏委员会细胞库。
RPMI-1640培养基、DMEM培养基、McCoy′s 5A培养基、胎牛血清(fetal bovine serum,FBS)和谷氨酰胺GlutaMAX Supplement均购于美国Gbico公司。磷酸盐缓冲液(PBS)、0.25%胰蛋白酶溶液和青霉素-链霉素溶液(100×)均购于吉诺生物医药技术有限公司。KDM5C单克隆抗体、山羊抗兔和抗鼠荧光二抗购于武汉爱博泰克生物科技有限公司,上皮-间充质转化(epithelial-mesenchymal transition,EMT)相关蛋白抗体试剂盒和α微管蛋白(α-tubulin)抗体购于美国CST公司。Transwell小室购于美国Corning公司。脂质体核酸转染试剂购于翊圣生物科技有限公司。TRIzol试剂购于Thermo Fisher公司,RNA测序服务由杭州联川生物技术有限公司提供。
1.1.2 主要仪器
恒温CO₂培养箱(HERA cell 150i,美国Thermo Fisher公司),核酸、蛋白检测仪(NANO Drop2000,美国Thermo Fisher公司),蛋白电泳装置(1645050,美国Bio-Rad公司),odyssey红外成像系统(美国LI-COR Biosciences公司),倒置荧光相差显微镜(日本OLYMPUS公司)。
1.2 实验方法
1.2.1 细胞培养
786-O细胞使用含10%FBS、100 U/mL青霉素、100 μg/mL链霉素和1% GlutaMAX的1640培养基进行培养。Caki-1细胞使用McCoy′s 5A培养基,含10%FBS、100 U/mL青霉素、100 μg/mL链霉素。293-T细胞使用10%FBS、100 U/mL青霉素、100 μg/mL链霉素的DMEM培养基进行培养。细胞的培养条件均为37 ℃和5%CO2。
1.2.2 细胞中KDM5C及EMT相关蛋白的检测
使用蛋白质印迹法(Western blotting)检测肾透明细胞癌细胞的KDM5C的表达情况。制备全细胞裂解液,经过蛋白电泳、转膜、封闭、一抗、二抗孵育,最后用Odyssey红外成像系统进行检测。一抗采用KDM5C抗体、α-tubulin抗体、EMT相关蛋白抗体(稀释比例为1∶1000);二抗采用山羊抗兔、抗鼠荧光抗体(稀释比例为1∶5 000)。
1.2.3 KDM5C-sg质粒构建
通过CHOPCHOP(
1.2.4 慢病毒包装
转染前1 d将293-T细胞铺板,使用脂质体核酸转染试剂将构建好的重组质粒KDM5C-sg与包装质粒pMG2.D、psPAX2按2∶1∶2的比例与293-T细胞共同转染48 h,收集转染48 h、72 h的富含病毒颗粒的上清液并过滤,得到慢病毒液,冻存于-80 ℃冰箱储存。
1.2.5 构建稳定敲除KDM5C的人肾癌细胞株
取靶细胞铺板与6孔板中,使其融合度达30%~40%;按慢病毒感染复数(multiplicity of infection,MOI)=20加入病毒液并添加浓度为2.5 μg/mL凝聚胺进行感染,培养48 h更换培养液,并加入嘌呤霉素使其终浓度为2 μg/mL,继续培养48 h,细胞扩大培养后提取蛋白,使用Western blotting检测KDM5C敲除的效果。
1.2.6 细胞迁移和侵袭能力检测
将KMD5C敲除(KDM5C-sg)与未敲除(Control-sg)的786-O、Caki-1细胞按每孔3万个的数量加入Transwell和Invasion小室中,观察细胞的迁移与侵袭能力,分别重复3次实验。小室下层添加500 μL含10%FBS的RPMI-1640,小室中的培养基FBS浓度为1%,将小室放入37 ℃、5%CO2的温箱中培养24 h。经过4%多聚甲醛固定10 min,PBS洗涤3次,结晶紫染料染色30 min,再用PBS洗涤3次,最后用湿润的棉签擦去小室内未迁移的肿瘤细胞,显微镜下拍照,并统计穿过小室的细胞总数,取3个复孔的平均值表示肿瘤细胞迁移、侵袭的能力。
1.2.7 肾癌细胞RNA测序
将786-O Control-sg和 786-O KDM5C-sg细胞用TRIzol处理,每皿细胞加入1 mL,各重复3份,冻于-80℃冰箱中后将样品送到杭州联川生物技术有限公司进行测序分析。采用TruSeq Stranded mRNA Library Prep Kit(美国IIIumina公司)构建测序文库,使用High Sensitivity DNA Kit(美国Agilent公司)检测文库质量,测序平台为Illumina Novaseq 6000(美国IIIumina公司)。
1.2.8 测序数据分析
通过R 4.2.0对RNA测序的raw counts数据进行清洗、分析。使用Limma包、edgeR包和Deseq2包对2组细胞的基因做差异表达分析,将符合P<0.05且log2FC>1的基因作为上调基因;P<0.05且log2FC<-1作为下调基因,并取交集。使用clusterProfiler包做GO、KEGG富集分析,找到差异基因富集的功能通路。使用肾癌细胞RNA测序找到的差异基因在TCGA数据库进行生存分析和单因素COX回分析(P<0.01),使用glmnet包构建LASSO回归模型,筛选与表型相关的基因。
1.3 统计学方法
应用GraphPad Prism 8.0软件对研究数据进行统计分析。定量资料以x±s表示,采用Student′s t检验进行组间。P<0.05表示差异具有统计学意义。癌症基因组图谱(The Cancer Genome Atlas,TCGA)中KDM5C表达量分析通过Timer2.0网站(
2 结果
2.1 KDM5C 在泛癌中的表达水平及突变情况
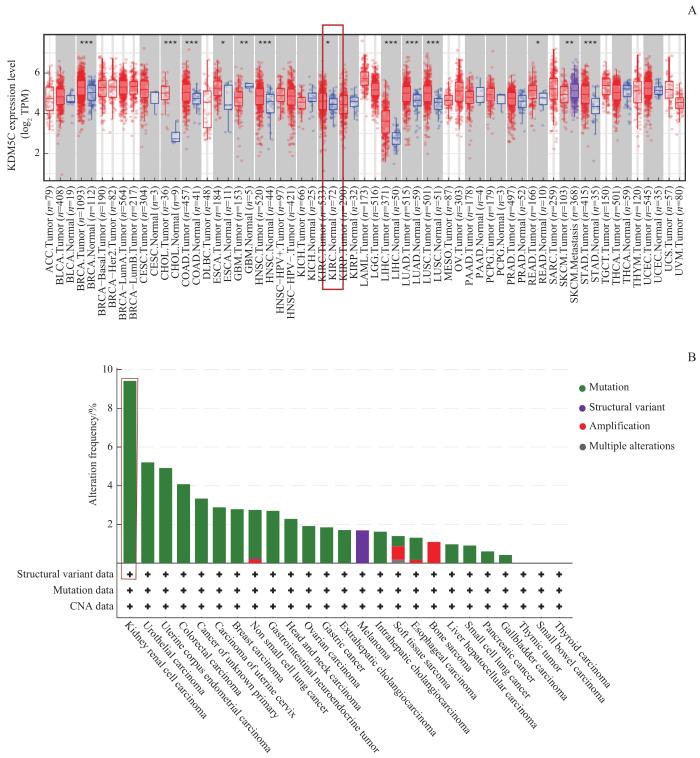
本研究使用Timer2.0在线平台对30种癌症的KDM5C转录水平进行分析(图1A)。结果显示KDM5C在乳腺癌(breast carcinoma,BRCA)、胆管癌(cholangio carcinoma,CHOL)、结肠癌(colon adenocarcinoma,COAD)、头颈部鳞状细胞癌(head and meck squamous cell carcinoma,HNSC)、肝细胞癌(liver hepatocellular carcinoma,LICH)、肺腺癌(lung adenocarcinoma,LUAD)、肺鳞癌(lung squamous cell carcinoma,LUSC)、胃癌(stomach adenocarcinoma,STAD)、肾透明细胞癌(renal clear cell carcinoma,RCCC)、食管癌(esophageal carcinoma,ESCA)、直肠腺癌(Rectum adenocarcinoma,READ)11种癌症中高表达。接着使用cBioportal在线网站分析KDM5C在泛癌中的突变情况,结果显示肾透明细胞癌具有最高的突变率(图1B)。肾透明细胞癌作为一种易发生表观遗传学改变的癌症,突变分析结果也提示KDM5C对其发生发展可能具有重要作用。
图1
图1
KDM5C 在不同癌症中的表达情况
Note: A. Expression of KDM5C mRNA in tumor samples and controls in the TCGA database. B. The mutation ratio of KDM5C in different cancers in the TCGA database. Red boxes indicate KDM5C.
Fig 1
Expression of KDM5C in various cancer cells
2.2 构建稳定敲除 KDM5C 的肾癌细胞株及其表型的检测
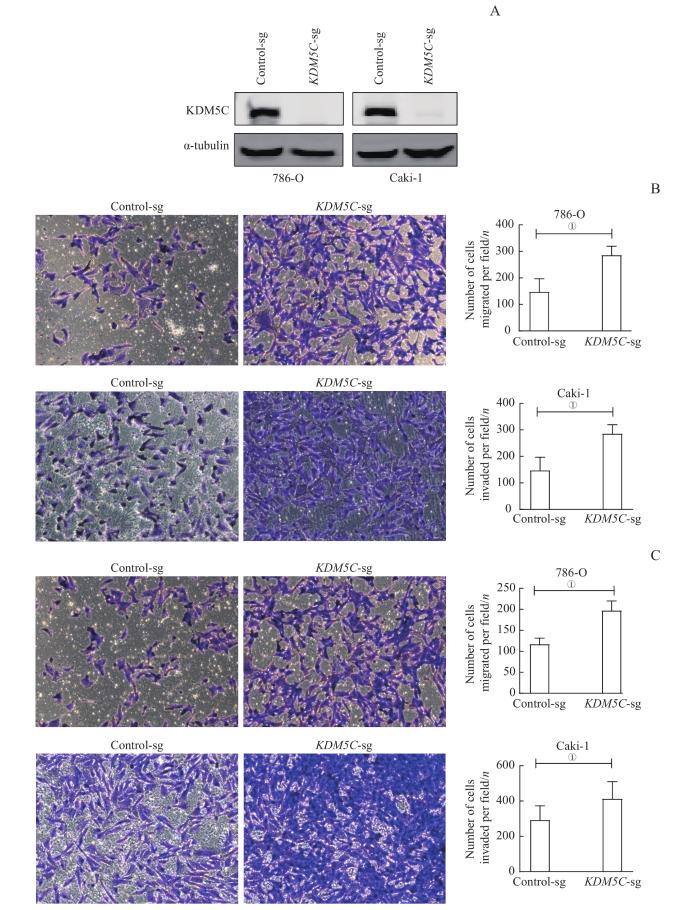
本研究通过转染Control-sg质粒、KDM5C-sg质粒、慢病毒感染靶细胞786-O、Caki-1构建筛选KDM5C稳定敲除的人肾癌细胞株;采用Western blotting检测KDM5C蛋白的表达,验证敲除效率。结果(图2A)表明KDM5C在靶细胞中表达明显降低,即稳转株制备成功。
图2
图2
KDM5C 敲除对肾透明细胞癌产生的影响
Note: A. Transfection efficiency of KDM5C-sgRNA in 786-O and Caki-1 verified by Western blotting. B. Effect of transfection of KDM5C-sgRNA plasmid on migration of 786-O and Caki-1 cells. C. Effect of transfection of KDM5C-sgRNA plasmid on invasion of 786-O and Caki-1 cells. ①P=0.000, compared with Control-sg.
Fig 2
Effect of KDM5C knockout on renal clear cell carcinoma cell
使用Transwell小室和Invasion小室对转染Control-sg组和KDM5C-sg组的786-O及Caki-1细胞的迁移和侵袭能力进行检测,结果(图2B、C)表明,KDM5C敲除后2组人肾癌细胞迁移和侵袭的能力均得到了增强(P=0.000),差异具有统计学意义。
2.3 RNA测序数据预测 KDM5C 突变后促癌的机制
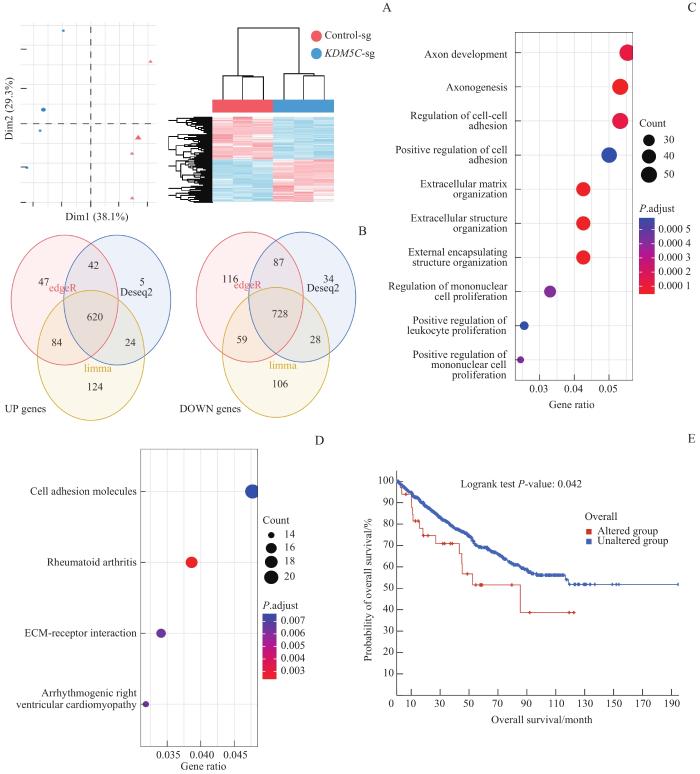
用R 4.2.0对786-O Control-sg和786-O KDM5C-sg 2组肾癌细胞株RNA测序数据进行差异分析。主成分分析和层次聚类结果(图3A)表明2组样品均被区分开,具有很好的差异性;Limma包、edgeR包和Deseq2包的差异分析结果(图3B、C)取交集后显示有620个上调基因(P<0.05且log2 FC>1)和728个下调基因(P<0.05且log2 FC<-1);接着对得到的差异基因进行GO功能分析(图3C),结果表明KDM5C敲除后,富集到了细胞黏附分子和细胞外基质结构,提示其功能改变可能与黏附迁移相关;最后进行KEGG富集分析(图3D),结果提示富集到细胞黏附分子通路以及EMT受体的相互作用通路,进一步提示KDM5C敲除后可能引起786-O细胞的转移能力得到增强。使用cBioportal在线网站分析了TCGA数据库里1 100位肾癌患者中KDM5C突变对预后的影响,其中KDM5C发生突变的患者有48位,未发生突变的有1 052位;结果(图3E)显示突变患者的预后情况较未突变的差(P=0.042),差异具有统计学意义。
图3
图3
KDM5C 敲除后肾癌细胞的RNA测序
Note: A. PCA plot and heatmap of the two groups. B. Venn plots of upregulated and downregulated genes in 786-O_KDM5C-sg cells compared to 786-O_-control-sg cells. C/D. Go analysis (C) and KEGG analysis (D) on the sequencing data. E. KM plot of the prognosis of KIRC patients with and without KDM5C mutation.
Fig 3
RNA sequencing analysis of renal carcinoma cells with KDM5C knockout
2.4 敲除 KDM5C 对EMT相关基因和蛋白的影响
采用Western blotting检测稳定敲除KDM5C的细胞中的EMT相关蛋白的表达情况,结果(图4)显示KDM5C敲除后786-O和Caki-1细胞内β-catenin、Vimentin、Snail蛋白表达水平均发生上调,使癌细胞更容易发生转移,与前述KDM5C敲除后人肾癌细胞迁移、侵袭能力增强的表型以及RNA测序富集到的功能通路一致。
图4
图4
KDM5C 敲除对EMT相关蛋白产生的影响
Fig 4
Effects of KDM5C knockout on EMT-related proteins expression
2.5 敲除 KDM5C 后上调的EMT基因的鉴定及其对预后的影响
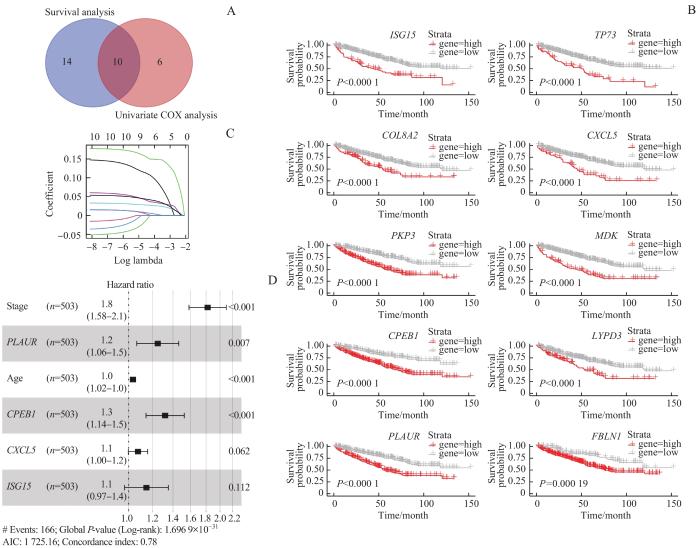
将RNA测序中的上调基因与数据库dbEMT 2.0(
图5
图5
KDM5C 敲除后上调的EMT相关基因对预后的影响
Note: A. Venn plot showed the common genes of survival analysis (P<0.01) and cox proportional-hazards model (P<0.01). B. KM plots showed the prognosis of 10 common genes in the TCGA database. C. LASSO coefficient profiles of genes significantly associated with OS. D. The forest plots showed the univariate COX analysis of the four genes significantly associated with OS.
Fig 5
Effect of up-regulated EMT-related gene on prognosis after KDM5C knockout
3 讨论
组蛋白的翻译后修饰作为一种表观遗传,在调控基因的功能、决定细胞的分化命运和维持细胞存活具有重要作用[12]。大多数的肾透明细胞癌的发生通常被认为需要两个过程,首先发生的是Von Hippel Lindau(VHL)基因的失活。单独的VHL基因失活会诱导细胞产生衰老,需要其他基因的进一步突变才能驱动肾透明细胞癌的发生发展,这一点也在细胞实验和小鼠动物模型中被证实[13-14]。其中,KDM5C基因突变则被列为了促其发生发展的重要因子,其他的易突变的基因还包括PBRM1、SETD2、BAP1、UTX和MLL2[4,15-16]。值得注意的是这些基因编码的蛋白大多都与表观遗传修饰相关,提示了表观遗传学的改变在肾透明细胞癌的进展中占有重要的地位。
本研究通过公共数据库分析发现KDM5C在肾透明细胞癌中具有较高的突变率,并通过构建稳定敲除KDM5C的人肾透明细胞癌细胞株检测其表型,发现其迁移和侵袭能力得到了增强,且EMT相关上皮标记基因在蛋白表达水平发生改变,提示失活的KDM5C会导致EMT相关基因表达失调。肾癌细胞RNA测序数据的分析发现KDM5C敲除后会导致癌细胞的黏附分子、EMT相关基因表达改变,与上述得到的表型一致。KDM5C作为组蛋白赖氨酸去甲基化酶,其失活后可导致调控的靶基因启动子区域的H3K4me3水平升高,促进靶基因的异常转录激活;对RNA测序后得到的上调的EMT相关基因进行单因素COX分析和生存分析,初步筛选出关联的基因,再通过LASSO回归分析、风险森林模型进一步缩小入选基因的范围,最终得到与KDM5C敲除引起上皮-间质转化高度相关的PLAUR基因(P=0.007),提示KDM5C可能对其产生调控作用,但具体的作用机制还有待后续的探索。PLAUR的表达上调与肿瘤转移密切相关,可见于胃癌[17]、胰腺癌[18]和宫颈癌[19];研究表明PLAUR编码的受体蛋白表达于恶性肿瘤中,而不表达于正常组织,与相应配体结合后可激活细胞外基质蛋白水解酶,促进肿瘤细胞发生转移。
综上所述,本研究提出组蛋白赖氨酸去甲基化酶KDM5C失活后通过干扰EMT相关基因表达、使黏附分子表达异常,导致肾透明细胞癌更易发生转移,进而促进癌症的发展。阐明KDM5C失活与转移表型之间的关系可以帮助我们进一步理解表观遗传修饰改变对肾透明癌进展的影响,同时也为改善临床具有该基因突变的患者的治疗效果提供新的思路。
作者贡献声明
郑英霞、陈宁黛参与了实验构思,陈宁黛、周冰倩、陈哲逸、陈诗宇参与了实验实施,郑英霞和陈宁黛参与了论文的写作和修改。所有作者均阅读并同意了最终稿件的提交。
AUTHOR's CONTRIBUTIONS
The study was designed by ZHEN Yingxia and CHEN Ningdai. The experiment was conducted by CHEN Ningdai, ZHOU Bingqian, CHEN Zheyi, and CHEN Shiyu. The manuscript was drafted and revised by ZHEN Yingxia and CHEN Ningdai. All the authors have read the last version of paper and consented for submission.
利益冲突声明
所有作者声明不存在利益冲突。
COMPETING INTERESTS
All authors disclose no relevant conflict of interests.
参考文献