癌-睾丸抗原(cancer-testis antigen,CTA)是一类在恶性肿瘤和睾丸组织中高表达,而在其他正常组织中不表达的特定蛋白,通常被视为肿瘤生物标志物和免疫治疗靶点[1]。迄今已有数百个CTA被鉴定报道,并且大部分CTA以扩增子(amplicon)的形式分布于多个染色体的不同区域。肉瘤抗原1(sarcoma antigen 1,SAGE1)是CTA的重要组分之一,位于X染色体长臂(Xq26.3)区域,与紧邻的另一CTA组分——CT45A(CTA family 45 member A)家族串联基因阵列一起,共同构成了CT45A-SAGE1扩增子结构。该扩增子结构的两侧被2个单拷贝基因INTS6L和MMGT1所包围隔离。从进化角度而言,CT45A-SAGE1扩增子的祖先基因肇始于胎盘类哺乳动物,然而SAGE1基因被发现仅仅存在于灵长类动物的基因组中,说明CT45A-SAGE1扩增子在生物进化上属于一个非常新的基因家族。从表达模式而言,生理条件下SAGE1仅在睾丸和垂体等少数组织中高表达,在其他组织器官中不表达或者痕量表达,然而在诸如肺癌、膀胱癌以及头颈癌等多种恶性肿瘤中SAGE1均被发现过量表达[2]。值得注意的是,尽管有报道明确表明SAGE1的异常激活与肿瘤进展、侵袭性和不良预后之间存在密切关联,但是目前对于SAGE1的生理功能、结构基础、相互作用网络等等分子机制方面的认知甚少,有待进一步深入的挖掘和系统性的研究[3]。
我们使用 InterProScan(
整合体复合物(integrator complex,INT)是一种由14个亚基组成的大分子复合物,能够结合Pol Ⅱ,参与mRNA剪接、3'端加工和转录终止等过程[6-7]。有报道[8]明确表明,该复合物中INTS6亚基的羧基末端结构域(INTS6CTD)与INTS3亚基的羧基末端结构域(INTS3CTD)之间存在直接相互作用。同时也有研究[9-10]报道,INTS6可以和INTS3、单链DNA结合蛋白1(human single stranded DNA binding protein 1,hSSB1)相互结合,形成INTS6-INTS3-hSSB1亚复合物,参与DNA的损伤修复。有趣的是,我们发现INTS6CTD与SAGE1CTD之间具有较高的序列同源性,基于INTS6CTD与INTS3CTD之间的结合模式信息,我们合理地推测SAGE1与INTS3之间极有可能也存在直接相互作用,并且该相互作用在灵长类动物中具有普遍性。
相较于INT复合物这种在多细胞真核生物中古老又保守的基因家族而言,SAGE1是灵长类动物特有的、具有生物进化价值的一类特殊基因。在癌症研究中,灵长类动物由于其与人类在进化上的相似性[11-13],能够更为准确地模拟人类疾病的生物学特性[14-15]。因此通过对不同灵长类动物SAGE1蛋白的研究,可以进一步了解SAGE1的进化及其生理病理意义。进化上灵长类动物大致可以分为:猿类(人和黑猩猩等为代表)、旧世界猴类[恒河猴(Rhesus)和食蟹猴等为代表]、新世界猴类[狨猴(Marmoset)和卷尾猴等为代表]以及原猴类(狐猴和眼镜猴等为代表)四大类别。在我们实验室对人源SAGE1前期工作探索的基础上,我们选择恒河猴、狨猴和倭狐猴(Mouse Lemur,下文简称Lemur)这3种代表性灵长类动物的SAGE1作为研究对象展开相关研究工作[15-17]。通过构建哺乳动物细胞体外过表达质粒,采用免疫共沉淀的形式确认了3种灵长类动物的SAGE1与INTS3具有相互作用。通过大肠埃希菌(Escherichia coli,E. coli)外源蛋白表达体系和蛋白纯化体系,分别获得了SAGE1羧基末端截短体蛋白(SAGE1CTD)、INTS3羧基末端截短体蛋白(INTS3CTD)的生物样品,然后借助表面等离子体共振技术分析了它们的结合动力学特性,并使用广角激光散射测定了INTS3CTD与SAGE1CTD的结合比例。此外,我们还利用分子对接工具,预测了INTS3CTD-SAGE1CTD复合物的三维结构模型,并将其与INTS6羧基末端截短体蛋白(INTS6CTD)与INTS3CTD形成的INTS3CTD-INTS6CTD复合物进行比较。我们的研究结果证明了灵长类动物中INTS3与SAGE1之间的相互作用具有极高的保守性,提供了对SAGE1在灵长类动物中出现和进化的初步理解,为后续SAGE1相关机制研究、完整的INTS3与SAGE1复合物的高分辨率结构的解析等奠定了坚实的理论基础。
1 材料与方法
1.1 实验材料
1.1.1 表达载体、菌株与细胞
pGEX-6P1载体、pET-28a载体、pcDNA3.4-3×FLAG-N载体、pcDNA3.4-STREP-N载体、E. coli Rosetta(DE3)菌株、E. coli DH5α菌株、HEK293T细胞均源自本实验室保存。
1.1.2 实验试剂与主要仪器
限制性核酸内切酶、T4 DNA连接酶(Thermo Fisher,美国),无缝克隆重组酶(ClonExpress Ⅱ,南京诺唯赞),位点特异性突变试剂盒(QuikChange Ⅱ,Agilent;美国),三羟基甲基氨基甲烷-盐酸(Tris-HCl)、氯化钠(NaCl)、甘油、乙二胺四乙酸(EDTA)、乙二醇双四乙酸(EGTA)、磷酸缓冲液(PBS)、吐温-20(Tween-20)、聚乙二醇辛基苯基醚(Triton-X-100)、二硫苏糖醇(DTT)、4-羟乙基哌嗪乙磺酸(HEPES)、苯甲基磺酰氟(PMSF)、亮肽素、苯甲脒、异丙基-β-D-硫代半乳糖苷(IPTG)、氨苄青霉素、卡那霉素、咪唑[生工生物工程(上海)股份有限公司],Cocktail蛋白酶抑制剂(Roche,瑞士),DMEM培养基(Thermo Fisher,美国)、胎牛血清(FBS,Gibico;美国),脂质体转染试剂(Lipofectamine 3000,Thermo Fisher;美国),青霉素-链霉素双抗溶液(Thermo Fisher,美国),FLAG M2 beads(M8823,Sigma;美国),STREP Tactin XT resin(IBA,德国),LB培养基(青岛生工生物科技有限公司),考马斯亮蓝G250(P0017,碧云天),镍离子金属螯合亲和层析介质(Ni-NTA,Qiagen;德国),谷胱甘肽琼脂糖凝胶(glutathione sepharose 4B,Cytiva;美国);而重组酵母SUMO蛋白酶1(ULP1-His)以及GST-3C酶(PreScission Protease)均由本实验室表达、纯化、冻存。
冷冻离心机(5814R,Eppendorf,德国),电泳仪与电泳槽(Mini-PROTEAN,Bio-Rad;美国),超声波细胞破碎仪(VCX750,SONICS,美国),高速离心机(Avanti J-26XP,Beckman Coulter;美国),超声波清洗器(SB-5200DTN,宁波新芝生物),控温振荡培养箱(YR101,上海西雷生物),快速纯化液相色谱(fast protein liquid chromatography,FPLC)系统(ÄKTATM pure)、层析柱(SuperdexTM)、高通量分子互作分析系统(Biacore 8K)(Cytiva,美国),静态广角激光散射仪(DAWN HELEOS Ⅱ,Wyatt;美国)为实验室保有。
1.2 实验方法
1.2.1 引物、DNA合成和序列测定
均由生工生物工程(上海)股份有限公司完成。
1.2.2 质粒构建
委托生工生物工程(上海)股份有限公司进行DNA合成,获得了恒河猴、狨猴、倭狐猴和人类SAGE1的全长基因编码序列。将这些序列分别插入pcDNA3.4-3×FLAG-N质粒,得到全长SAGE1表达质粒,使用位点突变试剂盒将人类SAGE1的关键作用位点(M832、F838、Q840、R872、F873、K874)突变为丙氨酸,得到突变体SAGE1表达质粒;再将恒河猴、狨猴、倭狐猴和人类INTS3的全长基因编码序列,分别插入pcDNA3.4-STREP-N质粒,得到3种灵长类动物的转染用全长INTS3表达质粒。随后,利用PCR技术获得了3种灵长类动物SAGE1的CTD,并将SAGE1的CTD基因序列插入pGEX-6P1载体,这使得SAGE1的氨基端带上谷胱甘肽巯基转移酶(GST)纯化标签,得到恒河猴、狨猴和倭狐猴的氨基端带有谷胱甘肽巯基转移酶(GST)纯化标签的SAGE1CTD表达质粒。借助PCR获得了INTS3的CTD,并将其插入pET-28a载体,使得INTS3的氨基端带上His-SUMO纯化标签,获得氨基端带有His-SUMO纯化标签的INTS3CTD表达载体。
1.2.3 免疫共沉淀实验
本实验使用HEK293T作为哺乳动物细胞表达的细胞系,将状态良好的细胞按25%的密度铺培养皿培养,37 ℃培养6~12 h之后观察细胞状态良好即可进行转染。转染前再次观察细胞状态(密度控制在70%~80%),使用Lipo3000进行质粒转染。48 h后收集转染好的细胞,加入400 µL细胞裂解液(20 mmol/L Tris-HCl pH 7.4,150 mmol/L NaCl,10%甘油,0.5% Triton-X-100,1 mmol/L EDTA,1 mmol/L EGTA,4 µL Cocktail)轻柔重悬细胞,使用SONICS超声波细胞破碎仪以30%的功率超声处理。超声处理后的样品,在4 ℃下,16 900 ×g离心15 min,取40 µL上清液保存,剩余上清液加入到预平衡好的FLAG M2 beads/Strep resin中,4 ℃转盘上孵育过夜。使用冷冻离心机300 ×g离心1 min,吸弃上清液,用裂解液重悬清洗3次,每次离心1 min,离心弃上清液。加入40 µL 2×SDS-PAGE loading buffer(125 mmol/L Tris-HCl pH 6.8,10% SDS,0.2%溴酚蓝,25%甘油,2.5% DTT),99 ℃加热5 min变性,样品冻存于-20 ℃。样品通过蛋白质印迹法(Western blotting)进行鉴定。
1.2.4 蛋白表达与纯化
将含表达目的蛋白的Rosetta菌液用3 200 ×g离心15 min,弃上清液,用裂解液(50 mmol/L HEPES,400 mmol/L NaCl,10% 甘油,0.1 mmol/L亮肽素,1 mmol/L PMSF,1 mmol/L苯甲脒,cocktail,pH 8.0)重悬菌体后,采用超声法裂解菌体。菌体裂解后经39 000×g离心40 min,取上清液。表达His标签的蛋白用Ni-NTA介质在4 ℃低速旋转混合1 h,表达GST标签的蛋白用GST琼脂糖凝胶珠混合3 h,分别用含有咪唑的洗涤缓冲液(50 mmol/L HEPES,400 mmol/L NaCl,25 mmol/L咪唑,10%甘油,pH 8.0)和普通洗涤缓冲液(50 mmol/L HEPES,400 mmol/L NaCl,10%甘油,pH 8.0)洗去杂蛋白。再分别用含有咪唑的洗脱缓冲液(50 mmol/L HEPES,150 mmol/L NaCl,250 mmol/L 咪唑,pH 8.0)和含有GSH的洗脱缓冲液(50 mmol/L HEPES,150 mmol/L NaCl,300 mmol/L GSH,pH 8.0)洗脱介质得到目的蛋白。将目的蛋白浓缩至10 mL,使用离子交换层析和凝胶排阻层析分开收集流出组分,经十二烷基磺酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE)分析鉴定目的蛋白质。
1.2.5 表面等离子体共振
首先,该实验通过氨基羧基偶联的方式将SAGE1CTD蛋白偶联到仪器CM5芯片上。将SAGE1CTD蛋白溶解于仪器配套的不同pH的乙酸钠溶液中,进行pH筛选,以确定偶联最佳pH,确保最佳偶联效果。将SAGE1CTD蛋白用流动相缓冲液(50 mmol/L HEPES,150 mmol/L NaCl,pH 8.0)稀释10个梯度,使用配套甘氨酸作为再生液。选取数据最佳的几个梯度作为动力学分析,实验方法模板采用动力学法进行。动力学参数设定为:分析物流动相与固定相的接触时间为120 s,分离时间设定为180 s,流动相流速设置为30 µL/min。同时将梯度浓度输入到执行的实验循环信息中,实验结果将根据这些信息自动拟合结合解离曲线。该实验主要使用高通量分子互作分析系统Biacore 8K仪器来进行,数据分析采用Biacore insight evaluation软件。
1.2.6 广角激光散射
广角激光散射凝胶排阻层析(size-exclusion chromatography with multi-angle light scattering,SEC-MALS)是一种用于测量生物大分子的关键信息的技术,可以表征生物分子质量[18]。制备INTS3CTD-SAGE1CTD复合物样品2 mg/mL,使用上样针将样品上样到机器中,以SEC-MALS缓冲液(50 mmol/L HEPES,400 mmol/L NaCl,pH 8.0)作为流动相,样品在室温下以0.5 mL/min的流速在流动相中运行。使用ASTRA软件对SEC-MALS数据进行分析。
1.2.7 复合物结构预测与分析
ZDOCK server[22]是一个在线网络交互式服务器,用于执行蛋白质-蛋白质分子对接,模拟蛋白质与蛋白质之间的相互作用,并生成可能的复合物结构。这有助于理解蛋白质复合物的构建方式,以及预测潜在的相互作用界面。上传已有RoseTTAFold预测出的SAGE1CTD蛋白质结构文件与生物数据库已有的INTS3CTD晶体结构文件(PDB id:6WLG),设置好默认参数,ZDOCK会搜索和分析三维空间中INTS3CTD和SAGE1CTD之间所有可能的结合姿态,进行对接模拟。
2 结果
2.1 SAGE1的进化及其与INTS3相互作用的保守性
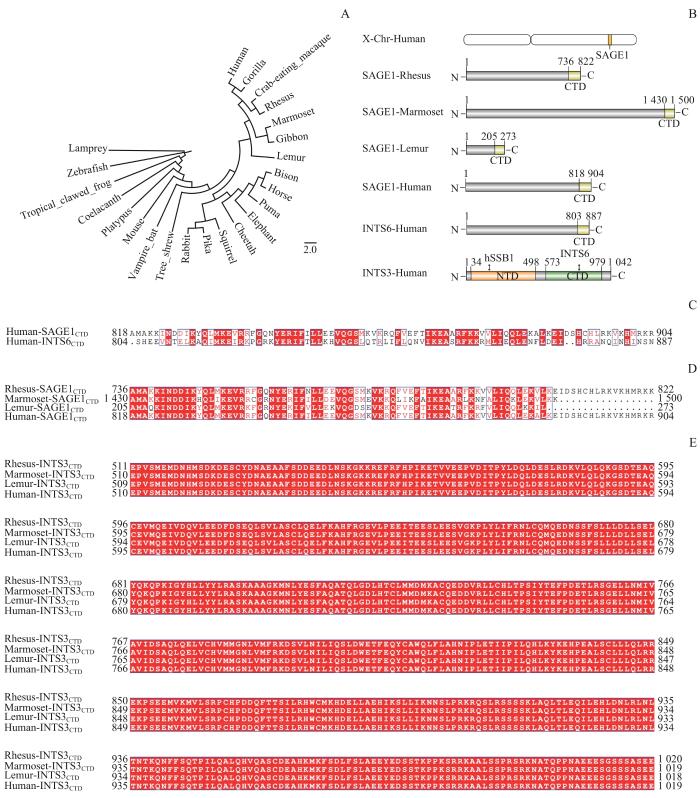
借助同源搜索,我们定义到INTS6是与SAGE1最接近的一个蛋白。为了探究SAGE1的进化,我们选择了多种代表性物种并构建了INTS6的进化谱系树。分析结果表明,INTS6是一个古老的基因,它的起源可以追溯到多细胞真核生物的早期进化阶段。从七鳃鳗到人类,脊椎动物的所有谱系都能观察到INTS6的存在(图1A)。我们得到一个线索,SAGE1或许是灵长类动物INTS6演化的一个分支。提出这一假设是因为我们在3种不同的灵长类动物的SAGE1中都识别出了其独特的CTD(图1B),而这一结构域在INTS6中也有所体现。氨基酸序列比对表明人类的SAGE1CTD和INTS6CTD在序列上呈现出部分保守性(图1C),并且这一发现不仅只局限于人类,进一步的序列比对揭示了人类与灵长类动物的SAGE1CTD也具有相当高的保守性(图1D)。与此同时,我们观察到INTS3CTD在灵长类动物中也展现了高度的保守性(图1E)。已有的研究显示了INTS6CTD与INTS3CTD之间的相互作用能力。基于这些发现,我们推测SAGE1极有可能与INTS3结合,形成相应的蛋白质复合物。
图1
图1
SAGE1与INTS6在进化上的相似性
Note: A. Evolution tree of INTS6. B. The SAGE1 full-length sequence and C-terminal sequence domain pattern map of four species of human, rhesus, marmoset and mouse lemur, and the full-length domain pattern map of human INTS3 and INTS6. C. Sequence alignment between human SAGE1CTD and INTS6CTD. D. Sequence alignment between human SAGE1CTD and three different primates SAGE1CTD. E. Sequence alignment between human INTS3CTD and three different primates INTS3CTD.
Fig 1
Evolutionary similarity between SAGE1 and INTS6
2.2 3种灵长类SAGE1与INTS3存在相互作用且作用力强度相似
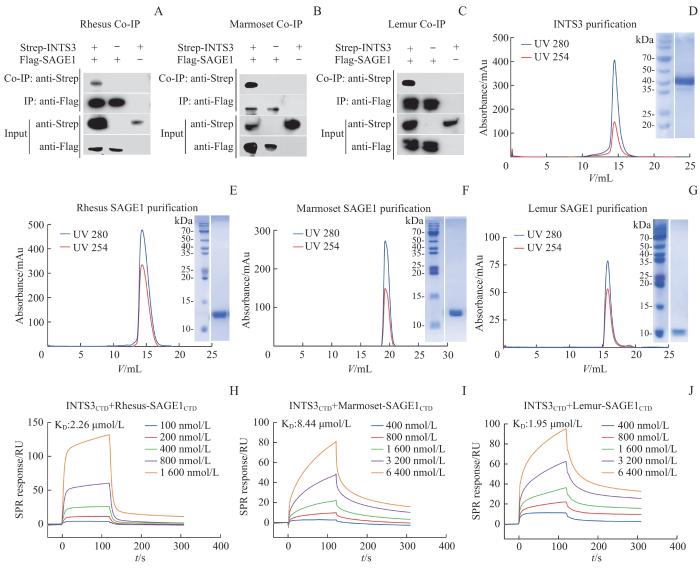
为了验证灵长类动物SAGE1与INTS3之间存在潜在相互作用,我们进行了免疫共沉淀分析。研究结果显示,恒河猴的INTS3与其SAGE1存在相互作用(图2A)。该结果同样出现在狨猴(图2B)和倭狐猴(图2C)免疫共沉淀样品组中。为了研究不同灵长类动物中INTS3CTD和SAGE1CTD之间的相互作用强度以及结合或解离速度的差异,我们纯化得到了INTS3CTD蛋白质(图2D)和3种灵长类动物的SAGE1CTD蛋白质(图2E~G),并采用了表面等离子体共振技术来评估这2种蛋白在体外相互作用的结合强度。通过动力学实验,我们获得了3种灵长类动物SAGE1CTD与INTS3CTD之间的结合解离曲线(图2H~J),根据动力学分析,观察到不同浓度下的表面等离子体共振信号响应在一段时间内缓慢上升,达到最大值后缓慢下降。这表明SAGE1CTD与INTS3CTD之间的相互作用呈现出慢结合和慢解离的趋势。为了进行比较,我们选择了基线稳定的空白组作为对照,并选取了5个响应值较合理的浓度梯度进行分析。通过分析软件计算,得到了恒河猴SAGE1CTD与INTS3CTD的解离常数为2.26 µmol/L,狨猴为8.44 µmol/L,倭狐猴为1.95 µmol/L。3种灵长类动物的平衡解离常数都在微摩尔数量级浮动,表明它们之间的结合强度非常相似。
图2
图2
SAGE1与INTS3的免疫共沉淀实验、蛋白纯化及解离常数测定
Note: A. Immunoprecipitation of full-length INTS3 and full-length SAGE1 in Rhesus. B. Immunoprecipitation of full-length INTS3 and full-length SAGE1 in Marmoset. C. Immunoprecipitation of full-length INTS3 and full-length SAGE1 in Lemur. D. Purification of human INTS3CTD. E. Purification of Rhesus SAGE1CTD. Gel filtration chromatography results shown on the left; SDS-PAGE analysis of fractions across the major peak shown on the right. F. Purification of Marmoset SAGE1CTD. G. Purification of Lemur SAGE1CTD. H. Rhesus SAGE1CTD binding to INTS3CTD in a concentration-dependent manner with a slow kinetics. I. Marmoset SAGE1CTD binding to INTS3CTD in a concentration-dependent manner with a slow kinetics. J. Lemur SAGE1CTD binding to INTS3CTD in a concentration-dependent manner with a slow kinetics. Co-IP—Co-immunoprecipitation; SPR—surface plasmon resonance.
Fig 2
Co-immunoprecipitation experiment, protein purification and dissociation constant determination of SAGE1 and INTS3
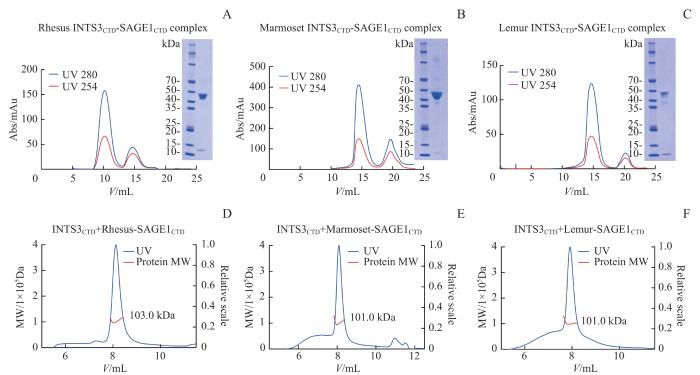
图3
图3
INTS3CTD-SAGE1CTD 复合物体外组装与SEC-MALS分析
Note: A. Rhesus INTS3CTD-SAGE1CTD complex assemble in vitro. B. Marmoset INTS3CTD-SAGE1CTD complex assemble in vitro. C. Lemur INTS3CTD-SAGE1CTD complex assemble in vitro. D. SEC-MALS analysis of purified Rhesus INTS3CTD-SAGE1CTD complex. E. SEC-MALS analysis of purified Marmoset INTS3CTD-SAGE1CTD complex. F. SEC-MALS analysis of purified Lemur INTS3CTD-SAGE1CTD complex. MW—molecular weight.
Fig 3
In vitro INTS3CTD-SAGE1CTD assembly and SEC-MALS analysis
2.3 INTS3与SAGE1的结合比例
通过SEC-MALS分析纯化的复合物(图3D~F),结果显示3种灵长类动物均表现出清晰的信号,并在8 mL流动相体积处产生吸收峰。同时,紫外线检测显示了单峰信号的特征,再次验证了INTS3CTD-SAGE1CTD复合物的形成。具体而言,恒河猴INTS3CTD-SAGE1CTD复合物的分子质量测定为103.0 kDa(单个INTS3CTD理论分子质量为47.1 kDa,单个恒河猴SAGE1CTD理论分子质量为10.5 kDa);而狨猴和倭狐猴的INTS3CTD-SAGE1CTD复合物分子质量都测定为101.0 kDa(单个狨猴、倭狐猴SAGE1CTD理论分子质量为8.3 kDa),所有检测结果均在可接受的误差范围内。该结果提示,INTS3与SAGE1以2∶1的比例形成复合物,和INTS3与INTS6的作用模式相同。
2.4 INTS3CTD-SAGE1CTD 复合物初步结构分析
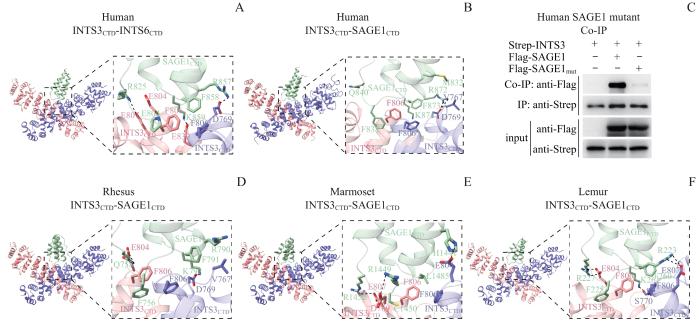
根据公共数据库中已有的INTS3CTD-INTS6CTD结构(PDB id:7BV7),我们推测3种灵长类动物和人类的INTS3CTD-SAGE1CTD复合物也具有相似的结构。为了获取它们的三维结构信息,我们借助蛋白质结构预测工具RoseTTAFold2,将SAGE1CTD的氨基酸序列输入进云端结构模拟程序,然后与PDB数据库中的INTS3CTD(PDB id:6WLG)进行分子对接。结果显示,该复合物无一例外呈现出典型的蝴蝶形结构(图4),其中“蝴蝶”的头部由SAGE1CTD的4个α-螺旋组成,而“身体”则由2个INTS3CTD蛋白相互交叉形成二聚体。同时,我们将其与INTS3CTD-INTS6CTD结构进行了比较,发现对接结果与该结构高度相似(图4A),SAGE1与INTS6和INTS3的相互作用界面有一定重叠性。
图4
图4
INTS3CTD-SAGE1CTD 结构预测与相互作用界面分析、INTS3与人类突变体SAGE1免疫共沉淀实验
Note: A. The Human INTS3CTD-INTS6CTD structure published on PDB (id: 7BV7) and its interaction interface. H-bond is marked by black dash line. B. The Human INTS3CTD-SAGE1CTD model created by molecular docking and its interaction interface. C. Immunoprecipitation of full-length INTS3 and full-length SAGE1 mutant in Human. D. The Rhesus INTS3CTD-SAGE1CTD model created by molecular docking and its interaction interface. E. The Marmoset INTS3CTD-SAGE1CTD model created by molecular docking and its interaction interface. F. The Lemur INTS3CTD-SAGE1CTD model created by molecular docking and its interaction interface.
Fig 4
Structure prediction and interaction interface analysis of INTS3CTD-SAGE1CTD, and co-immunoprecipitation experiment of INTS3 with human mutant SAGE1
通过对人类INTS3CTD-SAGE1CTD复合物模拟结构分析,我们发现其相互作用主要由疏水相互作用介导,并伴有亲水相互作用辅助(图4B右)。随后,我们挑选了相互作用界面上6个关键互作位点(M832、F838、Q840、R872、F873、K874),将其全部突变为丙氨酸后,利用免疫共沉淀方法检测INTS3与突变体SAGE1的互作情况(图4C),结果显示,突变体SAGE1与INTS3的互作明显下降,这也验证了对接结果的可靠性。并且进一步观察分析后,我们发现这些位点大多在灵长类中具有保守性。3种灵长类动物INTS3CTD和SAGE1CTD的作用界面也主要由疏水相互作用介导,即恒河猴(图4D右)SAGE1CTD的F791、狨猴(图4E右)SAGE1CTD的L1485和倭狐猴(图4F右)SAGE1CTD的F260与2个INTS3CTD的F806参与形成疏水作用。此外,还有一些氨基酸残基形成了离子键,如位于恒河猴SAGE1CTD中的K792与INTS3CTD的D769,位于狨猴SAGE1CTD中的H1441与INTS3CTD的E804,位于倭狐猴SAGE1CTD中的R227与INTS3CTD的E804,它们之间的相互作用被氢键进一步加强。在3种灵长类动物以及人类INTS3CTD-SAGE1CTD复合物结构上具有一定保守性,都呈现一种“蝴蝶”状结构,以疏水相互作用为主,辅助亲水相互作用,同时SAGE1与INTS6和INTS3的相互作用界面有一定重叠性,暗示了可能存在着竞争性,需要未来进一步验证和探索。
3 讨论
我们使用了几种蛋白互作方法来验证SAGE1与INTS3之间的相互作用。首先,通过免疫共沉淀和蛋白质印迹的方法,我们证实了全长INTS3与3种灵长类动物全长SAGE1之间存在明确的相互作用,暗示了INTS3和SAGE1在基因表达和调控中的协同作用,并且这种作用在进化过程中是高度保守的。通过使用表面等离子体共振技术,得到INTS3-SAGE1的解离常数,并且其处在微摩尔级别,显示出较高的亲和性。这种级别的相互作用力可能在生物学上有其特定的重要性,需要进一步的实验来揭示。根据SEC-MALS的结果,INTS3-SAGE1复合物的组成比例为2∶1。此外,通过分子对接预测,我们揭示了截短体INTS3CTD-SAGE1CTD复合物的蛋白质结构模型。复合物的“蝴蝶型结构”提供了一个全新的视角,进一步解释了INTS3和SAGE1是如何进行结合的。此外,INTS3的二聚体形式可能对SAGE1的结合方式和其功能有重要的影响。
研究表明,INTS3具备与SAGE1和INTS6的相互作用能力,并且它们的相互作用界面存在一定的重叠,这暗示了SAGE1与INTS6之间可能存在潜在的竞争关系。INTS3作为整合体复合物的组成部分,在RNA的剪接、加工等生物过程中发挥关键作用。SAGE1与INTS6竞争性与INTS3结合可能影响转录功能的正常执行,进而调节相关基因的表达。这一发现对未来的转录调控分子机制研究具有重要的意义。
针对SAGE1的探讨显示,从低等灵长类到高等灵长类,其C端具有保守性,可能与INTS6竞争性结合INTS3,而N端呈现出显著的变异,通过模式图展示,倭狐猴、狨猴、恒河猴、人类的SAGE1 N端分别为204、1 429、735、805个氨基酸。在漫长的进化过程中,N端长度一直在不断地“优化”,最终塑造了人类SAGE1目前的N端长度,从而更为有效地执行其功能。
综上所述,本研究通过探索SAGE1与INTS3之间的相互作用,提供了对SAGE1起源的新见解,并为未来更深入地探讨它们的生物学功能打下了基础。为了更好地理解这两个蛋白质如何协同工作并参与到具体的生物过程中,还需要更多的实验验证和分析。进一步的结构生物学研究,可能为我们提供更多关于这两个蛋白质如何结合并发挥其功能的信息。
作者贡献声明
武健设计并指导了全部课题的研究,李晓畅完成了主要实验过程的实施、实验数据处理与文章撰写,李明月和雷鸣负责实验方案指导和实验结果分析指导以及整篇论文的修改。所有作者均阅读并同意最终稿件的提交。
AUTHOR's CONTRIBUTIONS
WU Jian designed and guided the whole research project. LI Xiaochang mainly completed the work of experiment execution, data analysis and processing and article writing. LI Mingyue and LEI Ming were mainly responsible for the guidance of experimental scheme and data analysis, as well as the revision of the whole paper. All authors have read the final manuscript and approved the submission.
利益冲突声明
所有作者声明不存在利益冲突。
COMPETING INTERESTS
All authors disclose no relevant conflict of interests.
参考文献