[1]
GBD 2019 LRI Collaborators. Age-sex differences in the global burden of lower respiratory infections and risk factors, 1990-2019: results from the Global Burden of Disease Study 2019[J]. Lancet Infect Dis, 2022, 22(11): 1626-1647.
[本文引用: 1]
[2]
Word Health Organization. Pneumonia[EB/OL]. [2023-01-10]. https://data.unicef.org/topic/child-health/pneumonia/.
[本文引用: 1]
[3]
PERIN J, MULICK A, YEUNG D, et al. Global, regional, and national causes of under-5 mortality in 2000-19: an updated systematic analysis with implications for the Sustainable Development Goals[J]. Lancet Child Adolesc Health, 2022, 6(2): 106-115.
[本文引用: 1]
[4]
SANGLA F, LEGOUIS D, MARTI P E, et al. One year after ICU admission for severe community-acquired pneumonia of bacterial, viral or unidentified etiology. What are the outcomes?[J]. PLoS One, 2020, 15(12): e0243762.
[本文引用: 1]
[5]
FERREIRA-COIMBRA J, SARDA C, RELLO J. Burden of community-acquired pneumonia and unmet clinical needs[J]. Adv Ther, 2020, 37(4): 1302-1318.
[本文引用: 1]
[6]
DROZ N, HSIA Y, ELLIS S, et al. Bacterial pathogens and resistance causing community acquired paediatric bloodstream infections in low-and middle-income countries: a systematic review and meta-analysis[J]. Antimicrob Resist Infect Control, 2019, 8: 207.
[本文引用: 1]
[7]
TORRES A, CILLONIZ C, NIEDERMAN M S, et al. Pneumonia[J]. Nat Rev Dis Primers, 2021, 7(1): 25.
[本文引用: 1]
[8]
GROUSD J A, RICH H E, ALCORN J F. Host-pathogen interactions in gram-positive bacterial pneumonia[J]. Clin Microbiol Rev, 2019, 32(3): e00107-e00118.
[本文引用: 1]
[9]
CHOUDHURI S. From Waddington′s epigenetic landscape to small noncoding RNA: some important milestones in the history of epigenetics research[J]. Toxicol Mech Methods, 2011, 21(4): 252-274.
[本文引用: 1]
[10]
LIU C, XU J H, CHEN Y H, et al. Characterization of genome-wide H3K27ac profiles reveals a distinct PM2.5 -associated histone modification signature[J]. Environ Health, 2015, 14: 65.
[本文引用: 1]
[11]
ZHAO L F, ZHANG M, BAI L R, et al. Real-world PM2.5 exposure induces pathological injury and DNA damage associated with miRNAs and DNA methylation alteration in rat lungs[J]. Environ Sci Pollut Res Int, 2022, 29(19): 28788-28803.
[本文引用: 1]
[12]
ZHANG Q, CAO X T. Epigenetic remodeling in innate immunity and inflammation[J]. Annu Rev Immunol, 2021, 39: 279-311.
[本文引用: 1]
[13]
YIN Y M, MORGUNOVA E, JOLMA A, et al. Impact of cytosine methylation on DNA binding specificities of human transcription factors[J]. Science, 2017, 356(6337): eaaj2239.
[本文引用: 1]
[14]
ZHONG Z H, FENG S H, DUTTKE S H, et al. DNA methylation-linked chromatin accessibility affects genomic architecture in Arabidopsis [J]. Proc Natl Acad Sci U S A, 2021, 118(5): e2023347118.
[本文引用: 1]
[15]
DEKKERS K F, NEELE A E, JUKEMA J W, et al. Human monocyte-to-macrophage differentiation involves highly localized gain and loss of DNA methylation at transcription factor binding sites[J]. Epigenetics Chromatin, 2019, 12(1): 34.
[本文引用: 1]
[16]
XIA Y Y, HE F, WU X Y, et al. GABA transporter sustains IL-1β production in macrophages[J]. Sci Adv, 2021, 7(15): eabe9274.
[本文引用: 1]
[17]
SINGER B D, MOCK J R, AGGARWAL N R, et al. Regulatory T cell DNA methyltransferase inhibition accelerates resolution of lung inflammation[J]. Am J Respir Cell Mol Biol, 2015, 52(5): 641-652.
[本文引用: 1]
[18]
MCGRATH-MORROW S A, NDEH R, HELMIN K A, et al. DNA methylation regulates the neonatal CD4+ T-cell response to pneumonia in mice[J]. J Biol Chem, 2018, 293(30): 11772-11783.
[本文引用: 1]
[19]
BANNISTER S, KIM B, DOMÍNGUEZ-ANDRÉS J, et al. Neonatal BCG vaccination is associated with a long-term DNA methylation signature in circulating monocytes[J]. Sci Adv, 2022, 8(31): eabn4002.
[本文引用: 1]
[20]
HEIKKINEN A, BOLLEPALLI S, OLLIKAINEN M. The potential of DNA methylation as a biomarker for obesity and smoking[J]. J Intern Med, 2022, 292(3): 390-408.
[本文引用: 1]
[21]
AMPOMAH P B, CAI B S, SUKKA S R, et al. Macrophages use apoptotic cell-derived methionine and DNMT3A during efferocytosis to promote tissue resolution[J]. Nat Metab, 2022, 4(4): 444-457.
[本文引用: 1]
[22]
COLE E, BROWN T A, PINKERTON K E, et al. Perinatal exposure to environmental tobacco smoke is associated with changes in DNA methylation that precede the adult onset of lung disease in a mouse model[J]. Inhal Toxicol, 2017, 29(10): 435-442.
[本文引用: 1]
[23]
CHEN H, LI G, CHAN Y L, et al. Maternal E-cigarette exposure in mice alters DNA methylation and lung cytokine expression in offspring[J]. Am J Respir Cell Mol Biol, 2018, 58(3): 366-377.
[本文引用: 1]
[24]
MILLÁN-ZAMBRANO G, BURTON A, BANNISTER A J, et al. Histone post-translational modifications - cause and consequence of genome function[J]. Nat Rev Genet, 2022, 23(9): 563-580.
[本文引用: 1]
[25]
DILLON S C, ZHANG X, TRIEVEL R C, et al. The SET-domain protein superfamily: protein lysine methyltransferases[J]. Genome Biol, 2005, 6(8): 227.
[本文引用: 1]
[26]
LI Y, LI G H, ZHANG L, et al. G9a promotes inflammation in Streptococcus pneumoniae induced pneumonia mice by stimulating M1 macrophage polarization and H3K9me2 methylation in FOXP1 promoter region[J]. Ann Transl Med, 2022, 10(10): 583.
[本文引用: 2]
[27]
WU S Q, TIAN X C, MAO Q, et al. Azithromycin attenuates wheezing after pulmonary inflammation through inhibiting histone H3K27me3 hypermethylation mediated by EZH2[J]. Clin Epigenetics, 2023, 15(1): 12.
[本文引用: 1]
[28]
NITSCH S, ZORRO SHAHIDIAN L, SCHNEIDER R. Histone acylations and chromatin dynamics: concepts, challenges, and links to metabolism[J]. EMBO Rep, 2021, 22(7): e52774.
[本文引用: 1]
[29]
NAGESH P T, HUSSAIN M, GALVIN H D, et al. Histone deacetylase 2 is a component of influenza A virus-induced host antiviral response[J]. Front Microbiol, 2017, 8: 1315.
[本文引用: 1]
[30]
MULLICAN S E, GADDIS C A, ALENGHAT T, et al. Histone deacetylase 3 is an epigenomic brake in macrophage alternative activation[J]. Genes Dev, 2011, 25(23): 2480-2488.
[本文引用: 1]
[31]
YAO Y, LIU Q P, ADRIANTO I, et al. Histone deacetylase 3 controls lung alveolar macrophage development and homeostasis[J]. Nat Commun, 2020, 11(1): 3822.
[本文引用: 1]
[32]
FENG Q Q, SU Z L, SONG S Y, et al. Histone deacetylase inhibitors suppress RSV infection and alleviate virus-induced airway inflammation[J]. Int J Mol Med, 2016, 38(3): 812-822.
[本文引用: 1]
[33]
DAI J P, GU L M, SU Y, et al. Inhibition of curcumin on influenza A virus infection and influenzal pneumonia via oxidative stress, TLR2/4, p38/JNK MAPK and NF-κB pathways[J]. Int Immunopharmacol, 2018, 54: 177-187.
[本文引用: 1]
[34]
LIU L, ZHOU X M, SHETTY S, et al. HDAC6 inhibition blocks inflammatory signaling and caspase-1 activation in LPS-induced acute lung injury[J]. Toxicol Appl Pharmacol, 2019, 370: 178-183.
[本文引用: 1]
[35]
FABIAN M R, SONENBERG N, FILIPOWICZ W. Regulation of mRNA translation and stability by microRNAs[J]. Annu Rev Biochem, 2010, 79: 351-379.
[本文引用: 1]
[36]
JIANG K F, YANG J, GUO S, et al. Peripheral circulating exosome-mediated delivery of miR-155 as a novel mechanism for acute lung inflammation[J]. Mol Ther, 2019, 27(10): 1758-1771.
[本文引用: 1]
[37]
ZHANG D, LEE H, WANG X Y, et al. A potential role of microvesicle-containing miR-223/142 in lung inflammation[J]. Thorax, 2019, 74(9): 865-874.
[38]
ZHANG X, HUANG F, YANG D Y, et al. Identification of miRNA-mRNA crosstalk in respiratory syncytial virus- (RSV-) associated pediatric pneumonia through integrated miRNAome and transcriptome analysis[J]. Mediators Inflamm, 2020, 2020: 8919534.
[本文引用: 1]
[39]
GONZALO-CALVO D D, BENÍTEZ I D, PINILLA L, et al. Circulating microRNA profiles predict the severity of COVID-19 in hospitalized patients[J]. Transl Res, 2021, 236: 147-159.
[本文引用: 1]
[40]
GARCÍA-HIDALGO M C, GONZÁLEZ J, BENÍTEZ I D, et al. Identification of circulating microRNA profiles associated with pulmonary function and radiologic features in survivors of SARS-CoV-2-induced ARDS[J]. Emerg Microbes Infect, 2022, 11(1): 1537-1549.
[本文引用: 1]
[41]
SUN Q Y, HAO Q Y, PRASANTH K V. Nuclear long noncoding RNAs: key regulators of gene expression[J]. Trends Genet, 2018, 34(2): 142-157.
[本文引用: 1]
[42]
RINN J L, CHANG H Y. Long noncoding RNAs: molecular modalities to organismal functions[J]. Annu Rev Biochem, 2020, 89: 283-308.
[本文引用: 1]
[43]
LIU S, LIU J Q, YANG X, et al. Cis -acting lnc-Cxcl2 restrains neutrophil-mediated lung inflammation by inhibiting epithelial cell CXCL2 expression in virus infection[J]. Proc Natl Acad Sci U S A, 2021, 118(41): e2108276118.
[本文引用: 1]
[44]
CHI X W, DING B C, ZHANG L J, et al. lncRNA GAS5 promotes M1 macrophage polarization via miR-455-5p/SOCS3 pathway in childhood pneumonia[J]. J Cell Physiol, 2019, 234(8): 13242-13251.
[本文引用: 1]
[45]
GU H Y, ZHU Y F, ZHOU Y, et al. LncRNA MALAT1 affects Mycoplasma pneumoniae pneumonia via NF-κB regulation[J]. Front Cell Dev Biol, 2020, 8: 563693.
[本文引用: 1]
[46]
MEMCZAK S, JENS M, ELEFSINIOTI A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency[J]. Nature, 2013, 495(7441): 333-338.
[本文引用: 1]
[47]
ZHAO T, ZHENG Y L, HAO D Z, et al. Blood circRNAs as biomarkers for the diagnosis of community-acquired pneumonia[J]. J Cell Biochem, 2019, 120(10): 16483-16494.
[本文引用: 1]
[48]
KHAN H N, BRANDS X, AUFIERO S, et al. The circular RNA landscape in specific peripheral blood mononuclear cells of critically ill patients with sepsis[J]. Crit Care, 2020, 24(1): 423.
[本文引用: 1]
[49]
HANSEN T B, JENSEN T I, CLAUSEN B H, et al. Natural RNA circles function as efficient microRNA sponges[J]. Nature, 2013, 495(7441): 384-388.
[本文引用: 1]
[50]
ARORA S, SINGH P, DOHARE R, et al. Unravelling host-pathogen interactions: ceRNA network in SARS-CoV-2 infection (COVID-19)[J]. Gene, 2020, 762: 145057.
[本文引用: 1]
[51]
QU Z Y, MENG F, SHI J Z, et al. A novel intronic circular RNA antagonizes influenza virus by absorbing a microRNA that degrades CREBBP and accelerating IFN-β production[J]. mBio, 2021, 12(4): e0101721.
[本文引用: 1]
[52]
BARBAGALLO D, PALERMO C I, BARBAGALLO C, et al. Competing endogenous RNA network mediated by circ_3205 in SARS-CoV-2 infected cells[J]. Cell Mol Life Sci, 2022, 79(2): 75.
[本文引用: 1]
[53]
ROUNDTREE I A, EVANS M E, PAN T, et al. Dynamic RNA modifications in gene expression regulation[J]. Cell, 2017, 169(7): 1187-1200.
[本文引用: 1]
[54]
AN Y Y, DUAN H. The role of m6 A RNA methylation in cancer metabolism[J]. Mol Cancer, 2022, 21(1): 14.
[本文引用: 1]
[55]
TONG J Y, WANG X F, LIU Y B, et al. Pooled CRISPR screening identifies m6 A as a positive regulator of macrophage activation[J]. Sci Adv, 2021, 7(18): eabd4742.
[本文引用: 1]
[56]
LIU Y H, LIU Z J, TANG H, et al. The N 6 -methyladenosine (m6 A)-forming enzyme METTL3 facilitates M1 macrophage polarization through the methylation of STAT1 mRNA[J]. Am J Physiol Cell Physiol, 2019, 317(4): C762-C775.
[本文引用: 1]
[57]
YU R Q, LI Q M, FENG Z H, et al. m6 A reader YTHDF2 regulates LPS-induced inflammatory response[J]. Int J Mol Sci, 2019, 20(6): 1323.
[本文引用: 1]
[58]
LU M J, ZHANG Z J, XUE M G, et al. N 6 -methyladenosine modification enables viral RNA to escape recognition by RNA sensor RIG-I[J]. Nat Microbiol, 2020, 5(4): 584-598.
[本文引用: 1]
[59]
LU M J, XUE M G, WANG H T, et al. Nonsegmented negative-sense RNA viruses utilize N 6 -methyladenosine (m6 A) as a common strategy to evade host innate immunity[J]. J Virol, 2021, 95(9): e01939-e01920.
[本文引用: 1]
1
... 肺炎是最常见的下呼吸道感染性疾病,也是影响全球健康的重要原因[1 ] .其中,5岁以下儿童是受累最严重的人群,每43 s就有1名儿童死于肺炎[2 ] ,死亡率仅次于早产相关并发症[3 ] .严重肺炎患者还可能留有不同程度的后遗症,如肺功能改变、认知能力减弱、运动耐受降低等,严重影响患者的生活质量[4 ] .值得注意的是,随着耐药病原体不断增加,部分患者即使在接受最佳抗菌治疗后死亡率仍较高[5 -6 ] .免疫系统包括固有免疫和适应性免疫,免疫细胞在识别病原体后活化增殖分化为效应细胞并产生多种效应分子,在机体抗感染免疫中发挥了关键作用,与个体肺炎易感性密切相关[7 -8 ] .因此,从机体免疫功能着手改善肺炎患者的生存和预后至关重要. ...
1
... 肺炎是最常见的下呼吸道感染性疾病,也是影响全球健康的重要原因[1 ] .其中,5岁以下儿童是受累最严重的人群,每43 s就有1名儿童死于肺炎[2 ] ,死亡率仅次于早产相关并发症[3 ] .严重肺炎患者还可能留有不同程度的后遗症,如肺功能改变、认知能力减弱、运动耐受降低等,严重影响患者的生活质量[4 ] .值得注意的是,随着耐药病原体不断增加,部分患者即使在接受最佳抗菌治疗后死亡率仍较高[5 -6 ] .免疫系统包括固有免疫和适应性免疫,免疫细胞在识别病原体后活化增殖分化为效应细胞并产生多种效应分子,在机体抗感染免疫中发挥了关键作用,与个体肺炎易感性密切相关[7 -8 ] .因此,从机体免疫功能着手改善肺炎患者的生存和预后至关重要. ...
1
... 肺炎是最常见的下呼吸道感染性疾病,也是影响全球健康的重要原因[1 ] .其中,5岁以下儿童是受累最严重的人群,每43 s就有1名儿童死于肺炎[2 ] ,死亡率仅次于早产相关并发症[3 ] .严重肺炎患者还可能留有不同程度的后遗症,如肺功能改变、认知能力减弱、运动耐受降低等,严重影响患者的生活质量[4 ] .值得注意的是,随着耐药病原体不断增加,部分患者即使在接受最佳抗菌治疗后死亡率仍较高[5 -6 ] .免疫系统包括固有免疫和适应性免疫,免疫细胞在识别病原体后活化增殖分化为效应细胞并产生多种效应分子,在机体抗感染免疫中发挥了关键作用,与个体肺炎易感性密切相关[7 -8 ] .因此,从机体免疫功能着手改善肺炎患者的生存和预后至关重要. ...
1
... 肺炎是最常见的下呼吸道感染性疾病,也是影响全球健康的重要原因[1 ] .其中,5岁以下儿童是受累最严重的人群,每43 s就有1名儿童死于肺炎[2 ] ,死亡率仅次于早产相关并发症[3 ] .严重肺炎患者还可能留有不同程度的后遗症,如肺功能改变、认知能力减弱、运动耐受降低等,严重影响患者的生活质量[4 ] .值得注意的是,随着耐药病原体不断增加,部分患者即使在接受最佳抗菌治疗后死亡率仍较高[5 -6 ] .免疫系统包括固有免疫和适应性免疫,免疫细胞在识别病原体后活化增殖分化为效应细胞并产生多种效应分子,在机体抗感染免疫中发挥了关键作用,与个体肺炎易感性密切相关[7 -8 ] .因此,从机体免疫功能着手改善肺炎患者的生存和预后至关重要. ...
1
... 肺炎是最常见的下呼吸道感染性疾病,也是影响全球健康的重要原因[1 ] .其中,5岁以下儿童是受累最严重的人群,每43 s就有1名儿童死于肺炎[2 ] ,死亡率仅次于早产相关并发症[3 ] .严重肺炎患者还可能留有不同程度的后遗症,如肺功能改变、认知能力减弱、运动耐受降低等,严重影响患者的生活质量[4 ] .值得注意的是,随着耐药病原体不断增加,部分患者即使在接受最佳抗菌治疗后死亡率仍较高[5 -6 ] .免疫系统包括固有免疫和适应性免疫,免疫细胞在识别病原体后活化增殖分化为效应细胞并产生多种效应分子,在机体抗感染免疫中发挥了关键作用,与个体肺炎易感性密切相关[7 -8 ] .因此,从机体免疫功能着手改善肺炎患者的生存和预后至关重要. ...
1
... 肺炎是最常见的下呼吸道感染性疾病,也是影响全球健康的重要原因[1 ] .其中,5岁以下儿童是受累最严重的人群,每43 s就有1名儿童死于肺炎[2 ] ,死亡率仅次于早产相关并发症[3 ] .严重肺炎患者还可能留有不同程度的后遗症,如肺功能改变、认知能力减弱、运动耐受降低等,严重影响患者的生活质量[4 ] .值得注意的是,随着耐药病原体不断增加,部分患者即使在接受最佳抗菌治疗后死亡率仍较高[5 -6 ] .免疫系统包括固有免疫和适应性免疫,免疫细胞在识别病原体后活化增殖分化为效应细胞并产生多种效应分子,在机体抗感染免疫中发挥了关键作用,与个体肺炎易感性密切相关[7 -8 ] .因此,从机体免疫功能着手改善肺炎患者的生存和预后至关重要. ...
1
... 肺炎是最常见的下呼吸道感染性疾病,也是影响全球健康的重要原因[1 ] .其中,5岁以下儿童是受累最严重的人群,每43 s就有1名儿童死于肺炎[2 ] ,死亡率仅次于早产相关并发症[3 ] .严重肺炎患者还可能留有不同程度的后遗症,如肺功能改变、认知能力减弱、运动耐受降低等,严重影响患者的生活质量[4 ] .值得注意的是,随着耐药病原体不断增加,部分患者即使在接受最佳抗菌治疗后死亡率仍较高[5 -6 ] .免疫系统包括固有免疫和适应性免疫,免疫细胞在识别病原体后活化增殖分化为效应细胞并产生多种效应分子,在机体抗感染免疫中发挥了关键作用,与个体肺炎易感性密切相关[7 -8 ] .因此,从机体免疫功能着手改善肺炎患者的生存和预后至关重要. ...
1
... 肺炎是最常见的下呼吸道感染性疾病,也是影响全球健康的重要原因[1 ] .其中,5岁以下儿童是受累最严重的人群,每43 s就有1名儿童死于肺炎[2 ] ,死亡率仅次于早产相关并发症[3 ] .严重肺炎患者还可能留有不同程度的后遗症,如肺功能改变、认知能力减弱、运动耐受降低等,严重影响患者的生活质量[4 ] .值得注意的是,随着耐药病原体不断增加,部分患者即使在接受最佳抗菌治疗后死亡率仍较高[5 -6 ] .免疫系统包括固有免疫和适应性免疫,免疫细胞在识别病原体后活化增殖分化为效应细胞并产生多种效应分子,在机体抗感染免疫中发挥了关键作用,与个体肺炎易感性密切相关[7 -8 ] .因此,从机体免疫功能着手改善肺炎患者的生存和预后至关重要. ...
1
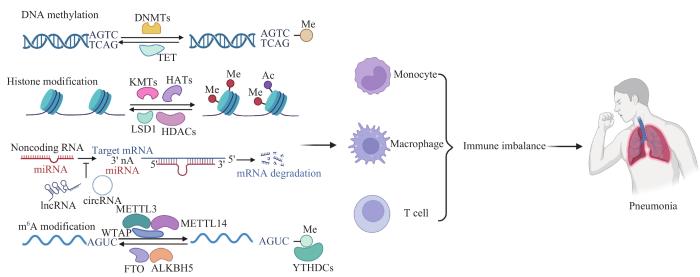
... 1942年,WADDINGTON提出“表观遗传学”的概念,并将其定义为表型变化而基因型不发生变化,用来解释发育过程中表型发生改变的机制,包括DNA甲基化、组蛋白修饰、非编码RNA、RNA修饰等[9 ] .表观遗传修饰仅有染色质结构或核酸化学性质的变化,而不涉及遗传密码改变,这一特征使其具有可塑性及灵活性,能快速响应环境变化,在调控免疫应答和维持免疫稳态过程中起着关键作用[10 -11 ] .越来越多的研究表明,表观遗传修饰调控的免疫激活和免疫抑制与肺部感染结局密切相关(图1 ):在肺部感染病原体后,这些表观遗传机制通过调节机体炎症反应和免疫应答,包括不同免疫细胞的发育和分化,感染信号的识别和传递,及针对病原体的细胞因子和效应分子的产生而影响不同个体肺炎的发生发展过程,在感染被清除后这种影响仍可持续存在[12 ] .同时,病原体可以利用表观遗传修饰来调节宿主和病原体基因的表达以进行免疫逃避.本文拟对近年来在肺炎发病过程中与感染免疫应答相关的表观遗传学研究进行总结,并探讨其在未来肺炎诊疗中的临床潜力. ...
1
... 1942年,WADDINGTON提出“表观遗传学”的概念,并将其定义为表型变化而基因型不发生变化,用来解释发育过程中表型发生改变的机制,包括DNA甲基化、组蛋白修饰、非编码RNA、RNA修饰等[9 ] .表观遗传修饰仅有染色质结构或核酸化学性质的变化,而不涉及遗传密码改变,这一特征使其具有可塑性及灵活性,能快速响应环境变化,在调控免疫应答和维持免疫稳态过程中起着关键作用[10 -11 ] .越来越多的研究表明,表观遗传修饰调控的免疫激活和免疫抑制与肺部感染结局密切相关(图1 ):在肺部感染病原体后,这些表观遗传机制通过调节机体炎症反应和免疫应答,包括不同免疫细胞的发育和分化,感染信号的识别和传递,及针对病原体的细胞因子和效应分子的产生而影响不同个体肺炎的发生发展过程,在感染被清除后这种影响仍可持续存在[12 ] .同时,病原体可以利用表观遗传修饰来调节宿主和病原体基因的表达以进行免疫逃避.本文拟对近年来在肺炎发病过程中与感染免疫应答相关的表观遗传学研究进行总结,并探讨其在未来肺炎诊疗中的临床潜力. ...
1
... 1942年,WADDINGTON提出“表观遗传学”的概念,并将其定义为表型变化而基因型不发生变化,用来解释发育过程中表型发生改变的机制,包括DNA甲基化、组蛋白修饰、非编码RNA、RNA修饰等[9 ] .表观遗传修饰仅有染色质结构或核酸化学性质的变化,而不涉及遗传密码改变,这一特征使其具有可塑性及灵活性,能快速响应环境变化,在调控免疫应答和维持免疫稳态过程中起着关键作用[10 -11 ] .越来越多的研究表明,表观遗传修饰调控的免疫激活和免疫抑制与肺部感染结局密切相关(图1 ):在肺部感染病原体后,这些表观遗传机制通过调节机体炎症反应和免疫应答,包括不同免疫细胞的发育和分化,感染信号的识别和传递,及针对病原体的细胞因子和效应分子的产生而影响不同个体肺炎的发生发展过程,在感染被清除后这种影响仍可持续存在[12 ] .同时,病原体可以利用表观遗传修饰来调节宿主和病原体基因的表达以进行免疫逃避.本文拟对近年来在肺炎发病过程中与感染免疫应答相关的表观遗传学研究进行总结,并探讨其在未来肺炎诊疗中的临床潜力. ...
1
... 1942年,WADDINGTON提出“表观遗传学”的概念,并将其定义为表型变化而基因型不发生变化,用来解释发育过程中表型发生改变的机制,包括DNA甲基化、组蛋白修饰、非编码RNA、RNA修饰等[9 ] .表观遗传修饰仅有染色质结构或核酸化学性质的变化,而不涉及遗传密码改变,这一特征使其具有可塑性及灵活性,能快速响应环境变化,在调控免疫应答和维持免疫稳态过程中起着关键作用[10 -11 ] .越来越多的研究表明,表观遗传修饰调控的免疫激活和免疫抑制与肺部感染结局密切相关(图1 ):在肺部感染病原体后,这些表观遗传机制通过调节机体炎症反应和免疫应答,包括不同免疫细胞的发育和分化,感染信号的识别和传递,及针对病原体的细胞因子和效应分子的产生而影响不同个体肺炎的发生发展过程,在感染被清除后这种影响仍可持续存在[12 ] .同时,病原体可以利用表观遗传修饰来调节宿主和病原体基因的表达以进行免疫逃避.本文拟对近年来在肺炎发病过程中与感染免疫应答相关的表观遗传学研究进行总结,并探讨其在未来肺炎诊疗中的临床潜力. ...
1
... DNA甲基化是在DNA甲基化转移酶的作用下,在基因组胞嘧啶-磷酸-鸟嘌呤(cytosine-phosphoric acid-guanine,CpG)二核苷酸的胞嘧啶5′碳端共价结合一个甲基基团,通过阻止转录因子与DNA的结合或诱导染色质结构改变而影响相关基因的表达.DNA甲基化是最早被发现,也是研究最深入的表观遗传调控机制[13 -14 ] . ...
1
... DNA甲基化是在DNA甲基化转移酶的作用下,在基因组胞嘧啶-磷酸-鸟嘌呤(cytosine-phosphoric acid-guanine,CpG)二核苷酸的胞嘧啶5′碳端共价结合一个甲基基团,通过阻止转录因子与DNA的结合或诱导染色质结构改变而影响相关基因的表达.DNA甲基化是最早被发现,也是研究最深入的表观遗传调控机制[13 -14 ] . ...
1
... 研究发现,DNA甲基化异常在肺炎免疫失衡中扮演了重要角色,可通过调控多种免疫细胞功能来影响肺炎转归.巨噬细胞是肺部感染和炎症反应中最为重要的固有免疫细胞,其分化发育与功能执行都与DNA甲基化密切相关.DEKKERS等[15 ] 的研究发现,巨噬细胞的分化和激活伴随着大量差异甲基化的基因位点和显著的表观遗传重构.γ-氨基丁酸转运蛋白2(γ-aminobutyric acid transporter 2,GAT2)可通过增强巨噬细胞基因启动子区域内的DNA甲基化来促进巨噬细胞中的重要促炎因子白细胞介素-1β(interleukin-1β,IL-1β)的产生,而GAT2缺乏会减弱巨噬细胞介导的体内炎症反应[16 ] .除巨噬细胞外,DNA甲基化还可通过调控调节性T细胞(regulatory T cell,Treg)来参与肺炎的损伤修复.SINGER等[17 ] 在研究流感病毒诱导的小鼠肺炎中发现,接受DNA甲基转移酶抑制剂治疗的小鼠肺部Treg数量增加,并且肺部炎症得到有效缓解.此外,MCGRATH-MORROW等[18 ] 的研究发现,新生小鼠CD4+ T细胞通路关键基因包括BTB与CNC同源基因2(BTB and CNC homology 2,Bach2 )、富集AT序列的特异性结合蛋白1(special AT rich sequence binding protein-1,Satb1 )、Tet1 (ten eleven translocation 1)等启动子的DNA甲基化, 抑制了新生小鼠对肺炎的免疫反应,表明DNA甲基化可能在肺CD4+ T细胞的成熟和对感染的反应中发挥了重要作用,可作为新生儿肺炎潜在的治疗靶点.BANNISTER等[19 ] 对新生儿接种卡介苗后的外周血单核细胞基因序列分析中发现了687个差异甲基化位点,包括RNA编辑酶腺苷酸脱氨酶(adenosine deaminase acting on RNA,ADAR )、干扰素诱导的跨膜蛋白1(interferon-induced transmembrane protein 1,IFITM1 )、信号转导与转录激活因子1(signal transduction and activator of transcription 1,STAT1 )、干扰素调节因子1(interferon regulatory factor 1,IRF1 )和IRF7 等,这些基因主要富集于信号通路,揭示了新生儿卡介苗疫苗接种与单核细胞的DNA甲基化特征的相关性.多项研究已经证明了DNA甲基化在肺部感染免疫中的重要调控作用,但其具体的分子机制及临床应用转化仍需进一步研究. ...
1
... 研究发现,DNA甲基化异常在肺炎免疫失衡中扮演了重要角色,可通过调控多种免疫细胞功能来影响肺炎转归.巨噬细胞是肺部感染和炎症反应中最为重要的固有免疫细胞,其分化发育与功能执行都与DNA甲基化密切相关.DEKKERS等[15 ] 的研究发现,巨噬细胞的分化和激活伴随着大量差异甲基化的基因位点和显著的表观遗传重构.γ-氨基丁酸转运蛋白2(γ-aminobutyric acid transporter 2,GAT2)可通过增强巨噬细胞基因启动子区域内的DNA甲基化来促进巨噬细胞中的重要促炎因子白细胞介素-1β(interleukin-1β,IL-1β)的产生,而GAT2缺乏会减弱巨噬细胞介导的体内炎症反应[16 ] .除巨噬细胞外,DNA甲基化还可通过调控调节性T细胞(regulatory T cell,Treg)来参与肺炎的损伤修复.SINGER等[17 ] 在研究流感病毒诱导的小鼠肺炎中发现,接受DNA甲基转移酶抑制剂治疗的小鼠肺部Treg数量增加,并且肺部炎症得到有效缓解.此外,MCGRATH-MORROW等[18 ] 的研究发现,新生小鼠CD4+ T细胞通路关键基因包括BTB与CNC同源基因2(BTB and CNC homology 2,Bach2 )、富集AT序列的特异性结合蛋白1(special AT rich sequence binding protein-1,Satb1 )、Tet1 (ten eleven translocation 1)等启动子的DNA甲基化, 抑制了新生小鼠对肺炎的免疫反应,表明DNA甲基化可能在肺CD4+ T细胞的成熟和对感染的反应中发挥了重要作用,可作为新生儿肺炎潜在的治疗靶点.BANNISTER等[19 ] 对新生儿接种卡介苗后的外周血单核细胞基因序列分析中发现了687个差异甲基化位点,包括RNA编辑酶腺苷酸脱氨酶(adenosine deaminase acting on RNA,ADAR )、干扰素诱导的跨膜蛋白1(interferon-induced transmembrane protein 1,IFITM1 )、信号转导与转录激活因子1(signal transduction and activator of transcription 1,STAT1 )、干扰素调节因子1(interferon regulatory factor 1,IRF1 )和IRF7 等,这些基因主要富集于信号通路,揭示了新生儿卡介苗疫苗接种与单核细胞的DNA甲基化特征的相关性.多项研究已经证明了DNA甲基化在肺部感染免疫中的重要调控作用,但其具体的分子机制及临床应用转化仍需进一步研究. ...
1
... 研究发现,DNA甲基化异常在肺炎免疫失衡中扮演了重要角色,可通过调控多种免疫细胞功能来影响肺炎转归.巨噬细胞是肺部感染和炎症反应中最为重要的固有免疫细胞,其分化发育与功能执行都与DNA甲基化密切相关.DEKKERS等[15 ] 的研究发现,巨噬细胞的分化和激活伴随着大量差异甲基化的基因位点和显著的表观遗传重构.γ-氨基丁酸转运蛋白2(γ-aminobutyric acid transporter 2,GAT2)可通过增强巨噬细胞基因启动子区域内的DNA甲基化来促进巨噬细胞中的重要促炎因子白细胞介素-1β(interleukin-1β,IL-1β)的产生,而GAT2缺乏会减弱巨噬细胞介导的体内炎症反应[16 ] .除巨噬细胞外,DNA甲基化还可通过调控调节性T细胞(regulatory T cell,Treg)来参与肺炎的损伤修复.SINGER等[17 ] 在研究流感病毒诱导的小鼠肺炎中发现,接受DNA甲基转移酶抑制剂治疗的小鼠肺部Treg数量增加,并且肺部炎症得到有效缓解.此外,MCGRATH-MORROW等[18 ] 的研究发现,新生小鼠CD4+ T细胞通路关键基因包括BTB与CNC同源基因2(BTB and CNC homology 2,Bach2 )、富集AT序列的特异性结合蛋白1(special AT rich sequence binding protein-1,Satb1 )、Tet1 (ten eleven translocation 1)等启动子的DNA甲基化, 抑制了新生小鼠对肺炎的免疫反应,表明DNA甲基化可能在肺CD4+ T细胞的成熟和对感染的反应中发挥了重要作用,可作为新生儿肺炎潜在的治疗靶点.BANNISTER等[19 ] 对新生儿接种卡介苗后的外周血单核细胞基因序列分析中发现了687个差异甲基化位点,包括RNA编辑酶腺苷酸脱氨酶(adenosine deaminase acting on RNA,ADAR )、干扰素诱导的跨膜蛋白1(interferon-induced transmembrane protein 1,IFITM1 )、信号转导与转录激活因子1(signal transduction and activator of transcription 1,STAT1 )、干扰素调节因子1(interferon regulatory factor 1,IRF1 )和IRF7 等,这些基因主要富集于信号通路,揭示了新生儿卡介苗疫苗接种与单核细胞的DNA甲基化特征的相关性.多项研究已经证明了DNA甲基化在肺部感染免疫中的重要调控作用,但其具体的分子机制及临床应用转化仍需进一步研究. ...
1
... 研究发现,DNA甲基化异常在肺炎免疫失衡中扮演了重要角色,可通过调控多种免疫细胞功能来影响肺炎转归.巨噬细胞是肺部感染和炎症反应中最为重要的固有免疫细胞,其分化发育与功能执行都与DNA甲基化密切相关.DEKKERS等[15 ] 的研究发现,巨噬细胞的分化和激活伴随着大量差异甲基化的基因位点和显著的表观遗传重构.γ-氨基丁酸转运蛋白2(γ-aminobutyric acid transporter 2,GAT2)可通过增强巨噬细胞基因启动子区域内的DNA甲基化来促进巨噬细胞中的重要促炎因子白细胞介素-1β(interleukin-1β,IL-1β)的产生,而GAT2缺乏会减弱巨噬细胞介导的体内炎症反应[16 ] .除巨噬细胞外,DNA甲基化还可通过调控调节性T细胞(regulatory T cell,Treg)来参与肺炎的损伤修复.SINGER等[17 ] 在研究流感病毒诱导的小鼠肺炎中发现,接受DNA甲基转移酶抑制剂治疗的小鼠肺部Treg数量增加,并且肺部炎症得到有效缓解.此外,MCGRATH-MORROW等[18 ] 的研究发现,新生小鼠CD4+ T细胞通路关键基因包括BTB与CNC同源基因2(BTB and CNC homology 2,Bach2 )、富集AT序列的特异性结合蛋白1(special AT rich sequence binding protein-1,Satb1 )、Tet1 (ten eleven translocation 1)等启动子的DNA甲基化, 抑制了新生小鼠对肺炎的免疫反应,表明DNA甲基化可能在肺CD4+ T细胞的成熟和对感染的反应中发挥了重要作用,可作为新生儿肺炎潜在的治疗靶点.BANNISTER等[19 ] 对新生儿接种卡介苗后的外周血单核细胞基因序列分析中发现了687个差异甲基化位点,包括RNA编辑酶腺苷酸脱氨酶(adenosine deaminase acting on RNA,ADAR )、干扰素诱导的跨膜蛋白1(interferon-induced transmembrane protein 1,IFITM1 )、信号转导与转录激活因子1(signal transduction and activator of transcription 1,STAT1 )、干扰素调节因子1(interferon regulatory factor 1,IRF1 )和IRF7 等,这些基因主要富集于信号通路,揭示了新生儿卡介苗疫苗接种与单核细胞的DNA甲基化特征的相关性.多项研究已经证明了DNA甲基化在肺部感染免疫中的重要调控作用,但其具体的分子机制及临床应用转化仍需进一步研究. ...
1
... 研究发现,DNA甲基化异常在肺炎免疫失衡中扮演了重要角色,可通过调控多种免疫细胞功能来影响肺炎转归.巨噬细胞是肺部感染和炎症反应中最为重要的固有免疫细胞,其分化发育与功能执行都与DNA甲基化密切相关.DEKKERS等[15 ] 的研究发现,巨噬细胞的分化和激活伴随着大量差异甲基化的基因位点和显著的表观遗传重构.γ-氨基丁酸转运蛋白2(γ-aminobutyric acid transporter 2,GAT2)可通过增强巨噬细胞基因启动子区域内的DNA甲基化来促进巨噬细胞中的重要促炎因子白细胞介素-1β(interleukin-1β,IL-1β)的产生,而GAT2缺乏会减弱巨噬细胞介导的体内炎症反应[16 ] .除巨噬细胞外,DNA甲基化还可通过调控调节性T细胞(regulatory T cell,Treg)来参与肺炎的损伤修复.SINGER等[17 ] 在研究流感病毒诱导的小鼠肺炎中发现,接受DNA甲基转移酶抑制剂治疗的小鼠肺部Treg数量增加,并且肺部炎症得到有效缓解.此外,MCGRATH-MORROW等[18 ] 的研究发现,新生小鼠CD4+ T细胞通路关键基因包括BTB与CNC同源基因2(BTB and CNC homology 2,Bach2 )、富集AT序列的特异性结合蛋白1(special AT rich sequence binding protein-1,Satb1 )、Tet1 (ten eleven translocation 1)等启动子的DNA甲基化, 抑制了新生小鼠对肺炎的免疫反应,表明DNA甲基化可能在肺CD4+ T细胞的成熟和对感染的反应中发挥了重要作用,可作为新生儿肺炎潜在的治疗靶点.BANNISTER等[19 ] 对新生儿接种卡介苗后的外周血单核细胞基因序列分析中发现了687个差异甲基化位点,包括RNA编辑酶腺苷酸脱氨酶(adenosine deaminase acting on RNA,ADAR )、干扰素诱导的跨膜蛋白1(interferon-induced transmembrane protein 1,IFITM1 )、信号转导与转录激活因子1(signal transduction and activator of transcription 1,STAT1 )、干扰素调节因子1(interferon regulatory factor 1,IRF1 )和IRF7 等,这些基因主要富集于信号通路,揭示了新生儿卡介苗疫苗接种与单核细胞的DNA甲基化特征的相关性.多项研究已经证明了DNA甲基化在肺部感染免疫中的重要调控作用,但其具体的分子机制及临床应用转化仍需进一步研究. ...
1
... 正常情况下多数CpG为去甲基化,与基因激活有关.环境改变可诱导DNA甲基化的发生,从而抑制基因表达[20 ] .已有研究表明,营养状况、饮食及空气污染等影响个体肺炎易感性的重要环境因素可通过改变DNA甲基化水平进而调控肺部感染免疫应答.AMPOMAH等[21 ] 的研究发现,人体必需氨基酸甲硫氨酸(体内重要的甲基供体)可通过增加巨噬细胞中的DNA甲基化水平来抑制脂多糖诱导的肺部炎症反应.此外,小鼠在产前暴露于烟草烟雾环境可能会通过改变肺组织细胞的DNA甲基化模式增加后代肺部炎症的发生风险,表明DNA甲基化对肺免疫功能的改变仍可能影响下一代[22 -23 ] .作为环境因素暴露所致不良生物效应的潜在调节因子,DNA甲基化与肺部感染免疫应答密切相关,为探讨环境改变与个体肺炎易感性之间的关系提供了新的思路. ...
1
... 正常情况下多数CpG为去甲基化,与基因激活有关.环境改变可诱导DNA甲基化的发生,从而抑制基因表达[20 ] .已有研究表明,营养状况、饮食及空气污染等影响个体肺炎易感性的重要环境因素可通过改变DNA甲基化水平进而调控肺部感染免疫应答.AMPOMAH等[21 ] 的研究发现,人体必需氨基酸甲硫氨酸(体内重要的甲基供体)可通过增加巨噬细胞中的DNA甲基化水平来抑制脂多糖诱导的肺部炎症反应.此外,小鼠在产前暴露于烟草烟雾环境可能会通过改变肺组织细胞的DNA甲基化模式增加后代肺部炎症的发生风险,表明DNA甲基化对肺免疫功能的改变仍可能影响下一代[22 -23 ] .作为环境因素暴露所致不良生物效应的潜在调节因子,DNA甲基化与肺部感染免疫应答密切相关,为探讨环境改变与个体肺炎易感性之间的关系提供了新的思路. ...
1
... 正常情况下多数CpG为去甲基化,与基因激活有关.环境改变可诱导DNA甲基化的发生,从而抑制基因表达[20 ] .已有研究表明,营养状况、饮食及空气污染等影响个体肺炎易感性的重要环境因素可通过改变DNA甲基化水平进而调控肺部感染免疫应答.AMPOMAH等[21 ] 的研究发现,人体必需氨基酸甲硫氨酸(体内重要的甲基供体)可通过增加巨噬细胞中的DNA甲基化水平来抑制脂多糖诱导的肺部炎症反应.此外,小鼠在产前暴露于烟草烟雾环境可能会通过改变肺组织细胞的DNA甲基化模式增加后代肺部炎症的发生风险,表明DNA甲基化对肺免疫功能的改变仍可能影响下一代[22 -23 ] .作为环境因素暴露所致不良生物效应的潜在调节因子,DNA甲基化与肺部感染免疫应答密切相关,为探讨环境改变与个体肺炎易感性之间的关系提供了新的思路. ...
1
... 正常情况下多数CpG为去甲基化,与基因激活有关.环境改变可诱导DNA甲基化的发生,从而抑制基因表达[20 ] .已有研究表明,营养状况、饮食及空气污染等影响个体肺炎易感性的重要环境因素可通过改变DNA甲基化水平进而调控肺部感染免疫应答.AMPOMAH等[21 ] 的研究发现,人体必需氨基酸甲硫氨酸(体内重要的甲基供体)可通过增加巨噬细胞中的DNA甲基化水平来抑制脂多糖诱导的肺部炎症反应.此外,小鼠在产前暴露于烟草烟雾环境可能会通过改变肺组织细胞的DNA甲基化模式增加后代肺部炎症的发生风险,表明DNA甲基化对肺免疫功能的改变仍可能影响下一代[22 -23 ] .作为环境因素暴露所致不良生物效应的潜在调节因子,DNA甲基化与肺部感染免疫应答密切相关,为探讨环境改变与个体肺炎易感性之间的关系提供了新的思路. ...
1
... 组蛋白包括H1、H2A、H2B、H3和H4,DNA包裹4种核心组蛋白(H2A、H2B、H3和H4)形成的八聚体结构是组成真核细胞染色质的基本结构单位.染色质结构是影响基因表达的重要因素,包括以固缩状态存在而避免基因转录的异染色质和以松散聚集状态存在、允许转录基因的常染色质.近来研究[24 ] 发现组蛋白修饰主要发生在H3和H4的N端,包括甲基化、乙酰化、磷酸化、泛素化、乳酸化等,可通过改变组蛋白的结构从而影响染色质状态以及相关基因的表达,是一种重要的基因表达调控的可逆性蛋白翻译后修饰. ...
1
... 组蛋白的甲基化涉及甲基与氨基酸侧链或氨基末端氮原子的连接,其中H3是组蛋白甲基化的主要部位,且精氨酸(arginine,Arg/R)和赖氨酸(lysine,Lys/K)残基是最常见的甲基化修饰位点.组蛋白赖氨酸甲基转移酶(histone lysine methyltransferase,KMT)都含有进化保守的Suvar, Enhancer of zeste, and Trithorax (SET)催化结构域,能催化S-腺苷甲硫氨酸中甲基添加到目标组蛋白赖氨酸残基的N端.根据SET结构域的结构特征,KMT共分为9个家族[zeste基因增强子(enhancer of zeste,EZ)、SET1、SET2、SET和myeloid-Nervy-DEAF-1(MYND)结构域蛋白(SET and MYND domain containing protein,SMYD)、色斑抑制因子39(suppressor of variegation 3-9,SUV39)、SUV4-20、视网膜母细胞瘤结合锌指蛋白(retinoblastoma-interacting zinc finger protein,RIZ)、SET8/PR-SET7和SET7/9],已在人类中鉴定出30种含SET结构域并具有KMT活性的蛋白质[25 ] ,且有研究[26 ] 表明组蛋白甲基化修饰与肺部感染病原体后的机体免疫平衡调控和炎症因子表达密切相关.巨噬细胞可分为M1型和M2型,其中M1型巨噬细胞可分泌多种促炎因子,具有强大的吞噬杀菌作用,但是过度的炎症反应可加重肺组织损伤.研究[26 ] 发现,组蛋白甲基化酶G9a可通过促进叉头框蛋白基因1(forkhead box gene P1,FOXP1 )基因启动子区域内的组蛋白H3第9位赖氨酸二甲基化(dimethylated histone H3 lysine 9,H3K9me2),进而促进M1型巨噬细胞活化,加重肺炎链球菌感染小鼠的肺部炎症反应;在敲低组蛋白甲基化酶G9a 后,肺炎链球菌感染小鼠肺部的炎症细胞浸润减少,肺泡灌洗液中促炎因子水平降低,肺部炎症损伤得到有效缓解.组蛋白甲基转移酶zeste基因增强子人类同源物2(enhancer of zeste homolog 2,EZH2)可特异性催化组蛋白H3第27位赖氨酸三甲基化(trimethylated histone H3 lysine 27,H3K27me3),在转录抑制中起重要作用,与肺部病原体感染以及感染后反复喘息发作密切相关.一项针对227名被诊断为重症肺炎的临床研究[27 ] 表明,喘息组患儿肺泡灌洗液中细胞因子IL-2和IL-6表达水平以及H3K27me3和EZH2表达水平显著高于非喘息组患儿,并且喘息患儿治疗后肺泡灌洗液中EZH2和H3K27me3的表达水平降低,表明EZH2介导的H2K27me3失衡可能与肺部炎症后喘息的发生有关;进一步研究发现,肺泡巨噬细胞中的H3K27me3水平及核因子κB(nuclear factor kappa B,NF-κB)信号通路激活在肺部感染后反复喘息发作中扮演了重要角色,但肺泡巨噬细胞中组蛋白甲基化水平如何调控各种炎症因子表达的具体机制仍不清楚. ...
2
... 组蛋白的甲基化涉及甲基与氨基酸侧链或氨基末端氮原子的连接,其中H3是组蛋白甲基化的主要部位,且精氨酸(arginine,Arg/R)和赖氨酸(lysine,Lys/K)残基是最常见的甲基化修饰位点.组蛋白赖氨酸甲基转移酶(histone lysine methyltransferase,KMT)都含有进化保守的Suvar, Enhancer of zeste, and Trithorax (SET)催化结构域,能催化S-腺苷甲硫氨酸中甲基添加到目标组蛋白赖氨酸残基的N端.根据SET结构域的结构特征,KMT共分为9个家族[zeste基因增强子(enhancer of zeste,EZ)、SET1、SET2、SET和myeloid-Nervy-DEAF-1(MYND)结构域蛋白(SET and MYND domain containing protein,SMYD)、色斑抑制因子39(suppressor of variegation 3-9,SUV39)、SUV4-20、视网膜母细胞瘤结合锌指蛋白(retinoblastoma-interacting zinc finger protein,RIZ)、SET8/PR-SET7和SET7/9],已在人类中鉴定出30种含SET结构域并具有KMT活性的蛋白质[25 ] ,且有研究[26 ] 表明组蛋白甲基化修饰与肺部感染病原体后的机体免疫平衡调控和炎症因子表达密切相关.巨噬细胞可分为M1型和M2型,其中M1型巨噬细胞可分泌多种促炎因子,具有强大的吞噬杀菌作用,但是过度的炎症反应可加重肺组织损伤.研究[26 ] 发现,组蛋白甲基化酶G9a可通过促进叉头框蛋白基因1(forkhead box gene P1,FOXP1 )基因启动子区域内的组蛋白H3第9位赖氨酸二甲基化(dimethylated histone H3 lysine 9,H3K9me2),进而促进M1型巨噬细胞活化,加重肺炎链球菌感染小鼠的肺部炎症反应;在敲低组蛋白甲基化酶G9a 后,肺炎链球菌感染小鼠肺部的炎症细胞浸润减少,肺泡灌洗液中促炎因子水平降低,肺部炎症损伤得到有效缓解.组蛋白甲基转移酶zeste基因增强子人类同源物2(enhancer of zeste homolog 2,EZH2)可特异性催化组蛋白H3第27位赖氨酸三甲基化(trimethylated histone H3 lysine 27,H3K27me3),在转录抑制中起重要作用,与肺部病原体感染以及感染后反复喘息发作密切相关.一项针对227名被诊断为重症肺炎的临床研究[27 ] 表明,喘息组患儿肺泡灌洗液中细胞因子IL-2和IL-6表达水平以及H3K27me3和EZH2表达水平显著高于非喘息组患儿,并且喘息患儿治疗后肺泡灌洗液中EZH2和H3K27me3的表达水平降低,表明EZH2介导的H2K27me3失衡可能与肺部炎症后喘息的发生有关;进一步研究发现,肺泡巨噬细胞中的H3K27me3水平及核因子κB(nuclear factor kappa B,NF-κB)信号通路激活在肺部感染后反复喘息发作中扮演了重要角色,但肺泡巨噬细胞中组蛋白甲基化水平如何调控各种炎症因子表达的具体机制仍不清楚. ...
... [26 ]发现,组蛋白甲基化酶G9a可通过促进叉头框蛋白基因1(forkhead box gene P1,FOXP1 )基因启动子区域内的组蛋白H3第9位赖氨酸二甲基化(dimethylated histone H3 lysine 9,H3K9me2),进而促进M1型巨噬细胞活化,加重肺炎链球菌感染小鼠的肺部炎症反应;在敲低组蛋白甲基化酶G9a 后,肺炎链球菌感染小鼠肺部的炎症细胞浸润减少,肺泡灌洗液中促炎因子水平降低,肺部炎症损伤得到有效缓解.组蛋白甲基转移酶zeste基因增强子人类同源物2(enhancer of zeste homolog 2,EZH2)可特异性催化组蛋白H3第27位赖氨酸三甲基化(trimethylated histone H3 lysine 27,H3K27me3),在转录抑制中起重要作用,与肺部病原体感染以及感染后反复喘息发作密切相关.一项针对227名被诊断为重症肺炎的临床研究[27 ] 表明,喘息组患儿肺泡灌洗液中细胞因子IL-2和IL-6表达水平以及H3K27me3和EZH2表达水平显著高于非喘息组患儿,并且喘息患儿治疗后肺泡灌洗液中EZH2和H3K27me3的表达水平降低,表明EZH2介导的H2K27me3失衡可能与肺部炎症后喘息的发生有关;进一步研究发现,肺泡巨噬细胞中的H3K27me3水平及核因子κB(nuclear factor kappa B,NF-κB)信号通路激活在肺部感染后反复喘息发作中扮演了重要角色,但肺泡巨噬细胞中组蛋白甲基化水平如何调控各种炎症因子表达的具体机制仍不清楚. ...
1
... 组蛋白的甲基化涉及甲基与氨基酸侧链或氨基末端氮原子的连接,其中H3是组蛋白甲基化的主要部位,且精氨酸(arginine,Arg/R)和赖氨酸(lysine,Lys/K)残基是最常见的甲基化修饰位点.组蛋白赖氨酸甲基转移酶(histone lysine methyltransferase,KMT)都含有进化保守的Suvar, Enhancer of zeste, and Trithorax (SET)催化结构域,能催化S-腺苷甲硫氨酸中甲基添加到目标组蛋白赖氨酸残基的N端.根据SET结构域的结构特征,KMT共分为9个家族[zeste基因增强子(enhancer of zeste,EZ)、SET1、SET2、SET和myeloid-Nervy-DEAF-1(MYND)结构域蛋白(SET and MYND domain containing protein,SMYD)、色斑抑制因子39(suppressor of variegation 3-9,SUV39)、SUV4-20、视网膜母细胞瘤结合锌指蛋白(retinoblastoma-interacting zinc finger protein,RIZ)、SET8/PR-SET7和SET7/9],已在人类中鉴定出30种含SET结构域并具有KMT活性的蛋白质[25 ] ,且有研究[26 ] 表明组蛋白甲基化修饰与肺部感染病原体后的机体免疫平衡调控和炎症因子表达密切相关.巨噬细胞可分为M1型和M2型,其中M1型巨噬细胞可分泌多种促炎因子,具有强大的吞噬杀菌作用,但是过度的炎症反应可加重肺组织损伤.研究[26 ] 发现,组蛋白甲基化酶G9a可通过促进叉头框蛋白基因1(forkhead box gene P1,FOXP1 )基因启动子区域内的组蛋白H3第9位赖氨酸二甲基化(dimethylated histone H3 lysine 9,H3K9me2),进而促进M1型巨噬细胞活化,加重肺炎链球菌感染小鼠的肺部炎症反应;在敲低组蛋白甲基化酶G9a 后,肺炎链球菌感染小鼠肺部的炎症细胞浸润减少,肺泡灌洗液中促炎因子水平降低,肺部炎症损伤得到有效缓解.组蛋白甲基转移酶zeste基因增强子人类同源物2(enhancer of zeste homolog 2,EZH2)可特异性催化组蛋白H3第27位赖氨酸三甲基化(trimethylated histone H3 lysine 27,H3K27me3),在转录抑制中起重要作用,与肺部病原体感染以及感染后反复喘息发作密切相关.一项针对227名被诊断为重症肺炎的临床研究[27 ] 表明,喘息组患儿肺泡灌洗液中细胞因子IL-2和IL-6表达水平以及H3K27me3和EZH2表达水平显著高于非喘息组患儿,并且喘息患儿治疗后肺泡灌洗液中EZH2和H3K27me3的表达水平降低,表明EZH2介导的H2K27me3失衡可能与肺部炎症后喘息的发生有关;进一步研究发现,肺泡巨噬细胞中的H3K27me3水平及核因子κB(nuclear factor kappa B,NF-κB)信号通路激活在肺部感染后反复喘息发作中扮演了重要角色,但肺泡巨噬细胞中组蛋白甲基化水平如何调控各种炎症因子表达的具体机制仍不清楚. ...
1
... 赖氨酸残基的组蛋白乙酰化也是研究最广泛的修饰之一,由组蛋白乙酰转移酶(histone acetyltransferase,HAT)和组蛋白去乙酰化酶(histone deacetylase,HDAC)进行调控.HAT以乙酰辅酶A为供体,催化乙酰基添加到组蛋白赖氨酸残基的N端从而中和残基上的正电荷,破坏组蛋白与DNA的相互连接,进而展开DNA组蛋白构象,允许转录因子与DNA相互作用来促进基因表达;反之,HDAC从组蛋白的N端除去乙酰基,导致染色质结构更加紧密,促进基因沉默[28 ] .组蛋白乙酰化的动态调控通过影响多种免疫细胞的分化发育和炎症通路的激活从而参与肺炎的发生发展过程.例如,HDAC可通过调节单核细胞的增殖、成熟和分化等影响肺部的炎症反应、细菌感染或病毒感染等过程[29 -30 ] .HDAC3属于Ⅰ类HDAC家族,是巨噬细胞炎症相关基因表达所必需的酶.YAO等[31 ] 对Hdac3 敲除小鼠的肺巨噬细胞进行RNA测序时发现,与机体炎症和免疫反应信号通路相关的基因表达下调,例如FMS-样酪氨酸激酶1(FMS-like tyrosine kinase 1,Flt1 )、丝裂原活化蛋白激酶3(mitogen-activated protein kinase 3,Mapk3 )、丝裂原活化蛋白激酶的激酶的激酶1(mitogen-activated protein kinase kinase kinase 1,Map3k1 )和Toll样受体2(Toll-like receptor 2,Tlr2 ),并且肺泡巨噬细胞发育所必需的过氧化物酶体增殖物激活受体-γ(peroxisome proliferator activated receptor-γ,Ppar-γ )基因亦下调,揭示了HDAC3在调控肺泡巨噬细胞发育和肺部炎症过程中所发挥的关键作用. ...
1
... 赖氨酸残基的组蛋白乙酰化也是研究最广泛的修饰之一,由组蛋白乙酰转移酶(histone acetyltransferase,HAT)和组蛋白去乙酰化酶(histone deacetylase,HDAC)进行调控.HAT以乙酰辅酶A为供体,催化乙酰基添加到组蛋白赖氨酸残基的N端从而中和残基上的正电荷,破坏组蛋白与DNA的相互连接,进而展开DNA组蛋白构象,允许转录因子与DNA相互作用来促进基因表达;反之,HDAC从组蛋白的N端除去乙酰基,导致染色质结构更加紧密,促进基因沉默[28 ] .组蛋白乙酰化的动态调控通过影响多种免疫细胞的分化发育和炎症通路的激活从而参与肺炎的发生发展过程.例如,HDAC可通过调节单核细胞的增殖、成熟和分化等影响肺部的炎症反应、细菌感染或病毒感染等过程[29 -30 ] .HDAC3属于Ⅰ类HDAC家族,是巨噬细胞炎症相关基因表达所必需的酶.YAO等[31 ] 对Hdac3 敲除小鼠的肺巨噬细胞进行RNA测序时发现,与机体炎症和免疫反应信号通路相关的基因表达下调,例如FMS-样酪氨酸激酶1(FMS-like tyrosine kinase 1,Flt1 )、丝裂原活化蛋白激酶3(mitogen-activated protein kinase 3,Mapk3 )、丝裂原活化蛋白激酶的激酶的激酶1(mitogen-activated protein kinase kinase kinase 1,Map3k1 )和Toll样受体2(Toll-like receptor 2,Tlr2 ),并且肺泡巨噬细胞发育所必需的过氧化物酶体增殖物激活受体-γ(peroxisome proliferator activated receptor-γ,Ppar-γ )基因亦下调,揭示了HDAC3在调控肺泡巨噬细胞发育和肺部炎症过程中所发挥的关键作用. ...
1
... 赖氨酸残基的组蛋白乙酰化也是研究最广泛的修饰之一,由组蛋白乙酰转移酶(histone acetyltransferase,HAT)和组蛋白去乙酰化酶(histone deacetylase,HDAC)进行调控.HAT以乙酰辅酶A为供体,催化乙酰基添加到组蛋白赖氨酸残基的N端从而中和残基上的正电荷,破坏组蛋白与DNA的相互连接,进而展开DNA组蛋白构象,允许转录因子与DNA相互作用来促进基因表达;反之,HDAC从组蛋白的N端除去乙酰基,导致染色质结构更加紧密,促进基因沉默[28 ] .组蛋白乙酰化的动态调控通过影响多种免疫细胞的分化发育和炎症通路的激活从而参与肺炎的发生发展过程.例如,HDAC可通过调节单核细胞的增殖、成熟和分化等影响肺部的炎症反应、细菌感染或病毒感染等过程[29 -30 ] .HDAC3属于Ⅰ类HDAC家族,是巨噬细胞炎症相关基因表达所必需的酶.YAO等[31 ] 对Hdac3 敲除小鼠的肺巨噬细胞进行RNA测序时发现,与机体炎症和免疫反应信号通路相关的基因表达下调,例如FMS-样酪氨酸激酶1(FMS-like tyrosine kinase 1,Flt1 )、丝裂原活化蛋白激酶3(mitogen-activated protein kinase 3,Mapk3 )、丝裂原活化蛋白激酶的激酶的激酶1(mitogen-activated protein kinase kinase kinase 1,Map3k1 )和Toll样受体2(Toll-like receptor 2,Tlr2 ),并且肺泡巨噬细胞发育所必需的过氧化物酶体增殖物激活受体-γ(peroxisome proliferator activated receptor-γ,Ppar-γ )基因亦下调,揭示了HDAC3在调控肺泡巨噬细胞发育和肺部炎症过程中所发挥的关键作用. ...
1
... 赖氨酸残基的组蛋白乙酰化也是研究最广泛的修饰之一,由组蛋白乙酰转移酶(histone acetyltransferase,HAT)和组蛋白去乙酰化酶(histone deacetylase,HDAC)进行调控.HAT以乙酰辅酶A为供体,催化乙酰基添加到组蛋白赖氨酸残基的N端从而中和残基上的正电荷,破坏组蛋白与DNA的相互连接,进而展开DNA组蛋白构象,允许转录因子与DNA相互作用来促进基因表达;反之,HDAC从组蛋白的N端除去乙酰基,导致染色质结构更加紧密,促进基因沉默[28 ] .组蛋白乙酰化的动态调控通过影响多种免疫细胞的分化发育和炎症通路的激活从而参与肺炎的发生发展过程.例如,HDAC可通过调节单核细胞的增殖、成熟和分化等影响肺部的炎症反应、细菌感染或病毒感染等过程[29 -30 ] .HDAC3属于Ⅰ类HDAC家族,是巨噬细胞炎症相关基因表达所必需的酶.YAO等[31 ] 对Hdac3 敲除小鼠的肺巨噬细胞进行RNA测序时发现,与机体炎症和免疫反应信号通路相关的基因表达下调,例如FMS-样酪氨酸激酶1(FMS-like tyrosine kinase 1,Flt1 )、丝裂原活化蛋白激酶3(mitogen-activated protein kinase 3,Mapk3 )、丝裂原活化蛋白激酶的激酶的激酶1(mitogen-activated protein kinase kinase kinase 1,Map3k1 )和Toll样受体2(Toll-like receptor 2,Tlr2 ),并且肺泡巨噬细胞发育所必需的过氧化物酶体增殖物激活受体-γ(peroxisome proliferator activated receptor-γ,Ppar-γ )基因亦下调,揭示了HDAC3在调控肺泡巨噬细胞发育和肺部炎症过程中所发挥的关键作用. ...
1
... 值得注意的是,近来有研究发现一些组蛋白修饰酶的抑制剂能够减轻肺损伤和炎症反应,可作为潜在的治疗靶点.呼吸道合胞病毒(respiratory syncytial virus,RSV)是婴儿和幼儿下呼吸道感染的主要病原体之一.HDAC抑制剂,如曲古抑菌素A(trichostatin A,TSA)和伏立诺他(suberoylanilide hydroxamic acid,SAHA)可上调干扰素α相关的信号通路,如NF-κB、中环氧化酶-2(cyclooxygenase-2,COX-2)、MAPK和STAT3,显著抑制RSV的复制,同时减少RSV诱导的促炎因子和氧化应激相关分子的产生[32 ] .姜黄素属于HDAC抑制剂,是从姜黄中提取的天然多酚,具有广泛的抗炎特性.在流感病毒感染后,姜黄素治疗可有效抑制人巨噬细胞中促炎因子的分泌和NF-κB 基因的表达,从而减轻甲型流感病毒诱导的急性肺损伤[33 ] .HDAC6是位于细胞质中的Ⅱ类HDAC家族成员,其特异性的抑制剂CAY10603可抑制NF-κB信号通路的激活和炎症小体活化,对脂多糖诱导的肺部炎症具有保护作用[34 ] .组蛋白修饰作为一种重要的调控基因表达的可逆性蛋白翻译后修饰,是临床诊疗的重要靶点.目前多项动物研究已经证实了组蛋白修饰酶抑制剂对肺炎的保护作用,未来可以开展相应的研究进一步探索其临床应用价值. ...
1
... 值得注意的是,近来有研究发现一些组蛋白修饰酶的抑制剂能够减轻肺损伤和炎症反应,可作为潜在的治疗靶点.呼吸道合胞病毒(respiratory syncytial virus,RSV)是婴儿和幼儿下呼吸道感染的主要病原体之一.HDAC抑制剂,如曲古抑菌素A(trichostatin A,TSA)和伏立诺他(suberoylanilide hydroxamic acid,SAHA)可上调干扰素α相关的信号通路,如NF-κB、中环氧化酶-2(cyclooxygenase-2,COX-2)、MAPK和STAT3,显著抑制RSV的复制,同时减少RSV诱导的促炎因子和氧化应激相关分子的产生[32 ] .姜黄素属于HDAC抑制剂,是从姜黄中提取的天然多酚,具有广泛的抗炎特性.在流感病毒感染后,姜黄素治疗可有效抑制人巨噬细胞中促炎因子的分泌和NF-κB 基因的表达,从而减轻甲型流感病毒诱导的急性肺损伤[33 ] .HDAC6是位于细胞质中的Ⅱ类HDAC家族成员,其特异性的抑制剂CAY10603可抑制NF-κB信号通路的激活和炎症小体活化,对脂多糖诱导的肺部炎症具有保护作用[34 ] .组蛋白修饰作为一种重要的调控基因表达的可逆性蛋白翻译后修饰,是临床诊疗的重要靶点.目前多项动物研究已经证实了组蛋白修饰酶抑制剂对肺炎的保护作用,未来可以开展相应的研究进一步探索其临床应用价值. ...
1
... 值得注意的是,近来有研究发现一些组蛋白修饰酶的抑制剂能够减轻肺损伤和炎症反应,可作为潜在的治疗靶点.呼吸道合胞病毒(respiratory syncytial virus,RSV)是婴儿和幼儿下呼吸道感染的主要病原体之一.HDAC抑制剂,如曲古抑菌素A(trichostatin A,TSA)和伏立诺他(suberoylanilide hydroxamic acid,SAHA)可上调干扰素α相关的信号通路,如NF-κB、中环氧化酶-2(cyclooxygenase-2,COX-2)、MAPK和STAT3,显著抑制RSV的复制,同时减少RSV诱导的促炎因子和氧化应激相关分子的产生[32 ] .姜黄素属于HDAC抑制剂,是从姜黄中提取的天然多酚,具有广泛的抗炎特性.在流感病毒感染后,姜黄素治疗可有效抑制人巨噬细胞中促炎因子的分泌和NF-κB 基因的表达,从而减轻甲型流感病毒诱导的急性肺损伤[33 ] .HDAC6是位于细胞质中的Ⅱ类HDAC家族成员,其特异性的抑制剂CAY10603可抑制NF-κB信号通路的激活和炎症小体活化,对脂多糖诱导的肺部炎症具有保护作用[34 ] .组蛋白修饰作为一种重要的调控基因表达的可逆性蛋白翻译后修饰,是临床诊疗的重要靶点.目前多项动物研究已经证实了组蛋白修饰酶抑制剂对肺炎的保护作用,未来可以开展相应的研究进一步探索其临床应用价值. ...
1
... miRNA是大小为19~25个核苷酸的内源性RNA,能够通过抑制信使RNA(messger RNA,mRNA)翻译或促进mRNA降解来调节转录后水平的基因表达[35 ] .大量研究[36 -38 ] 发现miRNA参与肺部感染免疫和炎症反应,并有学者[39 ] 提出miRNA谱可作为预测肺炎住院患者病情严重程度的指标,具有重要的肺炎临床诊断价值.此外,GARCÍA-HIDALGO等[40 ] 在评估新型冠状病毒(SARS-CoV-2)感染患者出院3个月后肺损伤程度与外周血的miRNA谱的关系时发现,miR-9-5p、miR-16-5p和miR-221-3p的水平与肺损伤的严重程度呈负相关;进一步进行京都基因与基因组百科全书数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)分析和基因本体(Gene Ontology,GO)富集分析发现,miRNA在多个炎症和免疫反应的信号分子通路[包括肿瘤坏死因子(tumor necrosis factor,TNF)信号通路,TLR信号通路,Ⅰ型干扰素(type Ⅰ interferon,IFN-Ⅰ)信号通路]中富集,为肺部后遗症发生的分子机制研究提供了新的方向. ...
1
... miRNA是大小为19~25个核苷酸的内源性RNA,能够通过抑制信使RNA(messger RNA,mRNA)翻译或促进mRNA降解来调节转录后水平的基因表达[35 ] .大量研究[36 -38 ] 发现miRNA参与肺部感染免疫和炎症反应,并有学者[39 ] 提出miRNA谱可作为预测肺炎住院患者病情严重程度的指标,具有重要的肺炎临床诊断价值.此外,GARCÍA-HIDALGO等[40 ] 在评估新型冠状病毒(SARS-CoV-2)感染患者出院3个月后肺损伤程度与外周血的miRNA谱的关系时发现,miR-9-5p、miR-16-5p和miR-221-3p的水平与肺损伤的严重程度呈负相关;进一步进行京都基因与基因组百科全书数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)分析和基因本体(Gene Ontology,GO)富集分析发现,miRNA在多个炎症和免疫反应的信号分子通路[包括肿瘤坏死因子(tumor necrosis factor,TNF)信号通路,TLR信号通路,Ⅰ型干扰素(type Ⅰ interferon,IFN-Ⅰ)信号通路]中富集,为肺部后遗症发生的分子机制研究提供了新的方向. ...
1
... miRNA是大小为19~25个核苷酸的内源性RNA,能够通过抑制信使RNA(messger RNA,mRNA)翻译或促进mRNA降解来调节转录后水平的基因表达[35 ] .大量研究[36 -38 ] 发现miRNA参与肺部感染免疫和炎症反应,并有学者[39 ] 提出miRNA谱可作为预测肺炎住院患者病情严重程度的指标,具有重要的肺炎临床诊断价值.此外,GARCÍA-HIDALGO等[40 ] 在评估新型冠状病毒(SARS-CoV-2)感染患者出院3个月后肺损伤程度与外周血的miRNA谱的关系时发现,miR-9-5p、miR-16-5p和miR-221-3p的水平与肺损伤的严重程度呈负相关;进一步进行京都基因与基因组百科全书数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)分析和基因本体(Gene Ontology,GO)富集分析发现,miRNA在多个炎症和免疫反应的信号分子通路[包括肿瘤坏死因子(tumor necrosis factor,TNF)信号通路,TLR信号通路,Ⅰ型干扰素(type Ⅰ interferon,IFN-Ⅰ)信号通路]中富集,为肺部后遗症发生的分子机制研究提供了新的方向. ...
1
... miRNA是大小为19~25个核苷酸的内源性RNA,能够通过抑制信使RNA(messger RNA,mRNA)翻译或促进mRNA降解来调节转录后水平的基因表达[35 ] .大量研究[36 -38 ] 发现miRNA参与肺部感染免疫和炎症反应,并有学者[39 ] 提出miRNA谱可作为预测肺炎住院患者病情严重程度的指标,具有重要的肺炎临床诊断价值.此外,GARCÍA-HIDALGO等[40 ] 在评估新型冠状病毒(SARS-CoV-2)感染患者出院3个月后肺损伤程度与外周血的miRNA谱的关系时发现,miR-9-5p、miR-16-5p和miR-221-3p的水平与肺损伤的严重程度呈负相关;进一步进行京都基因与基因组百科全书数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)分析和基因本体(Gene Ontology,GO)富集分析发现,miRNA在多个炎症和免疫反应的信号分子通路[包括肿瘤坏死因子(tumor necrosis factor,TNF)信号通路,TLR信号通路,Ⅰ型干扰素(type Ⅰ interferon,IFN-Ⅰ)信号通路]中富集,为肺部后遗症发生的分子机制研究提供了新的方向. ...
1
... miRNA是大小为19~25个核苷酸的内源性RNA,能够通过抑制信使RNA(messger RNA,mRNA)翻译或促进mRNA降解来调节转录后水平的基因表达[35 ] .大量研究[36 -38 ] 发现miRNA参与肺部感染免疫和炎症反应,并有学者[39 ] 提出miRNA谱可作为预测肺炎住院患者病情严重程度的指标,具有重要的肺炎临床诊断价值.此外,GARCÍA-HIDALGO等[40 ] 在评估新型冠状病毒(SARS-CoV-2)感染患者出院3个月后肺损伤程度与外周血的miRNA谱的关系时发现,miR-9-5p、miR-16-5p和miR-221-3p的水平与肺损伤的严重程度呈负相关;进一步进行京都基因与基因组百科全书数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)分析和基因本体(Gene Ontology,GO)富集分析发现,miRNA在多个炎症和免疫反应的信号分子通路[包括肿瘤坏死因子(tumor necrosis factor,TNF)信号通路,TLR信号通路,Ⅰ型干扰素(type Ⅰ interferon,IFN-Ⅰ)信号通路]中富集,为肺部后遗症发生的分子机制研究提供了新的方向. ...
1
... lncRNA是长度大于200个核苷酸的非编码RNA,通过与DNA、mRNA和蛋白质等相互作用,从组蛋白修饰、DNA甲基化及转录因子等多个层面调控基因表达[41 -42 ] .研究表明,lncRNA可以调节趋化因子、炎症相关细胞因子和其他基因的表达进而调控肺部感染免疫应答.例如,lnc-Cxcl2通过抑制病毒感染的上皮细胞中CXC趋化因子配体2(CXC chemokine ligand 2,CXCL2 )的表达从而减轻中性粒细胞介导的肺部炎症[43 ] .lncRNA生长阻滞特异性转录本5(growth arrest-specific transcript 5,GAS5 )可通过海绵吸附的方式影响miR-455-5p的功能,以提高儿童肺炎中的细胞因子信号转导抑制因子3(suppressor of cytokine signaling 3,SOCS3 )的表达,激活诱导型一氧化氮合酶产生NO,从而促进M1型巨噬细胞活化,加重肺部炎症反应[44 ] .lncRNA肺腺癌转移相关转录本1(metastasis-associated lung adenocarcinoma transcript 1,Malat-1 )通过促进NF-κB的表达和IL-8及肿瘤坏死因子α(tumor necrosis factor-α,TNF-α)的产生参与调节肺炎支原体感染引起的气道和肺部炎症,Malat-1 敲低可部分逆转肺炎支原体感染小鼠的肺部炎症损伤程度、血管通透性及炎症因子的分泌[45 ] . ...
1
... lncRNA是长度大于200个核苷酸的非编码RNA,通过与DNA、mRNA和蛋白质等相互作用,从组蛋白修饰、DNA甲基化及转录因子等多个层面调控基因表达[41 -42 ] .研究表明,lncRNA可以调节趋化因子、炎症相关细胞因子和其他基因的表达进而调控肺部感染免疫应答.例如,lnc-Cxcl2通过抑制病毒感染的上皮细胞中CXC趋化因子配体2(CXC chemokine ligand 2,CXCL2 )的表达从而减轻中性粒细胞介导的肺部炎症[43 ] .lncRNA生长阻滞特异性转录本5(growth arrest-specific transcript 5,GAS5 )可通过海绵吸附的方式影响miR-455-5p的功能,以提高儿童肺炎中的细胞因子信号转导抑制因子3(suppressor of cytokine signaling 3,SOCS3 )的表达,激活诱导型一氧化氮合酶产生NO,从而促进M1型巨噬细胞活化,加重肺部炎症反应[44 ] .lncRNA肺腺癌转移相关转录本1(metastasis-associated lung adenocarcinoma transcript 1,Malat-1 )通过促进NF-κB的表达和IL-8及肿瘤坏死因子α(tumor necrosis factor-α,TNF-α)的产生参与调节肺炎支原体感染引起的气道和肺部炎症,Malat-1 敲低可部分逆转肺炎支原体感染小鼠的肺部炎症损伤程度、血管通透性及炎症因子的分泌[45 ] . ...
1
... lncRNA是长度大于200个核苷酸的非编码RNA,通过与DNA、mRNA和蛋白质等相互作用,从组蛋白修饰、DNA甲基化及转录因子等多个层面调控基因表达[41 -42 ] .研究表明,lncRNA可以调节趋化因子、炎症相关细胞因子和其他基因的表达进而调控肺部感染免疫应答.例如,lnc-Cxcl2通过抑制病毒感染的上皮细胞中CXC趋化因子配体2(CXC chemokine ligand 2,CXCL2 )的表达从而减轻中性粒细胞介导的肺部炎症[43 ] .lncRNA生长阻滞特异性转录本5(growth arrest-specific transcript 5,GAS5 )可通过海绵吸附的方式影响miR-455-5p的功能,以提高儿童肺炎中的细胞因子信号转导抑制因子3(suppressor of cytokine signaling 3,SOCS3 )的表达,激活诱导型一氧化氮合酶产生NO,从而促进M1型巨噬细胞活化,加重肺部炎症反应[44 ] .lncRNA肺腺癌转移相关转录本1(metastasis-associated lung adenocarcinoma transcript 1,Malat-1 )通过促进NF-κB的表达和IL-8及肿瘤坏死因子α(tumor necrosis factor-α,TNF-α)的产生参与调节肺炎支原体感染引起的气道和肺部炎症,Malat-1 敲低可部分逆转肺炎支原体感染小鼠的肺部炎症损伤程度、血管通透性及炎症因子的分泌[45 ] . ...
1
... lncRNA是长度大于200个核苷酸的非编码RNA,通过与DNA、mRNA和蛋白质等相互作用,从组蛋白修饰、DNA甲基化及转录因子等多个层面调控基因表达[41 -42 ] .研究表明,lncRNA可以调节趋化因子、炎症相关细胞因子和其他基因的表达进而调控肺部感染免疫应答.例如,lnc-Cxcl2通过抑制病毒感染的上皮细胞中CXC趋化因子配体2(CXC chemokine ligand 2,CXCL2 )的表达从而减轻中性粒细胞介导的肺部炎症[43 ] .lncRNA生长阻滞特异性转录本5(growth arrest-specific transcript 5,GAS5 )可通过海绵吸附的方式影响miR-455-5p的功能,以提高儿童肺炎中的细胞因子信号转导抑制因子3(suppressor of cytokine signaling 3,SOCS3 )的表达,激活诱导型一氧化氮合酶产生NO,从而促进M1型巨噬细胞活化,加重肺部炎症反应[44 ] .lncRNA肺腺癌转移相关转录本1(metastasis-associated lung adenocarcinoma transcript 1,Malat-1 )通过促进NF-κB的表达和IL-8及肿瘤坏死因子α(tumor necrosis factor-α,TNF-α)的产生参与调节肺炎支原体感染引起的气道和肺部炎症,Malat-1 敲低可部分逆转肺炎支原体感染小鼠的肺部炎症损伤程度、血管通透性及炎症因子的分泌[45 ] . ...
1
... lncRNA是长度大于200个核苷酸的非编码RNA,通过与DNA、mRNA和蛋白质等相互作用,从组蛋白修饰、DNA甲基化及转录因子等多个层面调控基因表达[41 -42 ] .研究表明,lncRNA可以调节趋化因子、炎症相关细胞因子和其他基因的表达进而调控肺部感染免疫应答.例如,lnc-Cxcl2通过抑制病毒感染的上皮细胞中CXC趋化因子配体2(CXC chemokine ligand 2,CXCL2 )的表达从而减轻中性粒细胞介导的肺部炎症[43 ] .lncRNA生长阻滞特异性转录本5(growth arrest-specific transcript 5,GAS5 )可通过海绵吸附的方式影响miR-455-5p的功能,以提高儿童肺炎中的细胞因子信号转导抑制因子3(suppressor of cytokine signaling 3,SOCS3 )的表达,激活诱导型一氧化氮合酶产生NO,从而促进M1型巨噬细胞活化,加重肺部炎症反应[44 ] .lncRNA肺腺癌转移相关转录本1(metastasis-associated lung adenocarcinoma transcript 1,Malat-1 )通过促进NF-κB的表达和IL-8及肿瘤坏死因子α(tumor necrosis factor-α,TNF-α)的产生参与调节肺炎支原体感染引起的气道和肺部炎症,Malat-1 敲低可部分逆转肺炎支原体感染小鼠的肺部炎症损伤程度、血管通透性及炎症因子的分泌[45 ] . ...
1
... circRNA是由3′和5′末端在反向剪接后共价结合而形成一类特殊的非编码RNA.与线性RNA不同,circRNA为更稳定的封闭环状结构,不受RNA外切酶影响[46 ] .由于其对RNA外切酶活性的内在抗性,circRNA可作为肺炎等疾病诊断和预后的生物标志物[47 -48 ] .此外,circRNA富含miRNA结合位点,可通过充当miRNA海绵,抑制miRNA介导的mRNA的降解,在肺部炎症损伤中发挥重要作用,是肺炎治疗的潜在靶点[49 -50 ] .在病毒感染的细胞中,IRF3和环磷酸腺苷应答元件结合蛋白[cAMP-response element binding protein(CREB)binding protein,CREBBP]形成病毒激活因子的复合物,可直接促进IFN-β的转录,从而抑制病毒复制.有研究[51 ] 在对病毒感染的肺上皮细胞进行转录组测序时发现,病毒感染细胞的circRNA AIVR (antagonizes influenza virus replication)表达上调,上调的circRNA AIVR 可吸附miR-330-3p,导致CREBBP表达增加,从而促进IFN-β的产生来抑制流感病毒复制.此外,还有研究发现circRNA是影响病毒与宿主相互作用的重要分子,宿主细胞感染病毒后,病毒基因组合成相关circRNA,从而调控机体免疫应答.circ_3205是SARS-CoV-2在宿主细胞感染时合成的circRNA,可通过海绵吸附miR-298并上调其下游靶基因蛋白激酶Cε(protein kinase C epsilon,PKCε )mRNA的表达,参与SARS-CoV-2的感染免疫[52 ] .该项研究通过对病毒感染后细胞的circRNA进行序列分析,发现了病毒来源的circRNA,为探明病毒性肺炎发病机制提供了新方向. ...
1
... circRNA是由3′和5′末端在反向剪接后共价结合而形成一类特殊的非编码RNA.与线性RNA不同,circRNA为更稳定的封闭环状结构,不受RNA外切酶影响[46 ] .由于其对RNA外切酶活性的内在抗性,circRNA可作为肺炎等疾病诊断和预后的生物标志物[47 -48 ] .此外,circRNA富含miRNA结合位点,可通过充当miRNA海绵,抑制miRNA介导的mRNA的降解,在肺部炎症损伤中发挥重要作用,是肺炎治疗的潜在靶点[49 -50 ] .在病毒感染的细胞中,IRF3和环磷酸腺苷应答元件结合蛋白[cAMP-response element binding protein(CREB)binding protein,CREBBP]形成病毒激活因子的复合物,可直接促进IFN-β的转录,从而抑制病毒复制.有研究[51 ] 在对病毒感染的肺上皮细胞进行转录组测序时发现,病毒感染细胞的circRNA AIVR (antagonizes influenza virus replication)表达上调,上调的circRNA AIVR 可吸附miR-330-3p,导致CREBBP表达增加,从而促进IFN-β的产生来抑制流感病毒复制.此外,还有研究发现circRNA是影响病毒与宿主相互作用的重要分子,宿主细胞感染病毒后,病毒基因组合成相关circRNA,从而调控机体免疫应答.circ_3205是SARS-CoV-2在宿主细胞感染时合成的circRNA,可通过海绵吸附miR-298并上调其下游靶基因蛋白激酶Cε(protein kinase C epsilon,PKCε )mRNA的表达,参与SARS-CoV-2的感染免疫[52 ] .该项研究通过对病毒感染后细胞的circRNA进行序列分析,发现了病毒来源的circRNA,为探明病毒性肺炎发病机制提供了新方向. ...
1
... circRNA是由3′和5′末端在反向剪接后共价结合而形成一类特殊的非编码RNA.与线性RNA不同,circRNA为更稳定的封闭环状结构,不受RNA外切酶影响[46 ] .由于其对RNA外切酶活性的内在抗性,circRNA可作为肺炎等疾病诊断和预后的生物标志物[47 -48 ] .此外,circRNA富含miRNA结合位点,可通过充当miRNA海绵,抑制miRNA介导的mRNA的降解,在肺部炎症损伤中发挥重要作用,是肺炎治疗的潜在靶点[49 -50 ] .在病毒感染的细胞中,IRF3和环磷酸腺苷应答元件结合蛋白[cAMP-response element binding protein(CREB)binding protein,CREBBP]形成病毒激活因子的复合物,可直接促进IFN-β的转录,从而抑制病毒复制.有研究[51 ] 在对病毒感染的肺上皮细胞进行转录组测序时发现,病毒感染细胞的circRNA AIVR (antagonizes influenza virus replication)表达上调,上调的circRNA AIVR 可吸附miR-330-3p,导致CREBBP表达增加,从而促进IFN-β的产生来抑制流感病毒复制.此外,还有研究发现circRNA是影响病毒与宿主相互作用的重要分子,宿主细胞感染病毒后,病毒基因组合成相关circRNA,从而调控机体免疫应答.circ_3205是SARS-CoV-2在宿主细胞感染时合成的circRNA,可通过海绵吸附miR-298并上调其下游靶基因蛋白激酶Cε(protein kinase C epsilon,PKCε )mRNA的表达,参与SARS-CoV-2的感染免疫[52 ] .该项研究通过对病毒感染后细胞的circRNA进行序列分析,发现了病毒来源的circRNA,为探明病毒性肺炎发病机制提供了新方向. ...
1
... circRNA是由3′和5′末端在反向剪接后共价结合而形成一类特殊的非编码RNA.与线性RNA不同,circRNA为更稳定的封闭环状结构,不受RNA外切酶影响[46 ] .由于其对RNA外切酶活性的内在抗性,circRNA可作为肺炎等疾病诊断和预后的生物标志物[47 -48 ] .此外,circRNA富含miRNA结合位点,可通过充当miRNA海绵,抑制miRNA介导的mRNA的降解,在肺部炎症损伤中发挥重要作用,是肺炎治疗的潜在靶点[49 -50 ] .在病毒感染的细胞中,IRF3和环磷酸腺苷应答元件结合蛋白[cAMP-response element binding protein(CREB)binding protein,CREBBP]形成病毒激活因子的复合物,可直接促进IFN-β的转录,从而抑制病毒复制.有研究[51 ] 在对病毒感染的肺上皮细胞进行转录组测序时发现,病毒感染细胞的circRNA AIVR (antagonizes influenza virus replication)表达上调,上调的circRNA AIVR 可吸附miR-330-3p,导致CREBBP表达增加,从而促进IFN-β的产生来抑制流感病毒复制.此外,还有研究发现circRNA是影响病毒与宿主相互作用的重要分子,宿主细胞感染病毒后,病毒基因组合成相关circRNA,从而调控机体免疫应答.circ_3205是SARS-CoV-2在宿主细胞感染时合成的circRNA,可通过海绵吸附miR-298并上调其下游靶基因蛋白激酶Cε(protein kinase C epsilon,PKCε )mRNA的表达,参与SARS-CoV-2的感染免疫[52 ] .该项研究通过对病毒感染后细胞的circRNA进行序列分析,发现了病毒来源的circRNA,为探明病毒性肺炎发病机制提供了新方向. ...
1
... circRNA是由3′和5′末端在反向剪接后共价结合而形成一类特殊的非编码RNA.与线性RNA不同,circRNA为更稳定的封闭环状结构,不受RNA外切酶影响[46 ] .由于其对RNA外切酶活性的内在抗性,circRNA可作为肺炎等疾病诊断和预后的生物标志物[47 -48 ] .此外,circRNA富含miRNA结合位点,可通过充当miRNA海绵,抑制miRNA介导的mRNA的降解,在肺部炎症损伤中发挥重要作用,是肺炎治疗的潜在靶点[49 -50 ] .在病毒感染的细胞中,IRF3和环磷酸腺苷应答元件结合蛋白[cAMP-response element binding protein(CREB)binding protein,CREBBP]形成病毒激活因子的复合物,可直接促进IFN-β的转录,从而抑制病毒复制.有研究[51 ] 在对病毒感染的肺上皮细胞进行转录组测序时发现,病毒感染细胞的circRNA AIVR (antagonizes influenza virus replication)表达上调,上调的circRNA AIVR 可吸附miR-330-3p,导致CREBBP表达增加,从而促进IFN-β的产生来抑制流感病毒复制.此外,还有研究发现circRNA是影响病毒与宿主相互作用的重要分子,宿主细胞感染病毒后,病毒基因组合成相关circRNA,从而调控机体免疫应答.circ_3205是SARS-CoV-2在宿主细胞感染时合成的circRNA,可通过海绵吸附miR-298并上调其下游靶基因蛋白激酶Cε(protein kinase C epsilon,PKCε )mRNA的表达,参与SARS-CoV-2的感染免疫[52 ] .该项研究通过对病毒感染后细胞的circRNA进行序列分析,发现了病毒来源的circRNA,为探明病毒性肺炎发病机制提供了新方向. ...
1
... circRNA是由3′和5′末端在反向剪接后共价结合而形成一类特殊的非编码RNA.与线性RNA不同,circRNA为更稳定的封闭环状结构,不受RNA外切酶影响[46 ] .由于其对RNA外切酶活性的内在抗性,circRNA可作为肺炎等疾病诊断和预后的生物标志物[47 -48 ] .此外,circRNA富含miRNA结合位点,可通过充当miRNA海绵,抑制miRNA介导的mRNA的降解,在肺部炎症损伤中发挥重要作用,是肺炎治疗的潜在靶点[49 -50 ] .在病毒感染的细胞中,IRF3和环磷酸腺苷应答元件结合蛋白[cAMP-response element binding protein(CREB)binding protein,CREBBP]形成病毒激活因子的复合物,可直接促进IFN-β的转录,从而抑制病毒复制.有研究[51 ] 在对病毒感染的肺上皮细胞进行转录组测序时发现,病毒感染细胞的circRNA AIVR (antagonizes influenza virus replication)表达上调,上调的circRNA AIVR 可吸附miR-330-3p,导致CREBBP表达增加,从而促进IFN-β的产生来抑制流感病毒复制.此外,还有研究发现circRNA是影响病毒与宿主相互作用的重要分子,宿主细胞感染病毒后,病毒基因组合成相关circRNA,从而调控机体免疫应答.circ_3205是SARS-CoV-2在宿主细胞感染时合成的circRNA,可通过海绵吸附miR-298并上调其下游靶基因蛋白激酶Cε(protein kinase C epsilon,PKCε )mRNA的表达,参与SARS-CoV-2的感染免疫[52 ] .该项研究通过对病毒感染后细胞的circRNA进行序列分析,发现了病毒来源的circRNA,为探明病毒性肺炎发病机制提供了新方向. ...
1
... circRNA是由3′和5′末端在反向剪接后共价结合而形成一类特殊的非编码RNA.与线性RNA不同,circRNA为更稳定的封闭环状结构,不受RNA外切酶影响[46 ] .由于其对RNA外切酶活性的内在抗性,circRNA可作为肺炎等疾病诊断和预后的生物标志物[47 -48 ] .此外,circRNA富含miRNA结合位点,可通过充当miRNA海绵,抑制miRNA介导的mRNA的降解,在肺部炎症损伤中发挥重要作用,是肺炎治疗的潜在靶点[49 -50 ] .在病毒感染的细胞中,IRF3和环磷酸腺苷应答元件结合蛋白[cAMP-response element binding protein(CREB)binding protein,CREBBP]形成病毒激活因子的复合物,可直接促进IFN-β的转录,从而抑制病毒复制.有研究[51 ] 在对病毒感染的肺上皮细胞进行转录组测序时发现,病毒感染细胞的circRNA AIVR (antagonizes influenza virus replication)表达上调,上调的circRNA AIVR 可吸附miR-330-3p,导致CREBBP表达增加,从而促进IFN-β的产生来抑制流感病毒复制.此外,还有研究发现circRNA是影响病毒与宿主相互作用的重要分子,宿主细胞感染病毒后,病毒基因组合成相关circRNA,从而调控机体免疫应答.circ_3205是SARS-CoV-2在宿主细胞感染时合成的circRNA,可通过海绵吸附miR-298并上调其下游靶基因蛋白激酶Cε(protein kinase C epsilon,PKCε )mRNA的表达,参与SARS-CoV-2的感染免疫[52 ] .该项研究通过对病毒感染后细胞的circRNA进行序列分析,发现了病毒来源的circRNA,为探明病毒性肺炎发病机制提供了新方向. ...
1
... 迄今为止,已在真核细胞中鉴定出100多种类型的RNA化学修饰,包括N 1 -甲基腺苷(N 1 -methyladenosine,m1 A)、m6 A、5-甲基胞嘧啶(5-methylcytosine,m5 C)、N 7 -甲基鸟苷(N 7 -methylguanosine,m7 G)、RNA帽甲基化等[53 ] .其中,m6 A修饰是最丰富也是最保守的RNA修饰.m6 A修饰受到甲基化酶[甲基化转移酶样蛋白3(methyltransferase-like 3,METTL3)、METTL14和Wilms肿瘤1相关蛋白(Wilms' tumor 1-associating protein,WTAP)]、去甲基化酶[脂肪肥胖相关蛋白(fat mass and obesity-associated protein,FTO)和AlkB同源蛋白5(AlkB homologue 5,ALKBH5)]以及阅读蛋白[包括YTH结构域蛋白1(YTH domain containing protein 1,YTHDC1)、YTHDC2、YTH家族蛋白(YTH domain family protein,YTHDF)、胰岛素样生长因子2 mRNA结合蛋白(insulin-like growth factor 2 mRNA binding protein,IGF2BP)家族]三者之间的动态调控[54 ] .m6 A动态调控中的任一环节出现差错均可影响机体的肺部感染免疫应答及炎症反应.TONG等[55 ] 的研究发现,METTL3 敲除的巨噬细胞被脂多糖刺激后,吞噬杀菌能力减弱,产生的TNF-α低于正常水平,并增加了对细菌感染的易感性;然而,有研究[56 ] 表明METTL3通过调控STAT1 mRNA的m6 A修饰来驱动M1型巨噬细胞活化,加重了炎症反应.METTLL3调控的m6 A甲基化修饰对肺炎免疫的影响仍需要进一步研究.YTHDF2是一种m6 A阅读蛋白,有研究[57 ] 发现YTHDF2在脂多糖诱导的巨噬细胞炎症反应中起负性调节作用,敲低YTHDF2 显著促进脂多糖诱导的巨噬细胞促炎因子的表达,如IL-6、TNF-α、IL-1β和IL-12,从而加重肺部的炎症反应.目前研究表明,RNA m6 A修饰可通过调控巨噬细胞的功能影响肺部感染的炎症反应水平,但其具体的分子机制仍需进一步研究.RNA修饰是目前表观遗传研究的新兴领域之一,但目前研究主要集中在肿瘤免疫、哮喘、自身免疫性疾病等,在肺炎免疫细胞中的研究仍较少,迫切需要更多的研究以阐明RNA修饰在肺炎免疫调控中的生物学作用. ...
1
... 迄今为止,已在真核细胞中鉴定出100多种类型的RNA化学修饰,包括N 1 -甲基腺苷(N 1 -methyladenosine,m1 A)、m6 A、5-甲基胞嘧啶(5-methylcytosine,m5 C)、N 7 -甲基鸟苷(N 7 -methylguanosine,m7 G)、RNA帽甲基化等[53 ] .其中,m6 A修饰是最丰富也是最保守的RNA修饰.m6 A修饰受到甲基化酶[甲基化转移酶样蛋白3(methyltransferase-like 3,METTL3)、METTL14和Wilms肿瘤1相关蛋白(Wilms' tumor 1-associating protein,WTAP)]、去甲基化酶[脂肪肥胖相关蛋白(fat mass and obesity-associated protein,FTO)和AlkB同源蛋白5(AlkB homologue 5,ALKBH5)]以及阅读蛋白[包括YTH结构域蛋白1(YTH domain containing protein 1,YTHDC1)、YTHDC2、YTH家族蛋白(YTH domain family protein,YTHDF)、胰岛素样生长因子2 mRNA结合蛋白(insulin-like growth factor 2 mRNA binding protein,IGF2BP)家族]三者之间的动态调控[54 ] .m6 A动态调控中的任一环节出现差错均可影响机体的肺部感染免疫应答及炎症反应.TONG等[55 ] 的研究发现,METTL3 敲除的巨噬细胞被脂多糖刺激后,吞噬杀菌能力减弱,产生的TNF-α低于正常水平,并增加了对细菌感染的易感性;然而,有研究[56 ] 表明METTL3通过调控STAT1 mRNA的m6 A修饰来驱动M1型巨噬细胞活化,加重了炎症反应.METTLL3调控的m6 A甲基化修饰对肺炎免疫的影响仍需要进一步研究.YTHDF2是一种m6 A阅读蛋白,有研究[57 ] 发现YTHDF2在脂多糖诱导的巨噬细胞炎症反应中起负性调节作用,敲低YTHDF2 显著促进脂多糖诱导的巨噬细胞促炎因子的表达,如IL-6、TNF-α、IL-1β和IL-12,从而加重肺部的炎症反应.目前研究表明,RNA m6 A修饰可通过调控巨噬细胞的功能影响肺部感染的炎症反应水平,但其具体的分子机制仍需进一步研究.RNA修饰是目前表观遗传研究的新兴领域之一,但目前研究主要集中在肿瘤免疫、哮喘、自身免疫性疾病等,在肺炎免疫细胞中的研究仍较少,迫切需要更多的研究以阐明RNA修饰在肺炎免疫调控中的生物学作用. ...
1
... 迄今为止,已在真核细胞中鉴定出100多种类型的RNA化学修饰,包括N 1 -甲基腺苷(N 1 -methyladenosine,m1 A)、m6 A、5-甲基胞嘧啶(5-methylcytosine,m5 C)、N 7 -甲基鸟苷(N 7 -methylguanosine,m7 G)、RNA帽甲基化等[53 ] .其中,m6 A修饰是最丰富也是最保守的RNA修饰.m6 A修饰受到甲基化酶[甲基化转移酶样蛋白3(methyltransferase-like 3,METTL3)、METTL14和Wilms肿瘤1相关蛋白(Wilms' tumor 1-associating protein,WTAP)]、去甲基化酶[脂肪肥胖相关蛋白(fat mass and obesity-associated protein,FTO)和AlkB同源蛋白5(AlkB homologue 5,ALKBH5)]以及阅读蛋白[包括YTH结构域蛋白1(YTH domain containing protein 1,YTHDC1)、YTHDC2、YTH家族蛋白(YTH domain family protein,YTHDF)、胰岛素样生长因子2 mRNA结合蛋白(insulin-like growth factor 2 mRNA binding protein,IGF2BP)家族]三者之间的动态调控[54 ] .m6 A动态调控中的任一环节出现差错均可影响机体的肺部感染免疫应答及炎症反应.TONG等[55 ] 的研究发现,METTL3 敲除的巨噬细胞被脂多糖刺激后,吞噬杀菌能力减弱,产生的TNF-α低于正常水平,并增加了对细菌感染的易感性;然而,有研究[56 ] 表明METTL3通过调控STAT1 mRNA的m6 A修饰来驱动M1型巨噬细胞活化,加重了炎症反应.METTLL3调控的m6 A甲基化修饰对肺炎免疫的影响仍需要进一步研究.YTHDF2是一种m6 A阅读蛋白,有研究[57 ] 发现YTHDF2在脂多糖诱导的巨噬细胞炎症反应中起负性调节作用,敲低YTHDF2 显著促进脂多糖诱导的巨噬细胞促炎因子的表达,如IL-6、TNF-α、IL-1β和IL-12,从而加重肺部的炎症反应.目前研究表明,RNA m6 A修饰可通过调控巨噬细胞的功能影响肺部感染的炎症反应水平,但其具体的分子机制仍需进一步研究.RNA修饰是目前表观遗传研究的新兴领域之一,但目前研究主要集中在肿瘤免疫、哮喘、自身免疫性疾病等,在肺炎免疫细胞中的研究仍较少,迫切需要更多的研究以阐明RNA修饰在肺炎免疫调控中的生物学作用. ...
1
... 迄今为止,已在真核细胞中鉴定出100多种类型的RNA化学修饰,包括N 1 -甲基腺苷(N 1 -methyladenosine,m1 A)、m6 A、5-甲基胞嘧啶(5-methylcytosine,m5 C)、N 7 -甲基鸟苷(N 7 -methylguanosine,m7 G)、RNA帽甲基化等[53 ] .其中,m6 A修饰是最丰富也是最保守的RNA修饰.m6 A修饰受到甲基化酶[甲基化转移酶样蛋白3(methyltransferase-like 3,METTL3)、METTL14和Wilms肿瘤1相关蛋白(Wilms' tumor 1-associating protein,WTAP)]、去甲基化酶[脂肪肥胖相关蛋白(fat mass and obesity-associated protein,FTO)和AlkB同源蛋白5(AlkB homologue 5,ALKBH5)]以及阅读蛋白[包括YTH结构域蛋白1(YTH domain containing protein 1,YTHDC1)、YTHDC2、YTH家族蛋白(YTH domain family protein,YTHDF)、胰岛素样生长因子2 mRNA结合蛋白(insulin-like growth factor 2 mRNA binding protein,IGF2BP)家族]三者之间的动态调控[54 ] .m6 A动态调控中的任一环节出现差错均可影响机体的肺部感染免疫应答及炎症反应.TONG等[55 ] 的研究发现,METTL3 敲除的巨噬细胞被脂多糖刺激后,吞噬杀菌能力减弱,产生的TNF-α低于正常水平,并增加了对细菌感染的易感性;然而,有研究[56 ] 表明METTL3通过调控STAT1 mRNA的m6 A修饰来驱动M1型巨噬细胞活化,加重了炎症反应.METTLL3调控的m6 A甲基化修饰对肺炎免疫的影响仍需要进一步研究.YTHDF2是一种m6 A阅读蛋白,有研究[57 ] 发现YTHDF2在脂多糖诱导的巨噬细胞炎症反应中起负性调节作用,敲低YTHDF2 显著促进脂多糖诱导的巨噬细胞促炎因子的表达,如IL-6、TNF-α、IL-1β和IL-12,从而加重肺部的炎症反应.目前研究表明,RNA m6 A修饰可通过调控巨噬细胞的功能影响肺部感染的炎症反应水平,但其具体的分子机制仍需进一步研究.RNA修饰是目前表观遗传研究的新兴领域之一,但目前研究主要集中在肿瘤免疫、哮喘、自身免疫性疾病等,在肺炎免疫细胞中的研究仍较少,迫切需要更多的研究以阐明RNA修饰在肺炎免疫调控中的生物学作用. ...
1
... 迄今为止,已在真核细胞中鉴定出100多种类型的RNA化学修饰,包括N 1 -甲基腺苷(N 1 -methyladenosine,m1 A)、m6 A、5-甲基胞嘧啶(5-methylcytosine,m5 C)、N 7 -甲基鸟苷(N 7 -methylguanosine,m7 G)、RNA帽甲基化等[53 ] .其中,m6 A修饰是最丰富也是最保守的RNA修饰.m6 A修饰受到甲基化酶[甲基化转移酶样蛋白3(methyltransferase-like 3,METTL3)、METTL14和Wilms肿瘤1相关蛋白(Wilms' tumor 1-associating protein,WTAP)]、去甲基化酶[脂肪肥胖相关蛋白(fat mass and obesity-associated protein,FTO)和AlkB同源蛋白5(AlkB homologue 5,ALKBH5)]以及阅读蛋白[包括YTH结构域蛋白1(YTH domain containing protein 1,YTHDC1)、YTHDC2、YTH家族蛋白(YTH domain family protein,YTHDF)、胰岛素样生长因子2 mRNA结合蛋白(insulin-like growth factor 2 mRNA binding protein,IGF2BP)家族]三者之间的动态调控[54 ] .m6 A动态调控中的任一环节出现差错均可影响机体的肺部感染免疫应答及炎症反应.TONG等[55 ] 的研究发现,METTL3 敲除的巨噬细胞被脂多糖刺激后,吞噬杀菌能力减弱,产生的TNF-α低于正常水平,并增加了对细菌感染的易感性;然而,有研究[56 ] 表明METTL3通过调控STAT1 mRNA的m6 A修饰来驱动M1型巨噬细胞活化,加重了炎症反应.METTLL3调控的m6 A甲基化修饰对肺炎免疫的影响仍需要进一步研究.YTHDF2是一种m6 A阅读蛋白,有研究[57 ] 发现YTHDF2在脂多糖诱导的巨噬细胞炎症反应中起负性调节作用,敲低YTHDF2 显著促进脂多糖诱导的巨噬细胞促炎因子的表达,如IL-6、TNF-α、IL-1β和IL-12,从而加重肺部的炎症反应.目前研究表明,RNA m6 A修饰可通过调控巨噬细胞的功能影响肺部感染的炎症反应水平,但其具体的分子机制仍需进一步研究.RNA修饰是目前表观遗传研究的新兴领域之一,但目前研究主要集中在肿瘤免疫、哮喘、自身免疫性疾病等,在肺炎免疫细胞中的研究仍较少,迫切需要更多的研究以阐明RNA修饰在肺炎免疫调控中的生物学作用. ...
1
... 固有免疫系统是病毒感染时的第一道防线,必须区分宿主和病原体核酸,才能在不激活自身免疫反应的情况下产生保护性免疫反应.新近研究发现病毒的核苷酸修饰可作为免疫系统识别自身与非自身RNA的分子标记,在多种病毒免疫逃逸中发挥重要作用.LU等[58 ] 在人偏肺病毒(human metapneumovirus,HMPV)的研究中发现,具有m6 A修饰的HMPV RNA与细胞质RNA传感器维甲酸诱导基因-Ⅰ(retinoic acid-inducible gene-Ⅰ,RIG-Ⅰ )的结合能力减弱,IFN-Ⅰ生成受阻,进而HMPV复制和基因表达增加;这是病毒免疫逃逸的潜在机制之一.LU等[59 ] 对人呼吸道合胞病毒及流感病毒的研究也证实这一结论.与具有m6 A修饰的病毒相比,研究构建的m6 A修饰缺陷的人呼吸道合胞病毒及流感病毒感染细胞后能够以RIG-Ⅰ依赖的方式产生更高水平的IFN-Ⅰ;进一步研究证实,m6 A修饰缺陷的病毒能更有效地与RIG-Ⅰ结合,增强RIG-Ⅰ泛素化并促进RIG-Ⅰ构象重排和寡聚化.RNA m6 A修饰在肺炎病毒科和副黏病毒科中普遍保守,可作为增强IFN-Ⅰ反应的新策略,在病毒感染治疗和疫苗开发中具有重要应用. ...
1
... 固有免疫系统是病毒感染时的第一道防线,必须区分宿主和病原体核酸,才能在不激活自身免疫反应的情况下产生保护性免疫反应.新近研究发现病毒的核苷酸修饰可作为免疫系统识别自身与非自身RNA的分子标记,在多种病毒免疫逃逸中发挥重要作用.LU等[58 ] 在人偏肺病毒(human metapneumovirus,HMPV)的研究中发现,具有m6 A修饰的HMPV RNA与细胞质RNA传感器维甲酸诱导基因-Ⅰ(retinoic acid-inducible gene-Ⅰ,RIG-Ⅰ )的结合能力减弱,IFN-Ⅰ生成受阻,进而HMPV复制和基因表达增加;这是病毒免疫逃逸的潜在机制之一.LU等[59 ] 对人呼吸道合胞病毒及流感病毒的研究也证实这一结论.与具有m6 A修饰的病毒相比,研究构建的m6 A修饰缺陷的人呼吸道合胞病毒及流感病毒感染细胞后能够以RIG-Ⅰ依赖的方式产生更高水平的IFN-Ⅰ;进一步研究证实,m6 A修饰缺陷的病毒能更有效地与RIG-Ⅰ结合,增强RIG-Ⅰ泛素化并促进RIG-Ⅰ构象重排和寡聚化.RNA m6 A修饰在肺炎病毒科和副黏病毒科中普遍保守,可作为增强IFN-Ⅰ反应的新策略,在病毒感染治疗和疫苗开发中具有重要应用. ...