包括HCC在内的实体瘤中,由于肿瘤细胞快速生长而血管新生速度又无法及时匹配,肿瘤细胞通常处于低氧状态[4-5]。低氧微环境与肿瘤转移和不良预后密切相关,是许多实体瘤恶性进展和侵袭性转移的促进因素[6]。低氧诱导因子1(hypoxia inducible factor 1,HIF1)是一个由α-和β-亚基组成的异二聚体,是细胞适应低氧环境至关重要的调节因子。在缺氧条件下,HIF1α快速累积并被转运至细胞核,与HIF1β形成一个稳定的HIF1复合物,特异性结合到HIF1靶基因的启动子区域的低氧反应元件(hypoxia response element,HRE)A/GCGTG上,进而诱导靶基因转录[7]。HIF1靶基因编码的蛋白参与肿瘤发生的多个方面,包括葡萄糖和能量代谢、增殖、血管生成、侵袭和转移等。HIF1通路的激活与HCC在内的恶性肿瘤的侵袭性和不良临床预后密切相关[8]。
长链非编码RNA(long non-coding RNA,lncRNA)是细胞内一类长度大于200个核苷酸(nt)且不编码蛋白的RNA[9]。研究[10-11]表明,lncRNA主要通过与细胞大分子(如染色质DNA、蛋白质和RNA)的相互作用发挥功能。迄今为止,已有大量与癌症相关的lncRNA被报道能够调节肿瘤的生长、侵袭和转移,并被认为是癌症的潜在替代性生物标志物和治疗靶点[12-13]。虽然lncRNA在肿瘤中的功能与机制已有较多报道,但目前仍有大量lncRNA的功能未被注释。本研究通过大规模筛选发现此前尚未被报道的低氧诱导的lncRNA 68(hypoxia induced lncRNA 68,HILRNA68;基因名为TCONS_00027424,位于人类第19号染色体:55 524 358~55 526 406)在低氧下的多株HCC细胞系中表达水平显著升高,并通过细胞生长计数实验、双荧光素酶报告基因实验等方法,明确HILRNA68在低氧下的功能和相关机制。
1 材料与方法
1.1 实验材料
1.1.1 细胞系与质粒
人肝癌细胞SMMC-7721、PLC/PRF/5、MHCC-97H、MHCC-97L、Bel-7402,人胚肾293T(HEK-293T)细胞,均购自中国科学院细胞库。pGL3-basic质粒、pCAG-dR8.9包装质粒、pMDG(VSV-G)包膜蛋白质粒均来自Addgene全球质粒共享信息库。
1.1.2 主要试剂
胎牛血清(Sigma,德国),TRIzol(Invitrogen,美国),反转录随机引物(TaKaTa,日本),M-MLV反转录酶(Promega,德国),SYBRTM Green PCR预混液(Applied Biosystems,美国),硝酸纤维素膜(Bio-Rad,德国)。HIF1α鼠单克隆抗体(货号610958;BD,美国),HIF1β兔单克隆抗体(货号NB100-110)、HIF2α兔单克隆抗体(货号NB100-122)、丙酮酸脱氢酶激酶1(pyruvate dehydrogenase kinase 1,PDK1)兔单克隆抗体(货号MAB864)(Novus,美国),β-actin兔单克隆抗体(货号GB15003;武汉赛维尔生物科技有限公司,中国)。辣根过氧化物酶标记的二抗(Cell Signaling Technology,美国),ECL化学发光试剂盒(Merck Millipore,德国),荧光素酶检测试剂盒(Promega,美国),Transwell细胞培养小室(Corning,美国),RNA探针(广州锐博公司,中国),4%多聚甲醛、磷酸盐缓冲液(上海碧云天生物技术有限公司,中国)。
1.1.3 主要仪器
生物安全柜、CO2细胞培养箱来自美国Thermo公司,qPCR仪来自美国Applied Biosystems公司,荧光显微镜和Leica TCS Sp8STED共聚焦显微镜来自德国Leica公司。
1.2 实验方法
1.2.1 细胞培养与低氧处理细胞
细胞培养使用添加10%胎牛血清的高糖培养基,培养在细胞培养箱(37 ℃,5%CO2)中。使用6孔板进行培养,每孔种入3×105个细胞。细胞贴壁后放入低氧培养箱(1%O2,5%CO2,37 ℃)培养,按照实验需求确定低氧培养时间。
1.2.2 LncRNA芯片检测
收集肝癌细胞系SMMC-7721分别经常氧(normoxia)、低氧(hypoxia)处理12 h的3批RNA样本,进行lncRNA芯片检测。将芯片检测结果通过R语言DEseq2程序包进行分析,筛选低氧下表达变化的lncRNA。
1.2.3 蛋白质印迹法
细胞样品用添加溴酚蓝的1×SDS细胞裂解液裂解。将细胞样品在105 ℃下加热20 min,获得蛋白样品。采用蛋白质印迹法(Western blotting)进行实验。将蛋白样品用十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)分离,将凝胶上的蛋白转移到硝酸纤维素膜。用1×磷酸盐缓冲液配置5%脱脂牛奶,室温封闭1 h。按实验需要孵育一抗(稀释比例为1∶1 000),4 ℃孵育过夜。次日孵育辣根过氧化物酶标记的二抗(稀释比例为1∶2 000),室温孵育1 h。使用辣根过氧化物酶底物试剂盒进行显影。
1.2.4 实时荧光定量PCR
用TRIzol裂解细胞,每毫升TRIzol加入267 μL三氯甲烷,离心后将含有RNA的上清液转移到新的EP管中,并加入等量异丙醇,抽提mRNA。定容后将RNA反转录为互补DNA(complementary DNA,cDNA)。将cDNA、2×SYBR、上游引物、下游引物和无菌水充分混合,加入384孔板中。每组设置3个复孔,开始实时荧光定量PCR(quantitative real-time polymerase chain reaction,qRT-PCR)检测。按照反应程序进行扩增:95 ℃,30 s;95 ℃,5 s;60 ℃,20 s;72 ℃,30 s;60 ℃,1 min。进行40次循环。使用2-ΔΔCT方法分析目的基因的表达量。引物序列见表1。
表1 qRT-PCR引物序列
Tab 1
| Primer | Forword (5'→3') | Reverse (5'→3') |
|---|---|---|
| HILRNA44 | GGAACGAACTCTTGCGTGTG | AGGAGCGCACTTCCTCATTC |
| HILRNA68 | AAGCGGAGCCTGAAGATGTG | CCAACCCGGCTGCTACTTTAT |
| GLUT1 | CTGTCGTGTCGCTGTTTGTG | AAAGATGGCCACGATGCTCA |
| PDK1 | CTGGCTGTGGCTTCTCTAGC | CCGAAGTCCAGGAACTGCTT |
| PGK1 | TGTGGTCCTGAAAGCAGCAA | AGTTGACTTAGGGGCTGTGC |
| BNIP3L | GGACTCGGCTTGTTGTGTTG | TCGACTAGGTGGGACGACAT |
| VEGF | TAAGTCCTGGAGCGTTCCCT | ACGCGAGTCTGTGTTTTTGC |
| 18S rRNA | GTAACCCGTTGAACCCCATT | CCATCCAATCGGTAGTAGCG |
1.2.5 短发夹RNA稳转敲除细胞系构建
构建带有特定短发夹RNA(short hairpin RNA,shRNA)片段的敲除质粒。将生长密度大约80%的HEK-293T细胞用于慢病毒包装。取用灭菌的1.5 mL EP管,加入24 μL聚乙烯亚胺(polyethylenimine,PEI)和400 μL的Opti-MEM减血清培养基,振荡混匀后室温静置5 min。将pCAG-dR8.9包装质粒(4 μg)、pMDG (VSV-G)包膜蛋白质粒(2 μg)和目的质粒(6 μg)加入1.5 mL EP管中,加入400 μL的Opti-MEM减血清培养基,充分混匀后静置5 min。向质粒悬液中加入424 μL静置后的PEI悬液。振荡混匀后,室温静置15 min,缓慢均匀滴加到预先准备好的HEK-293T细胞的培养皿中,在细胞培养箱中继续培养。24 h后更换新鲜完全培养基。收集24 h和48 h的病毒液。将病毒液加入SMMC-7721和PLC/PRF/5的细胞培养皿中。培养48 h后加入嘌呤霉素筛选阳性细胞。检测HILRNA68的转录水平。成功构建的敲除细胞系用于后续实验。靶向序列见表2。
表2 shRNA靶向序列
Tab 2
| shRNA | Sequence (5'→3') |
|---|---|
| shHIF1α#1 | CCGCTGGAGACACAATCATAT |
| shHIF1α#2 | GTGATGAAAGAATTACCGAAT |
| shHIF1β#1 | GGCTCAAGGAGATCGTTTATT |
| shHIF1β#2 | GCCTACACTCTCCAACACAAT |
| shHIF2α | CAGTACCCAGACGGATTTCAA |
1.2.6 小干扰RNA转染实验
设计合成小干扰RNA(small interfering RNA,siRNA)。将细胞培养至生长密度为70%,用转染试剂Jet-PRIME将siRNA转染到细胞中。转染后对细胞进行恢复培养。48 h后在转录水平进行敲除效率检测。靶向序列见表3。
表3 siRNA靶向序列
Tab 3
| siRNA | Sequence (5'→3') |
|---|---|
| siNC | TTGTTACTCGCTCACATTTAA |
| si#1 | CGTACCTGGCCTTAGTAATTT |
| si#2 | GAGCCTGAAGATGTGATTAAA |
1.2.7 细胞生长计数实验
将HILRNA68敲低细胞及对照细胞分别种植在6孔板中,每孔2×105个细胞。次日起连续4 d检测细胞数量并统计分析。
1.2.8 Transwell细胞侵袭实验
将基质胶在冰上融化,用10倍体积的DMEM高糖培养基稀释基质胶。取100 μL稀释后的基质胶加入Transwell的上室中,37 ℃培养箱孵育2 h,待基质胶凝固后备用。取5×104个HILRNA68敲低细胞及其对照细胞,分别用200 μL不含胎牛血清的培养基重悬细胞,加入Transwell小室的上室。下室加入500 μL含10%胎牛血清的培养基,培养24 h。用4%多聚甲醛固定细胞,0.1%结晶紫染色10 min,将两室隔膜上室一侧的细胞擦净。用去离子水洗去浮色,使用扫描仪扫描细胞图像。使用ImageJ软件统计细胞数量。
1.2.9 双荧光素酶报告基因实验
将HILRNA68的启动子区(-2 000 nt~0)构建到萤火虫荧光素酶(firefly luciferase)报告基因pGL3-basic质粒中。取HILRNA68敲除(敲除组)及其对照细胞系(空载组),共转染含有萤火虫荧光素酶和海肾荧光素酶(renilla luciferase)的双荧光素酶质粒,培养24 h后收集样品。加入裂解液,室温裂解细胞15 min。向40 µL的荧光素酶检测试剂Ⅱ(luciferase assay reagent Ⅱ)中加入10 µL细胞裂解液,吹打混匀后,检测读数,即为萤火虫荧光素酶的活性。加入40 µL Stop&Glo试剂,再次读数,即为海肾荧光素酶的活性。计算每管萤火虫荧光素酶/海肾荧光素酶活性的比值(Luc/Ren)。再将空载组的比值定为单位1,即可得到敲除组相对于空载组的荧光素酶活性,也就是该敲除组基因转录的调控活性。
1.2.10 细胞核浆分离实验
用低渗缓冲液重悬4×107个细胞,在冰上静置10 min,用匀浆器匀浆,垂直匀浆40次。将匀浆后的液体转移到干净离心管中,1 000×g 4 ℃离心10 min,乳白色沉淀为细胞核成分,上清液为细胞浆成分。
1.2.11 RNA荧光原位杂交技术实验
用4%多聚甲醛固定细胞10 min,洗去多余的固定液,加入1 mL预冷的通透液,透化5 min后行RNA荧光原位杂交技术(RNA fluorescence in situ hybridization,RNA-FISH)实验。在切片背面圈定原位杂交的范围,用预杂交液封闭20 min,将切片置于避光湿盒中,吸取杂交液充分覆盖切片,杂交1 h。用洗液洗去多余的杂交液。加入荧光染料4',6-二脒基-2-苯基吲哚(4',6-diamidino-2-phenylindole,DAPI)染色,避光孵育5 min,洗去多余的DAPI染料。加入含有防荧光淬灭剂的封片剂,用盖玻片盖好后封片。在激光共聚焦荧光显微镜下观察,进行图像采集。
1.3 统计学分析
数据采用GraphPad Prism 9.0软件进行统计学分析。组内比较采用多重独立样本t检验。P<0.05表示差异具有统计学意义。
2 结果
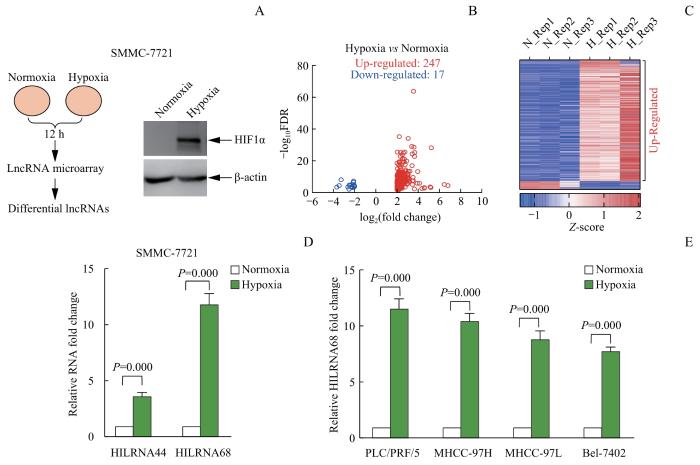
2.1 低氧下HILRNA68在肝癌细胞中表达显著升高
为寻找肝癌细胞中受低氧调控且有功能的lncRNA,本研究通过lncRNA芯片检测筛选低氧调控的lncRNA(图1A)。根据芯片结果,首先排除低丰度RNA(6个样本中表达总和<180),然后定义低氧组相较常氧对照组倍数变化(fold change)≥4,伪发现率(false discovery rate,FDR)≤0.05为低氧下表达显著变化的lncRNA。据此我们分别得到显著升高和降低的lncRNA 247个和17个(图1B、1C)。在SMMC-7721细胞中通过qRT-PCR验证,我们发现HILRNA44(基因名为uc003xew.3,位于人类第8号染色体:26 236 777~26 240 469)和HILRNA68(分别为芯片检测结果中升高水平位于第44和第68位)在低氧下均显著升高(均P=0.000),尤其是HILRNA68的表达量升高达到10倍以上(图1D)。考虑到HILRNA68在低氧下表达量升高倍数大、表达丰度高更便于后续研究,因而本研究重点关注其在肝癌细胞中的功能及其相关机制。根据UCSC(University of California,Santa Cruz Genome Browser Database)数据库的收录,HILRNA68在白细胞,以及肾上腺、睾丸、脑等组织中表达较高,而在肝脏中表达较低。这提示,HILRNA68在正常肝脏中的表达较低,而当发生肿瘤后可能由于低氧微环境的出现而升高。随后,在PLC/PRF/5、MHCC-97H、MHCC-97L、Bel-7402等多株肝癌细胞系中验证其低氧下的表达变化,更充分证明了HILRNA68在低氧下的显著升高(均P<0.05,图1E)在肝癌细胞中是普遍存在的,这初步提示其可能对肝癌细胞适应低氧微环境的过程中发挥重要功能。
图1
图1
低氧下HILRNA68在肝癌细胞中表达显著升高
Note:
Fig 1
HILRNA68 was up-regulated in HCC cells under hypoxia
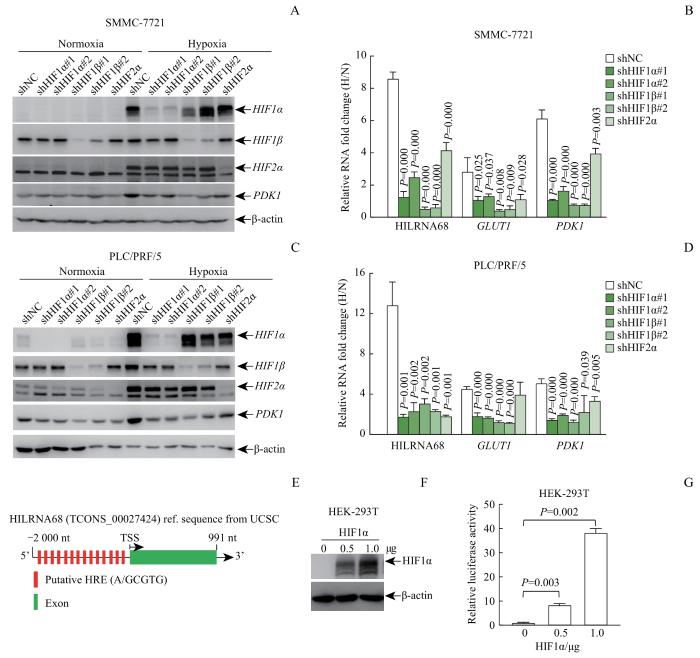
2.2 低氧下HILRNA68在肝癌细胞中的表达受低氧诱导因子调控
为研究HILRNA68在低氧下表达升高的分子机制,考虑到低氧诱导因子(hypoxia inducible factors,HIFs)是细胞适应低氧环境至关重要的转录因子,本研究在SMMC-7721(图2A)以及PLC/PRF/5(图2C)2株细胞中通过shRNA敲低了HIF1α、HIF1β以及HIF2α。通过将以上稳转细胞低氧处理并收集RNA进行qRT-PCR检测,发现HILRNA68与HIFs经典的靶基因葡萄糖转运蛋白1(glucose transporter 1,GLUT1)以及PDK1[7]类似,在SMMC-7721(图2B)以及PLC/PRF/5(图2D)2株细胞中敲低HIF1α、HIF1β以及HIF2α后其低氧下的升高均被显著抑制(均P<0.05)。以上结果提示低氧环境下HILRNA68的转录受HIFs调控。接下来为进一步明确HILRNA68是否为HIFs的靶基因,通过检测HILRNA68的启动子区,发现其-2 000 nt~0区共有14个潜在的HRE(图2E),这提示HILRNA68可能是HIFs的靶基因。随后,将HILRNA68的启动子区序列插入报告基因载体并通过与HIF1α的过表达载体共转后进行双荧光素酶报告基因实验(图2F、G),结果提示,HILRNA68的启动子区的报告基因活性随着HIF1α的表达升高而增强(图2G),这说明低氧环境下HIF1α可能直接结合在HILRNA68的启动子区并启动其表达。
图2
图2
低氧下HILRNA68在肝癌细胞中的表达受HIFs调控
Note: A. Western blotting analysis of SMMC-7721 cells indicated the knockdown efficiency of HIF1α/HIF2α/HIF1β. B. qRT-PCR analysis of indicated RNAs of SMMC-7721 cells with or without knockdown HIF1α/HIF2α/HIF1β under hypoxia. The P values indicate the differences between each group and the control group (shNC). C. Western blotting analysis of PLC/PRF/5 cells indicated the knockdown efficiency of HIF1α/HIF2α/HIF1β. D. qRT-PCR analysis of indicated RNA of PLC/PRF/5 cells with or without knockdown HIF1α/HIF2α/HIF1β under hypoxia. The P values indicate the differences between each group and the control group (shNC). E. Reference sequence diagram of HILRNA68. TSS—transcription start site. F. Western blotting analysis of HEK-293T cells transfected with HIF1α over-expression plasmid. G. Dual-luciferase assay results of HEK-293T transfected with HIF1α over-expression plasmid. H/N—hypoxia/normoxia.
Fig 2
Expression of HILRNA68 in HCC under hypoxia was regulated by HIFs
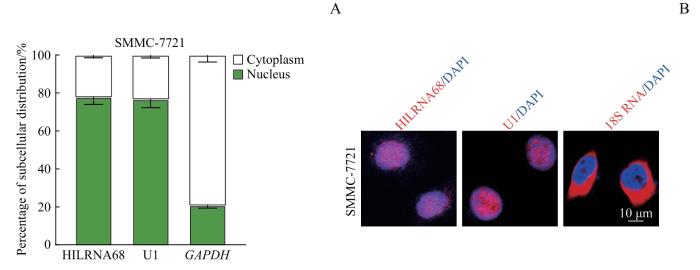
2.3 HILRNA68主要定位于细胞核
图3
图3
HILRNA68主要定位于细胞核
Note: A. Histogram about subcellular distribution of indicated RNAs in SMMC-7721 cells. U1 as a nucleus marker, GAPDH as a cytoplasm marker. B. Fluorescence of RNA-FISH results of SMMC-7721 hybridized with HILRNA68. DAPI indicates nucleus.
Fig 3
HILRNA68 was mainly located in the nucleus
表3 HILRNA68的编码潜力分析
Tab 3
| RNA | Coding probability | Label |
|---|---|---|
| HILRNA68 | 0.009 4 | Noncoding |
| HIF1α | 1.000 0 | Coding |
| XIST | 0.219 1 | Noncoding |
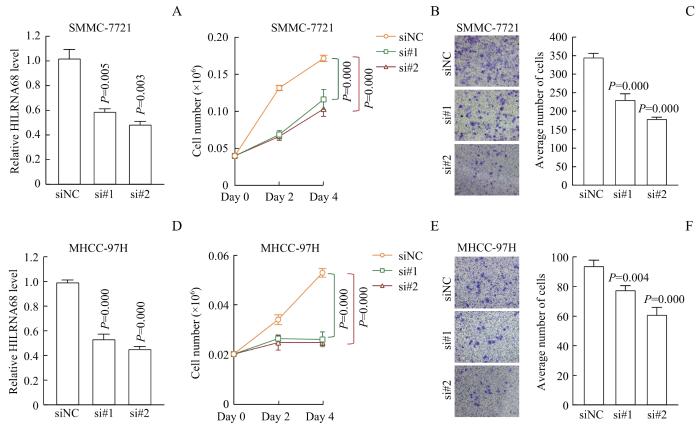
2.4 HILRNA68低氧下促进肝癌细胞的生长增殖与侵袭转移
为研究HILRNA68在低氧下的功能,我们设计了2个针对其的siRNA si#1与si#2,并在SMMC-7721(图4A)与MHCC-97H(图4D)2株细胞中对其进行了敲低,发现2个siRNA尤其是si#2,均能抑制其表达50%以上(图4A、D)。首先,进行低氧下细胞生长计数实验,结果提示,敲低HILRNA68后显著抑制SMMC-7721(图4B)与MHCC-97H(图4E)在低氧下的增殖(均P=0.000)。考虑到低氧条件下,肿瘤细胞会发生上皮间质转化进而侵袭能力增强[15],我们还开展了基质胶包被的Transwell细胞侵袭实验,用以研究HILRNA68是否参与调控肿瘤细胞的侵袭转移能力。通过实验发现,敲低HILRNA68后,SMMC-7721(图4C)与MHCC-97H(图4F)低氧下的侵袭转移能力均显著降低(均P<0.05)。以上结果说明,低氧下HILRNA68的升高会增强肝癌细胞低氧下的增殖与侵袭转移能力。
图4
图4
HILRNA68低氧下抑制肝癌细胞的生长增殖与侵袭转移
Note: A. qRT-PCR analysis of HILRNA68 knockdown efficiency in SMMC-7721. B. Cell proliferation results of SMMC-7721 cells with or without HILRNA68 knock-down. C. Histogram of Transwell cell invasion analysis of SMMC-7721 cells with or without HILRNA68 knock-down. D. qRT-PCR analysis of HILRNA68 knockdown efficiency in MHCC-97H. E. Cell proliferation results of MHCC-97H cells with or without HILRNA68 knock-down. F. Histogram of Transwell cell invasion analysis results of MHCC-97H cells with or without HILRNA68 knock-down. The P value indicates the differences between each group and the control group (siNC
Fig 4
HILRNA68 inhibited the tumor proliferation, invasion and metastasis of hepatocellular carcinoma cells under hypoxia
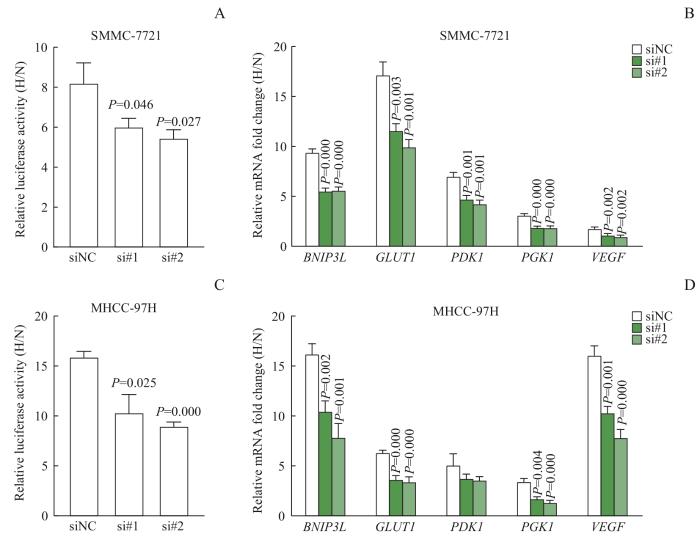
2.5 HILRNA68调控HIFs的转录活性
HIF1是细胞适应低氧微环境至关重要的转录因子,其调控肿瘤细胞低氧下的细胞增殖与侵袭转移能力。为研究HILRNA68在低氧下抑制细胞增殖与侵袭转移的分子机制,我们首先探究敲低HILRNA68是否会抑制低氧下HIF1α的转录活性。通过构建经典的HRE到报告基因载体,并进行双荧光素酶报告基因实验,我们发现,在SMMC-7721(图5A)与MHCC-97H(图5C)细胞中HILRNA68敲低后,HRE低氧下的报告基因活性相较于对照组被显著抑制(均P<0.05),这提示HILRNA68的敲低抑制了低氧下HIF1α的转录活性。为进一步证明以上结果,通过qRT-PCR检测SMMC-7721(图5B)与MHCC-97H(图5D)敲低HILRNA68后HIFs靶基因的升高,发现这些经典的靶基因如BNIP3L、GLUT1、PDK1、PGK1、VEGF等[15]低氧下的升高均在一定程度上被抑制(图5B、D)。以上结果说明,HILRNA68低氧下升高后参与促进HIFs的转录活性,帮助细胞适应低氧微环境。
图5
图5
HILRNA68调控HIFs的转录活性
Note: A. Luciferase activity of SMMC-7721 cells with or without HILRNA68 knock-down under hypoxia. B. qRT-PCR analysis of indicated mRNAs in SMMC-7721 under hypoxia. C. Luciferase activity of MHCC-97H cells with or without HILRNA68 knock-down under hypoxia. D. qRT-PCR analysis of indicated mRNAs in MHCC-97H cells under hypoxia. The P values indicate the differences between each group and the control group (siNC).
Fig 5
HILRNA68 regulated the transcriptional activity of HIFs
3 讨论
本研究中,我们通过lncRNA芯片寻找肝癌细胞中受低氧调控的lncRNA,并鉴定出目前尚未被报道的HILRNA68。深入研究发现,HILRNA68为HIFs的靶基因并促进低氧下细胞的生长增殖与侵袭转移;机制研究结果提示HILRNA68调控HIFs的转录活性。
肿瘤细胞,尤其是实体瘤细胞,具备快速且不受约束的增殖能力从而导致血管系统供应不足,因此其倾向于处于低氧的肿瘤微环境中[16]。低氧下,HIF1α快速累积并与HIF1β结合形成异二聚体[17],进而调控具有不同功能的靶基因的表达以帮助细胞适应低氧微环境,如GLUT1、HK2(hexokinase 2)、LDHA(lactate dehydrogenase A)、PDK1、PGK1等靶基因调节细胞的能量代谢过程[18];VEGF等促进血管新生[19]。本研究通过lncRNA芯片的大规模筛选,在肝癌细胞系中鉴定出一批在低氧下表达显著变化的lncRNA。其中lncRNA HILRNA68更是在多株细胞系中低氧下升高约10倍;调控机制研究发现其启动子区有14个HRE(A/GCGTG),并进一步证明HIF1α直接调控其表达。据此我们认为HILRNA68是一个新的HIF1α的靶基因。此外,我们注意到,在敲低HIF2α后,HILRNA68低氧下的表达升高同样被抑制,提示HILRNA68不仅是HIF1α的靶基因,同时也直接受HIF2α调控。
据报道,非编码RNA约占真核细胞转录组的90%,而这其中lncRNA有近7万个[20]。截至目前被功能注释的lncRNA仍然很少。迄今为止HILRNA68尚无文献报道。本研究除率先证明其直接受HIFs调控外,还通过siRNA敲低后的细胞功能实验证明其在低氧下的升高参与调控肝癌细胞低氧下的生长增殖与侵袭转移。考虑到低氧下HIFs调控的靶基因调控细胞在低氧下的增殖与侵袭能力,我们在随后的机制研究中通过双荧光素酶报告基因实验证明HILRNA68低氧下升高后可增强HIFs的转录活性。靶基因检测表明,低氧下敲低HILRNA68后显著抑制HIFs的靶基因如GLUT1、PGK1、VEGF等的升高。根据以上结果,我们初步推测低氧下肝癌细胞内HIFs与HILRNA68之间存在正反馈调节——低氧下HIFs转录激活HILRNA68的表达,而升高的HILRNA68进一步增强HIFs的转录活性,最终促进肝癌细胞的生长增殖与侵袭转移。HILRNA68调控HIFs转录活性的具体机制目前尚不明确。低氧下lncRNA调控HIFs的转录活性的机制研究已有相关报道[21-25]。如低氧下lncRNA HIFAL通过促进HIF1α的mRNA表达从而增强其转录活性[24];lncRNA HABON通过与HIF1α的结合并促进其降解,通过减少HIF1α低氧下的累积而抑制其活性[22]。这些报道提示HILRNA68可能通过增强HIFs的mRNA表达或者蛋白质稳定性进而增强其转录活性。据此,后续研究拟通过在敲除HILRNA68的细胞中检测HIFs的mRNA以及蛋白质水平,明确HILRNA68是否通过调节HIFs的表达量而调控其转录活性。也有研究报道,低氧下lncRNA调控HIFs蛋白质翻译后修饰从而增强其转录活性,例如lincRNA-p21通过破坏希佩尔林道蛋白(von Hippel-Lin dau,VHL)与HIF1α相互作用来稳定HIF1α[26]。我们猜测HILRNA68也有可能通过影响HIFs蛋白质翻译后修饰的方式调控其转录活性。目前,抑制HIF1α功能的药物已经在临床应用,例如硼替佐米和蒽环类药物。由于HIF信号通路影响广泛,药物不良反应严重。低氧下,HILRNA68被HIFs诱导表达,又能正反馈增强HIFs的转录活性,通过靶向降解HILRNA68,可以破坏HIFs信号的放大效应。靶向药物的研发中,通常用小分子或抗体靶向蛋白靶点,但受限于蛋白结构复杂,无法找到合适的结合位点,存在成药困难的问题。RNA靶向药物则不存在这类问题,因此针对HILRNA68设计反义寡核苷酸(antisense oligonucleotides,ASO),靶向降解实体瘤的HILRNA68对癌症治疗具有潜在价值。
低氧下肿瘤细胞中HIFs与HILRNA68之间的正反馈调节,最终促进了肿瘤细胞的生长增殖和侵袭转移。在肿瘤的发展进程中,正反馈调控模式具有重要作用。它通过形成自我增强的循环,加速肿瘤的发生和进展。PI3K/AKT信号通路、Wnt/β-catenin信号通路和Notch信号通路都是能够形成正反馈回路的肿瘤信号通路,能促进细胞增殖和肿瘤生长。肿瘤细胞释放的一些生长因子和细胞因子可以促进血管新生,形成肿瘤血管网,从而为肿瘤提供充足的营养,进一步促进肿瘤的生长和转移。正反馈调控模式在肿瘤发生发展中的重要作用体现在促进肿瘤生长增殖、血管新生、抑制凋亡和抗药性产生等方面,深入研究正反馈调控在肿瘤发展中的作用机制,有助于寻找新的治疗策略和靶点,提高肿瘤治疗效果。
在研究中,我们使用shRNA敲低HIFs,获得HIFLRNA68与HIF1α调控关系以及HILRNA68相关功能的结论。为了更真实全面地说明HILRNA68在低氧环境下发挥的功能,我们需要针对HILRNA68的基因组序列设计sgRNA,在基因组上实现敲除,再通过功能回复实验补充缺失的基因,明确细胞的功能是否恢复正常。我们的研究结论是基于肝癌细胞的体外培养实验得出的。然而,生物体内的肿瘤微环境复杂,调控肿瘤生长的机制多种多样。因此,尽管我们观察到HILRNA68在体外实验中受HIFs调控并参与正反馈调节,但在生物体内发挥的功能是否与体外实验一致仍然存在不确定性。因此,为了进一步验证我们的研究结论,我们需要进行更多的动物实验。低氧环境是实体瘤中常见的肿瘤微环境之一。我们的研究揭示了肝癌中HILRNA68与HIFs的相关机制,这种机制在肝癌以外的实体瘤中是否具有类似的作用需要更多的研究结果佐证。
综上所述,对HILRNA68与HIFs调控模式的深入研究,不仅有助于我们更好地理解肝癌的发生发展机制,还为未来开发针对HIFs和HILRNA68的治疗策略提出了重要的理论依据。HILRNA68作为HIFs的靶基因通过增强HIFs的转录活性促进肝癌细胞的增殖和侵袭,这一发现为肝癌的治疗和预防提供了新的思路和方向,为肝癌患者的治疗带来更多希望。
作者贡献声明
谭露负责大部分生物学实验操作、论文撰写,何平负责整体课题设计、结果分析以及论文撰写,沈少明参与课题讨论与指导。所有作者均阅读并同意了最终稿件的提交。
AUTHOR's CONTRIBUTIONS
TAN Lu performed the majority of biological experiments and wrote the paper. HE Ping designed the project and was responsible for result analysis and paper writing. SHEN Shaoming participated in project discussions. All authors have read the final manuscript and agreed to the submission.
利益冲突声明
所有作者声明本研究不存在利益冲突。
COMPETING INTERESTS
All authors disclose no relevant conflict of interests.
参考文献