乳腺癌是女性发病率最高的肿瘤,也是导致女性死亡的主要原因之一,在2020年超过肺癌成为女性癌症相关死亡的首要原因[1]。临床上,根据雌激素受体(estrogen receptor,ER)、孕激素受体(progesterone receptor,PR)和人类表皮生长因子受体2(human epidermal growth factor receptor 2,HER2)的表达情况,乳腺癌易感基因(breast cancer susceptibility gene,BRCA)表现为不同的亚型[2],包括激素受体阳性者(表达ER和/或PR)、HER2阳性或三阴性者(不表达ER、PR、HER2)[3]。乳腺癌的治疗手段包括手术治疗、放射治疗、化学治疗(简称化疗)、内分泌治疗和靶向治疗等。针对乳腺癌各主要病理亚型的治疗策略改善了许多乳腺癌患者的预后,除了化疗之外,ER阳性乳腺癌采用激素治疗,HER2基因扩增的乳腺癌采用HER2靶向药物治疗。近年来,乳腺癌的治疗仍然是一个挑战性的问题,尽管新的治疗方法和药物不断出现,但大多数患者仍然会在一段时间后出现复发或转移。因此,研究乳腺癌复发和转移的分子机制,寻找新的靶点,完善乳腺癌的有效筛查、诊断和治疗是目前我们面临的艰巨任务。
蛋白质精氨酸甲基化是一种重要的蛋白翻译后修饰,由一组蛋白质精氨酸甲基转移酶(protein arginine methyltransferase,PRMT)催化,在癌症的发展和转移中起着关键作用[4]。哺乳动物PRMT家族有9个成员。在哺乳动物细胞中发现的甲基化精氨酸主要有3种形式:Ⅰ型PRMT[PRMT1、PRMT2、PRMT3、CARM1(coactivator-associated arginine methyltransferase 1)/PRMT4、PRMT6和PRMT8]催化精氨酸残基的不对称二甲基化;Ⅱ型PRMT(PRMT5和PRMT9)主要催化对称二甲基化;Ⅲ型PRMT(PRMT7)介导单甲基化[5],主要参与DNA修复和转录、RNA加工及核苷酸-细胞质定位调控[6]等。研究[7]报道精氨酸甲基化家族成员在多种肿瘤中起重要作用。PRMT6是一种Ⅰ型甲基化转移酶,发生不对称二甲基精氨酸修饰,在细胞核中主要介导组蛋白修饰标记组蛋白H3第2位精氨酸不对称二甲基化(asymmetric dimethylation of histone H3 arginase 2,H3R2me2a)抑制基因表达[8]。此外,PRMT6还参与非组蛋白的精氨酸甲基化。HUANG等[9]研究表明PRMT6在精氨酸214位点甲基化染色质凝聚调节因子1(regulator of chromosome condensation 1,RCC1),抑制PRMT6表达可降低胶质母细胞瘤(glioblastoma,GBM)细胞的致瘤性。WONG等[10]研究报道PRMT6介导钙响应因子(calcium response factor,CaRF)发生甲基化,影响肝细胞癌的有氧糖酵解过程。目前关于PRMT6在乳腺癌中的作用研究较少。本研究结合生物信息学分析,检测PRMT6在乳腺癌中的表达水平,并通过体外实验验证PRMT6对乳腺癌细胞增殖和迁移能力的影响,探究其可能的作用机制,以期为乳腺癌的精准治疗提供新的生物标志物。
1 材料与方法
1.1 组织标本、主要材料和试剂
27例乳腺癌组织及其配对癌旁组织的组织芯片来自上海交通大学医学院附属第一人民医院。乳腺癌细胞MCF-7、SK-BR-3、MDA-MB-231、SUM159PT、SUM149PT和正常乳腺上皮细胞MCF-10A均购于中国科学院典型培养物保藏委员会细胞库。
免疫组织化学法(immunohistochemistry,IHC)试剂盒购自北京中杉金桥生物技术有限公司,达尔伯克改良伊格尔培养基(Dulbecco's modified Eagle's medium,DMEM)、胎牛血清、双抗、胰酶购自美国Gibco公司,细胞转染试剂Lipo3000购自美国赛默飞公司,RNA提取试剂盒购自苏州新赛美生物科技有限公司,RNA反转录试剂盒购自南京诺唯赞生物科技股份有限公司,实时荧光定量PCR试剂盒购自上海翌圣生物科技有限公司,PRMT6 siRNA购自安徽通用生物公司,兔源PRMT6抗体、兔源波形蛋白(Vimentin)抗体、兔源E-钙黏蛋白(E-cadherin)抗体购自美国Proteintech公司,兔源β-actin抗体购自美国Abclonal公司,兔源细胞周期蛋白D1(cyclin D1)抗体、鼠源N-钙黏蛋白(N-cadherin)抗体购自美国Cell Signaling Technology公司,Transwell小室购自美国Corning公司。
1.2 主要仪器
生物安全柜和细胞培养箱(美国赛默飞公司),光学显微镜(日本Nikon公司),离心机(德国Eppendorf公司),荧光定量PCR仪(美国ABI公司)。
1.3 实验方法
1.3.1 生物信息学分析
从癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库中下载33种癌组织与癌旁组织的转录组测序数据,采用R 4.0.1软件的TCGAplot包对组织中PRMT6 mRMA表达水平进行分析;利用基因表达谱交互分析(Gene Expression Profiling Interactive Analysis,GEPIA2)在线数据库查询PRMT6在乳腺癌组织和正常乳腺组织中的表达;利用人类蛋白质图谱(The Human Protein Atlas,HPA)数据库查询正常乳腺组织及乳腺癌组织类型,挑选组织完整且染色良好的IHC图片。下载基因表达综合(Gene Expression Omnibus,GEO)数据库中GSE210948数据集,GEO2R在线分析差异基因。通过clusterProfiler R包进行差异表达基因的京都基因与基因组数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路富集分析。
1.3.2 组织芯片中PRMT6表达量的检测
采用IHC对组织芯片中的乳腺癌组织和癌旁组织进行分析。将组织芯片置于65 ℃烘箱中烘烤1 h,经二甲苯脱蜡、乙醇梯度水化后,用抗原修复液水浴修复30 min,冷却至室温。采用内源性过氧化酶阻断剂阻断后进行一抗(PRMT6抗体,工作浓度为1∶50)4 ℃过夜孵育。加入反应增强液、增强酶标山羊抗小鼠/兔免疫球蛋白G(immunoglobulin G,IgG)聚合物分别孵育20 min,二氨基联苯胺(diaminobenzidine,DAB)显色,随后苏木精染细胞核,最后乙醇脱水后滴加中性树脂封片。统计PRMT6阳性细胞比例及染色程度,并进行病理评分。判读标准如下:①根据阳性染色深度,无明显阳性为0分,弱阳性为1分,中等强度阳性为2分,强阳性为3分。②根据阳性表达PRMT6的肿瘤上皮细胞的数量,无阳性细胞表达为0分,阳性细胞数量占视野全部肿瘤上皮细胞比例的0~25%为1分,阳性细胞数量占视野全部肿瘤上皮细胞比例的26%~50%为2分,阳性细胞数量占视野全部肿瘤上皮细胞比例的51%~75%为3分,阳性细胞数量占视野全部肿瘤上皮细胞比例的76%~100%为4分。最终得分情况由阳性染色深度和阳性细胞比例占比相乘得到。
1.3.3 细胞培养
细胞均使用DMEM在含有5% CO2的37 ℃恒温培养箱中培养。培养基中添加10%胎牛血清及100 U/mL青霉素和100 μg/mL链霉素。
1.3.4 细胞转染
取生长良好的细胞在6孔板中铺板,第2天细胞密度达70%左右为宜,小干扰RNA(small interfering RNA,siRNA)control、siPRMT6#1、siPRMT6#2采用lipofectamine3000转染,转染6 h后更换完全培养基,48 h后收集细胞用于后续实验。转染实验的siRNA序列见表1。
表1 siRNA序列
Tab 1
| siRNA | Forward (5'→3') | Reverse (5'→3') |
|---|---|---|
| Control | UUCUCCGAACGUGUCACGUTT | ACGUGACACGUUCGGAGAATT |
| siPRMT6#1 | CGGAACAGGUGGAUGCCAUTT | AUGGCAUCCACCUGUUCCGTT |
| siPRMT6#2 | CGGUGCAAGUGGAGCAAGATT | UCUUGCUCCACUUGCACCGTT |
1.3.5 细胞计数试剂盒8(cell counting kit-8,CCK-8)实验
将对照组细胞和敲低组细胞按照每孔1 000个接种于96孔板中,每组设置3个复孔,周边孔加入等体积PBS,置于细胞培养箱中。待细胞贴壁生长后,分别于第0、1、2、4日在每孔中加入10 μL的CCK-8试剂,孵育1 h后,检测每孔在450 nm波长处的吸光度值。根据吸光度值绘制细胞生长曲线。
1.3.6 平板克隆形成实验
将对照组细胞和敲低组细胞按照1 000个/孔种于6孔板中。每日观察细胞状态,3~4 d进行1次细胞换液,培养10~14 d。撤去培养基,PBS清洗2次,每孔加入4%多聚甲醛固定15 min,弃甲醛,加入1%结晶紫溶液染色20 min。染色完成后用PBS清洗3遍,晾干。拍照并统计细胞克隆数。
1.3.7 细胞划痕实验
采用细胞划痕实验检测已转染细胞的迁移能力。将对照组细胞和敲低组细胞接种于6孔板中,当细胞密度达95%时,使用10 μL枪头尖端刮划细胞产生划痕,将培养基更换为无血清培养基继续培养24~48 h,使用倒置显微镜记录伤口愈合的情况。
1.3.8 Transwell实验
采用8 μm聚对苯二甲酸乙二醇酯[poly (ethylene terephthalate),PET](聚酯)膜Corning Transwell inserts插入式细胞培养皿,收集转染24 h后的对照组和敲低组细胞进行计数,按照105个/孔细胞密度接种于insert小室中,共200 μL细胞悬液,下室加入600 μL 20% DMEM培养基。培养24 h后,取出小室进行固定染色,最后擦去小室内部未迁移到下面的细胞,晾干,使用高倍显微镜进行拍照计数。
1.3.9 RNA提取和实时荧光定量PCR(quantitative real-time PCR,qRT-PCR)检测
收集转染24 h后的各组细胞,使用RNA提取试剂盒提取总RNA,使用反转录试剂盒合成cDNA,real-time PCR反应体系20 μL:SYBR Green Master Mix 10 μL,上游引物(10 μmol/L)0.4 μL,下游引物(10 μmol/L)0.4 μL,模板cDNA 1 μL、ddH2O 8.2 μL。反应条件:预变性95 ℃ 5 min;变性95 ℃ 10 s,退火/延伸60 ℃ 30 s,循环40次。PRMT6正向引物序列为5'-TACCGCCT GGGTATCCTTCG-3',反向引物序列为5'-CCTGTTC CGGCAACTCTACA-3';GAPDH正向引物序列为5'-GGAGCGAGATCCCTCCAAAAT-3',反向引物序列为5'-GGCTGTTGTCATACTTCTCATGG-3';CCND1正向引物序列为5'- CCGTCCATGCGGAAGATC-3',反向引物序列为5' -GAAGACCTCCTCCTCGCACT-3'。采用2-ΔΔCT法计算基因的相对表达量。
1.3.10 Western blotting
收集转染48 h后的细胞,用含有蛋白酶抑制剂和磷酸酶抑制剂的裂解缓冲液在冰上提取细胞总蛋白,按照比例加入上样缓冲液,煮沸10 min进行蛋白变性,获得蛋白。取适量蛋白样品加入10%的SDS-PAGE胶的泳道内,恒压80 V进行电泳,后于100 V/90 min条件下转膜,5%脱脂奶粉室温封闭1 h,加入适量一抗(工作浓度按官网说明书配制)4 ℃冰箱内过夜孵育;TBST洗涤后加入二抗(工作浓度为1∶5 000)室温孵育1 h,再次TBST溶液洗涤。按1∶1比例配制发光显影液,使用化学发光检测仪进行曝光。使用ImageJ软件分析灰度值。
1.3.11 细胞周期分析
收集转染48 h后的细胞,PBS洗涤细胞2遍,用300 mL PBS重悬细胞,缓慢滴加700 mL预冷的无水乙醇,4 ℃固定过夜。将固定的细胞高速离心,PBS洗涤2遍后,100 mL PBS重悬细胞,加入核糖核酸酶A(ribonuclease A,RNase A)和碘化丙啶(propidium iodide,PI)染料,常温避光孵育30 min。用BD Fortessa流式细胞仪进行检测。
1.4 统计学方法
使用GraphPad Prism 9.0软件进行统计分析。定量数据以x±s表示,组间比较采用Student′s t检验或非参数检验。P<0.05为差异具有统计学意义。
2 结果
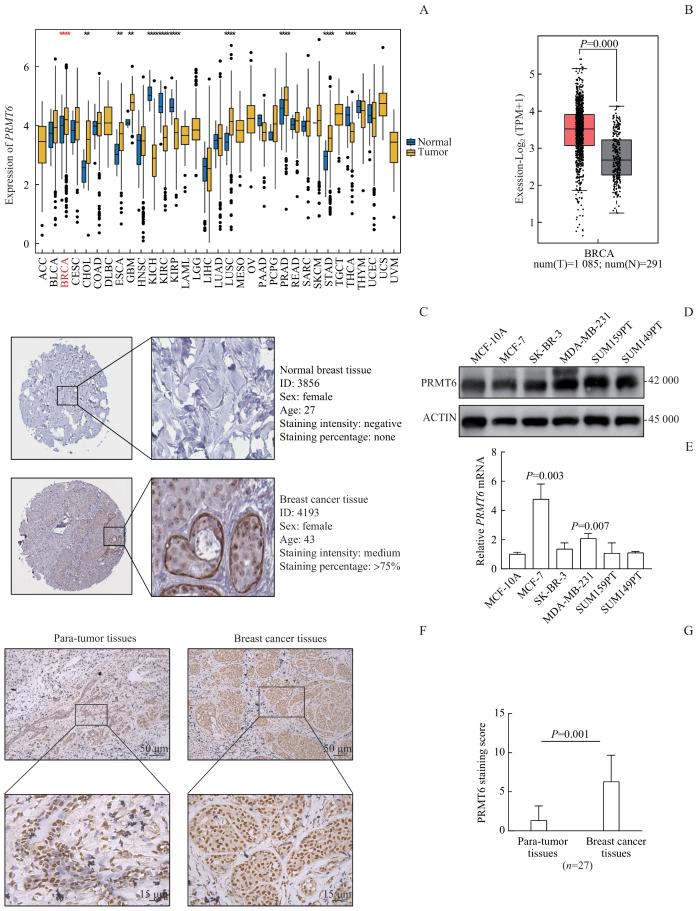
2.1 PRMT6 在乳腺癌中高表达
首先,对PRMT6进行泛癌种表达分析,利用TCGAplot R包对来自TCGA数据库中33种癌组织及其癌旁组织的转录组测序数据进行可视化分析,结果发现相较于正常组织,PRMT6 mRNA在包括乳腺癌在内的多种原发肿瘤组织中高表达(图1A)。进一步通过GEPIA2在线数据库中的乳腺癌临床数据分析显示,与正常乳腺组织相比,乳腺癌组织中的PRMT6表达显著增加(图1B)。此外,通过HPA数据库获得了PRMT6在正常乳腺组织和乳腺癌组织中的表达情况,结果显示乳腺癌组织中的PRMT6蛋白表达水平明显上升,与正常乳腺组织相比呈现出明显差异(图1C)。使用IHC检测了27例临床乳腺癌组织及其配对癌旁组织中PRMT6蛋白的表达情况,结果发现在乳腺癌组织中PRMT6的表达水平高于配对癌旁组织中的表达,差异具有统计学意义(图1F、G,P=0.001)。以上结果说明PRMT6在乳腺癌组织中高表达。随后,采用Western blotting和qRT-PCR检测PRMT6在乳腺癌细胞系MCF-7、SK-BR-3、MDA-MB-231、SUM159PT、SUM149PT和正常乳腺上皮细胞MCF-10A中的表达,结果显示,与MCF-10A细胞相比,PRMT6在乳腺癌细胞中明显高表达(图1D、E)。
图1
图1
PRMT6 在乳腺癌中高表达
Note: A. Expression of PRMT6 mRNA in different primary cancers from TCGA database. B. GEPIA2 database was used to analyze the relative expression of PRMT6 mRNA in breast cancer tissues and normal breast tissues. T represents breast cancer tissue samples (red), and N represents the normal breast tissue samples (black). C. Representative images of the immunohistochemical level of PRMT6 in breast cancer tissues and normal breast tissues from the HPA database. D/E. Detection of the expression level of PRMT6 in different breast cancer cells by Western blotting (D) and qRT-PCR (E). F. Detection of PRMT6 expression in breast cancer tissues and para-tumor tissues by IHC. G. PRMT6 staining score in breast cancer tissues and para-tumor tissues.
Fig 1
PRMT6 was upregulated in breast cancer
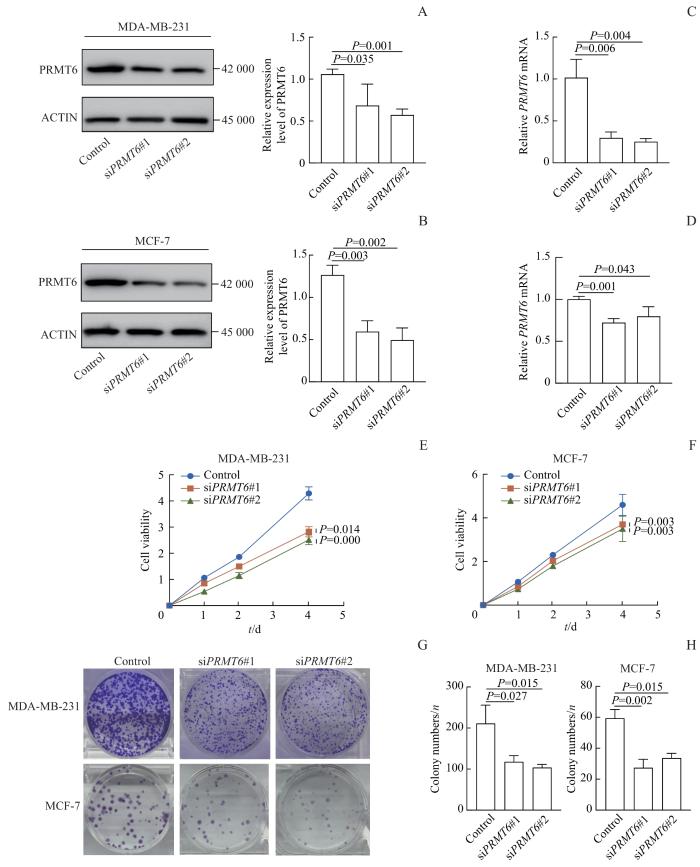
2.2 沉默 PRMT6 抑制乳腺癌细胞的增殖
图2
图2
沉默 PRMT6 抑制乳腺癌细胞的增殖
Note: A/B. Transfection efficiency of PRMT6 siRNAs in MDA-MB-231 cell line (A) and MCF-7 cell line (B) verified by Western blotting. C/D. Transfection efficiency of PRMT6 siRNAs in MDA-MB-231 cell line (C) and MCF-7 cell line (D) detected by qRT-PCR. E/F. Effect of PRMT6 siRNA on the proliferation of MDA-MB-231 and MCF-7 cells detected by CCK-8 assay. G. Representative images of colony formation in PRMT6 knock down MDA-MB-231 cells and MCF-7 cells. H. Colony formation efficiency of MDA-MB-231 and MCF-7 cell lines after 10-d incubation.
Fig 2
Deletion of PRMT6 suppressed breast cancer cell proliferation
为进一步证实PRMT6表达对乳腺癌细胞增殖的影响,对已转染的两种乳腺癌细胞系进行克隆形成实验。结果显示,沉默PRMT6表达后,MDA-MB-231和MCF-7细胞的克隆形成能力均显著降低(图2G、H;P=0.027,P=0.015;P=0.002,P=0.015)。以上结果表明,敲低PRMT6抑制乳腺癌细胞增殖。
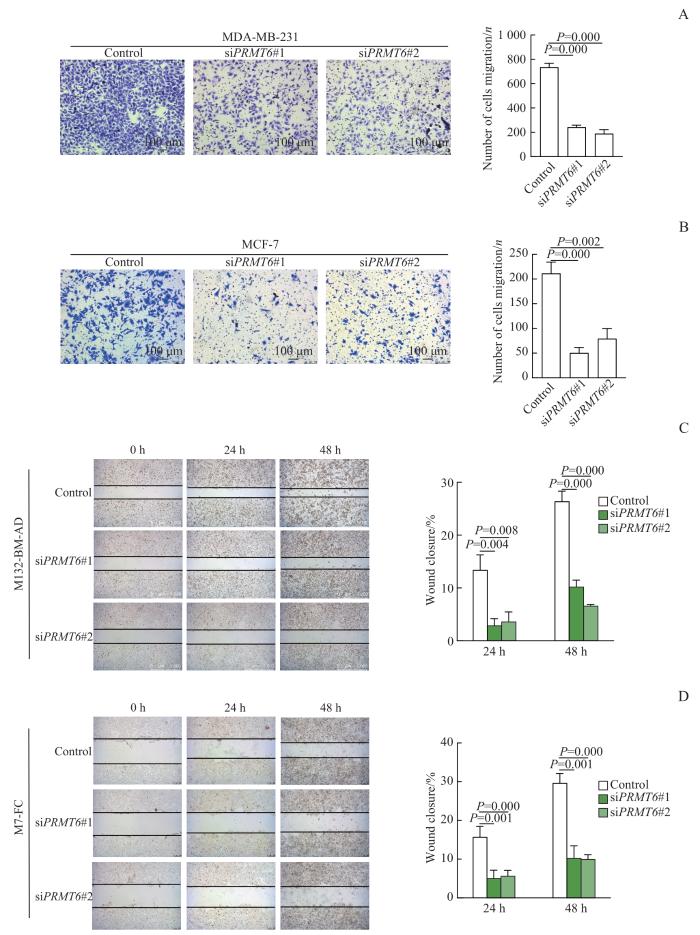
2.3 沉默 PRMT6 抑制乳腺癌细胞的迁移
图3
图3
沉默 PRMT6 抑制乳腺癌细胞的迁移
Note: A/B. After knocking down PRMT6 in MDA-MB-231 and MCF-7 cells, Transwell assay was performed to detect the migration ability of MDA-MB-231 cell line (A) and MCF-7 cell line (B). C/D. After knocking down PRMT6 in MDA-MB-231 and MCF-7 cells, wound healing assay was performed to detect the migration ability of MDA-MB-231 cell line (C) and MCF-7 cell line (D).
Fig 3
Deletion of PRMT6 suppressed breast cancer cell migration
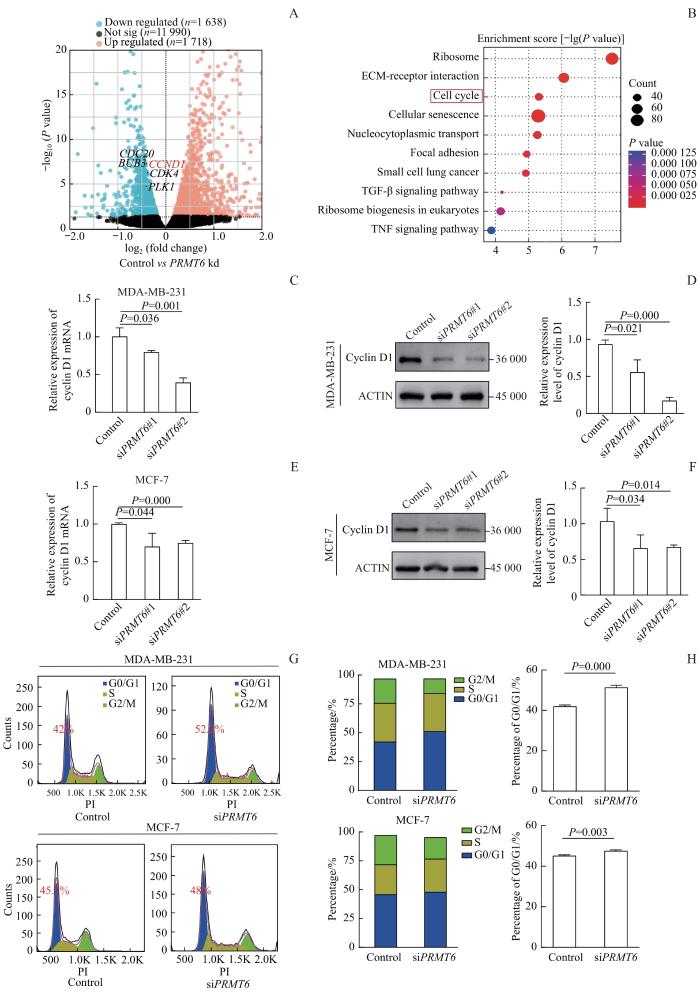
2.4 PRMT6 调控细胞周期通路
为了探究PRMT6的表达影响肿瘤细胞增殖的可能机制,本研究分析了GEO数据库中的GSE210948数据集[11]。结果显示(图4A),敲低PRMT6表达后,有1 638个基因下调,1 718个基因上调。对这些差异基因进行KEGG通路富集分析,富集到的排序靠前的通路包括细胞周期通路、ECM-受体相互作用通路、癌症通路、TNF信号通路等癌症相关的通路(图4B)。结果发现细胞周期通路中细胞分裂周期20(cell division cycle 20,CDC20)、BUB3有丝分裂检查点蛋白(BUB3 mitotic checkpoint protein,BUB3)、细胞周期蛋白D1(cyclin D1,CCND1)、细胞周期蛋白依赖性激酶4(cyclin dependent kinase 4,CDK4)、E2F转录因子4(E2F transcription factor 4,E2F4)、Polo样激酶1(polo like kinase 1,PLK1)、细胞周期蛋白B1(cyclin B1,CCNB1)、酪氨酸3-单加氧酶/色氨酸5-单加氧酶激活蛋白(tyrosine 3-monooxygenase/tryptophan 5-monooxygenase activation protein eta,YWHAH)、细胞周期蛋白H(cyclin H,CCNH)基因表达下调(P<0.05),其中,CCND1是差异化表达最显著的基因之一。CCND1作为一种经典的细胞周期调节因子,在癌症的发病机制中起核心作用[12]。有研究报道CCND1在乳腺癌发生阶段起到重要作用,并且与乳腺癌转移密切相关[13]。因此,将选择CCND1进行下一步研究。
图4
图4
PRMT6 调控细胞周期通路
Note: A. Volcano plot of differentially expressed genes between the control and PRMT6 knockdown groups. B. KEGG analysis of differentially expressed genes between the control and PRMT6 knockdown groups. C/E. After knocking down PRMT6 in MDA-MB-231 and MCF-7 cells, transcription level of cyclin D1 in MDA-MB-231 (C) and MCF-7 cells (E) was detected by qRT-PCR. D/F. After knocking down PRMT6 in MDA-MB-231 and MCF-7 cells, expression of cyclin D1 in MDA-MB-231 (D) and MCF-7 cells (F) was detected by Western blotting. G/H. Cell cycle analysis was performed after PRMT6 knockdown in MDA-MB-231 (G) and MCF-7 cells (H).
Fig 4
Regulation of PRMT6 on the cell cycle pathway
为了探究PRMT6表达对乳腺癌细胞增殖的影响是否和细胞周期通路相关,本研究对细胞周期通路中的关键基因cyclin D1的表达进行检测。使用qRT-PCR对已转染PRMT6 siRNA(siPRMT6#1、siPRMT6#2)的MDA-MB-231和MCF-7细胞中cyclin D1的转录水平进行检测,结果显示,与对照组相比,敲低PRMT6后,cyclin D1的转录水平显著下降,差异有统计学意义(图4C、E,P=0.036,P=0.001;P=0.044,P=0.000)。同时,使用Western blotting检测cyclin D1的蛋白水平,结果显示,PRMT6表达下调后,cyclin D1蛋白水平也明显下调,差异具有统计学意义(图4D、F;P=0.021,P=0.000;P=0.034,P=0.014)。本研究又利用流式细胞术进行了细胞周期分析,结果发现敲低PRMT6后,G0/G1期的细胞数量增加(P=0.000,P=0.003),G2/M期的细胞减少(图4G、H)。以上结果说明,PRMT6可能影响cyclin D1的转录和蛋白水平,进而通过调控细胞周期通路中的G1/S转变,促进乳腺癌细胞的增殖。
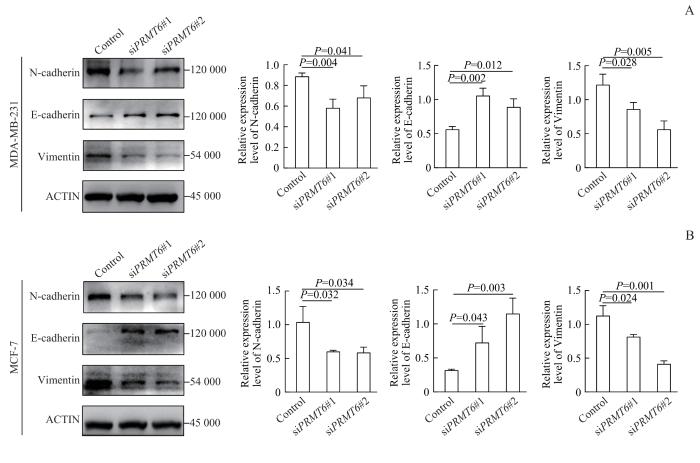
2.5 PRMT6 表达对乳腺癌细胞EMT相关蛋白的影响
上皮-间充质转化(epithelial-mesenchymal transition,EMT)是肿瘤发展中的一个重要环节,与肿瘤的复发转移密切相关。EMT是上皮细胞失去其形态和功能逐渐转化为间质样细胞的过程[14],上皮表型标志物E-cadherin和间质表型标志物N-cadherin、Vimentin起到关键作用。为了进一步分析PRMT6的表达影响乳腺癌细胞迁移的潜在机制,使用Western blotting检测EMT相关蛋白的表达。结果显示,在已转染PRMT6 siRNA的MDA-MB-231和MCF7细胞中,下调PRMT6的表达时,E-cadherin表达水平上升,N-cadherin和Vimentin的表达水平均下降,差异有统计学意义(图5A、B,均P<0.05)。以上结果说明,敲低PRMT6的表达可以抑制乳腺癌细胞的EMT过程。
图5
图5
敲低 PRMT6 后EMT相关蛋白的表达
Note: A/B. Expression of EMT-associated proteins in MDA-MB-231 (A) and MCF-7 cells (B) detected by Western blotting after knocking down PRMT6.
Fig 5
Expression of EMT-related proteins after PRMT6 knockdown
3 讨论
肿瘤转移是癌症相关死亡的主要原因。作为危害女性健康的第一大疾病,乳腺癌的复发转移是多因素、多途径导致,其具体的相关机制还未明确。因此,本研究选择乳腺癌作为研究对象,从中挖掘与其发生和发展有关的关键分子,进一步探索乳腺癌潜在的治疗靶点。PRMT家族介导的催化作用在基因转录调控中发挥着非常重要的作用,特别是精氨酸甲基化对于基因表达的调控。深入研究PRMT家族有助于阐明精氨酸甲基化在肿瘤的发生和发展中的作用及其分子机制。
本研究首先分析TCGA数据库中PRMT6 mRNA在多种肿瘤中的表达情况,发现PRMT6在包括乳腺癌在内的多个部位的原发肿瘤组织高表达;进一步通过生物信息学在线数据库GEPIA2和HPA发现,相比于正常乳腺上皮组织,乳腺癌中PRMT6蛋白的表达显著增加;同时利用免疫组织化学染色检测了27例乳腺癌组织及其配对癌旁组织中PRMT6的表达,结果显示PRMT6蛋白在乳腺癌组织中的表达显著高于癌旁组织,提示PRMT6在乳腺癌中可能作为一种促癌基因起作用。为了探究PRMT6在乳腺癌细胞中的生物学功能,选择PRMT6表达量相对较高的人乳腺癌细胞系MDA-MB-231和MCF-7,利用小干扰siRNA技术沉默PRMT6后,结果发现乳腺癌细胞的增殖能力有显著降低。通过对GSE210948数据集中的转录组数据进行KEGG分析,发现PRMT6与细胞周期通路相关。为了进一步证实分析的结果,本研究对细胞周期通路中的关键基因cyclin D1在mRNA水平和蛋白水平进行验证,发现敲低PRMT6后,cyclin D1 mRNA表达和蛋白表达均下调。并且通过细胞周期分析发现敲低PRMT6后,G0/G1期细胞增多,G2/M期细胞减少。细胞周期蛋白D1是细胞周期的关键调控因子,它在调节从G1期到S期的细胞周期过渡中发挥重要作用。异常的细胞周期蛋白D1表达导致细胞增殖调节机制失控,导致肿瘤发生、发展[20]。有文献报道在白血病细胞中,PRMT6与转录因子LEF1共同调节cyclin D1的活性,影响肿瘤细胞的增殖和凋亡信号通路,进而影响肿瘤的生长和扩散[21]。本研究发现,PRMT6可能影响cyclin D1的转录和蛋白水平,进而通过调控细胞周期通路中G1/S转变,促进乳腺癌细胞的增殖。但PRMT6对细胞周期蛋白D1的调控究竟是通过组蛋白甲基化直接影响其表达,还是通过非组蛋白甲基化修饰起作用,仍需进一步的分子机制研究。
为了探究PRMT6的表达对乳腺癌细胞迁移的影响,本研究通过Transwell实验和细胞划痕实验发现,沉默PRMT6明显抑制了乳腺癌细胞的迁移能力,并且EMT相关上皮表型标志物E-cadherin表达增加、间质表型标志物N-cadherin和Vimentin表达减少,提示PRMT6可能通过调控EMT相关蛋白的表达来促进乳腺癌细胞的迁移,但其具体的调控机制仍需进一步的研究。
综上,本研究发现PRMT6在乳腺癌组织中高表达,PRMT6过表达可促进乳腺癌细胞的增殖和迁移。初步探究其可能的分子机制提示PRMT6可能通过影响细胞周期蛋白D1的表达,调控细胞周期通路,促进乳腺癌的进展。随着药物开发工具的不断发展,针对PRMT6的特定抑制剂研发已取得一定进展。EPZ020411作为PRMT6第一种有效的小分子抑制剂,可降低体内H3R2me2a水平[22],目前为止,EPZ020411仍处于临床前研究阶段。我们将进一步完善PRMT6小分子抑制剂对乳腺癌发生和发展的作用,开展更深层次的分子机制研究以及体内相关实验,为乳腺癌的筛查与治疗提供更多可能性。
作者贡献声明
韩依杉负责实验设计和实验操作,徐梓淇负责生物信息学分析,韩依杉、陶梦玉、范广建和余波负责论文的写作和修改。所有作者均阅读并同意了最终稿件的提交。
AUTHOR's CONTRIBUTIONS
The study design and experiment were completed by HAN Yishan. Bioinformatics analysis was performed by XU Ziqi. The manuscript was drafted and revised by HAN Yishan, TAO Mengyu, FAN Guangjian and YU Bo. All the authors have read the last version of paper and consented for submission.
利益冲突声明
所有作者声明不存在利益冲突。
COMPETING INTERESTS
All authors disclose no relevant conflict of interests.
参考文献