液态活检作为新兴检测手段,通过血液等体液检测癌症标志物如循环肿瘤细胞(circulating tumor cell,CTC),循环核酸(circulating nucleic acid,CNA),细胞外囊泡(extracellular vesicle,EV),肿瘤诱导的血小板、蛋白质和代谢产物等[1-3],能够助力癌症筛查、分型与病程监测[4-5]。游离DNA(cell-free DNA,cfDNA)因其甲基化模式、突变、融合、拷贝数变异等特征已用于非侵入性的疾病诊断和预后评估[6-10];同时,游离RNA(cell-free RNA,cfRNA)、微RNA(microRNA,miRNA)、环状RNA(circularRNA,circRNA),以及信使RNA(messenger RNA,mRNA)和长链非编码RNA(long non-coding RNA,lncRNA)也可作为生物标志物[11-14]。液体活检在癌症诊断、预后评估中展现巨大潜力[15],推进了癌症早期发现与治疗策略的优化[16]。然而,液体活检面临血液中游离核酸含量少且个体差异大[17]、cfDNA和cfRNA片段化[18]等挑战,传统的实时荧光定量PCR(quantitative real-time PCR,qPCR)等检测方法难以精准捕捉并有效分析这些低丰度分子。因此,提高血液中核酸提取量成为亟待解决的关键问题。目前,商业化cfDNA与RNA提取试剂盒多基于离心柱法和磁珠法[19],但在准确性、效率及成本上仍有改进空间。
针对血液中核酸提取的低含量、高变异性与成本效益等痛点,本研究系统且较为全面地比较不同试剂盒及方法在白细胞基因组DNA(genomic DNA,gDNA)、血浆cfDNA及cfRNA提取中的表现。本研究采用qPCR作为标准化检测技术,精确测定各提取方法下核酸的提取量。通过合理的实验设计与对比分析,不仅聚焦于cfDNA与cfRNA提取的优化,还将白细胞gDNA提取纳入考量,以期构建一个全面覆盖血液样本中不同形式核酸提取的体系。本研究旨在筛选出高效、准确且成本效益佳的提取方案,为科研工作者提供实用指南,帮助提升实验效率和数据可靠性,并加速血液生物标志物研究的临床转化。
1 对象与方法
1.1 研究对象
纳入2020年至2024年期间在多家医院收集的145例血液样本。样本包括来自温州医科大学附属第一医院神经内科的20例阿尔茨海默病患者(样本编号1~20)及12例健康对照(样本编号34~45),来自上海交通大学医学院附属第九人民医院骨科的5例纤维化患者(样本编号27~31),以及来自复旦大学附属肿瘤医院大肠外科的108例结直肠癌患者(其中8例为Ⅱ期结直肠癌患者,样本编号21~26、32、33;其余100例为Ⅲ、Ⅳ期结直肠癌患者)。
1.2 方法
1.2.1 主要试剂及仪器
MolPure® Cell/Tissue DNA Kit(Kit A)、MolPure® Blood RNA Kit(Kit E)、Hieff UNICON® Universal Blue qPCR SYBR Green Master Mix购自翌圣生物科技(上海)股份有限公司,QIAamp MinElute Virus Spin Kit(Kit B)购自美国QIAGEN,VAHTS Serum/Plasma Circulating DNA Kit(Kit C)购自南京诺唯赞生物科技股份有限公司,敏迈®游离DNA提取试剂盒(Kit D)、敏迈®游离RNA提取试剂盒(Kit F)购自艾铂图生物(江苏)有限公司,SuperScript Ⅳ VILO Master Mix购自美国Thermo Fisher,Liferivere磁珠法试剂盒购自上海之江生物科技股份有限公司。
EX3600 Plus核酸提取仪购自上海之江生物科技股份有限公司,CFX Opus96 qPCR仪购自美国Bio-Rad,Nano-300微量分光光度计购自杭州奥盛仪器有限公司。
1.2.2 血液样本离心处理及储存方法
血液样本收集于10 mL EDTA-K2抗凝管中,在2~8 ℃条件下保存。为减少血细胞或细胞碎片污染,3 h内进行两步法离心处理。首先在4 ℃下以300×g离心20 min,分别吸取上清液和白细胞层,白细胞层标记后储存于-80℃。然后将上清液继续在4 ℃、3 200×g下离心10 min,取上清液。血浆充分混匀后分装成2~4个1 mL样品,立即存放于‒80 ℃,直至进行cfDNA或cfRNA提取。所有血浆仅冻融1次,以减少对结果的影响。
1.2.3 qPCR实验方案
对提取出的DNA或RNA样本,使用96孔板在 CFX Opus96 实时PCR检测系统上进行qPCR检测。10 μL qPCR反应体系由5 μL酶、4 μmol/L 上下游引物各1 μL、2 μL ddH2O、1 μL模板组成。qPCR运行条件:95 ℃ 3 min,95 ℃ 10 s,60 ℃ 30 s,60个循环。
1.2.4 标准曲线的建立
根据血液DNA不同的特征需求,在LOC105374975基因rs199032位点附近设计2组特异性扩增子,扩增片段长度分别为103碱基对(base pair,bp)和71 bp。引物序列见表1。特异性扩增子均由生工生物工程(上海)股份有限公司合成。
表1 qPCR引物序列
Tab 1
| Name | Forward (5'→3') | Reverse (5'→3') |
|---|---|---|
| Amplicon 1 | GCTCTTCCTCTCACATCTTTATTTAACC | TCCACACTGCAGTGTGAACAG |
| Amplicon 2 | GCTCTTCCTCTCACATCTTTATTTAACC | AGAAGTAATCAGATTCTGGAAGAGCTG |
| Amplicon 3 | GAAGGTGAAGGTCGGACAAC | CGAGTTAAAAGCAGCCTGGT |
自HeLa细胞系提取gDNA,经Nano-300微量分光光度计定量,浓度为12.7 ng/μL。通过梯度稀释得到浓度分别为1.27、0.127 ng/μL的gDNA样本:取20 μL原DNA溶液,加入180 μL 1×TE(Tris-EDTA缓冲液)稀释,得到1.27 ng/μL的样品1;再取20 μL样品1溶液,加入180 μL 1×TE稀释,得到0.127 ng/μL的样品2。
分别将3种浓度的DNA样品与2组特异性扩增子和PCR扩增预混液混合,进行qPCR扩增反应,得到3个样品的循环数阈值(cycle threshold,CT)。利用Origin软件对CT值与浓度的对数值进行线性拟合,得到标准曲线方程。
在后续定量分析中,基于标准曲线的线性拟合结果,通过以下公式将DNA浓度(ng/μL)转换为每毫升样品含有的拷贝数:
式中,阿伏伽德罗常数=6.022×1023;片段大小单位为bp。
1.2.5 gDNA的提取与定量
从血液中分离出白细胞,采用Kit A(柱法)和EX3600 Plus核酸提取仪提取gDNA。2种方法使用相同的血液样本。根据试剂盒使用说明书,对200 μL 白细胞样本进行gDNA提取,采用两步洗脱法(分别使用50、40 μL洗脱液)优化DNA回收效率。核酸提取仪按照“ZJ-Blood gDNA”程序提取gDNA,并收集洗脱的核酸溶液。使用qPCR方法对提取的DNA进行定量,选取扩增子1进行扩增,扩增体系与条件同“1.2.3”。
1.2.6 cfDNA的提取与定量
使用Kit B(柱法)、Kit C(磁珠法)和Kit D(磁珠法)提取血浆样本中的cfDNA,操作按照各试剂盒说明书进行。洗脱时采用两步法(分别使用30 μL和20 μL洗脱液),以最大化回收DNA。通过qPCR对提取的DNA进行定量,选取扩增子1进行qPCR扩增,扩增体系和条件与“1.2.3”相同。
1.2.7 cfDNA的提取优化与定量
对Kit B提取方法进行优化,包括增大血浆初始上样体积、减少洗脱液体积、采用2次洗脱(分别使用30、20 μL洗脱液)并延长洗脱孵育时间。选取不同上样体积的样本进行对比:样本11上样体积0.3、0.9和1.8 mL,样本12和13上样体积分别为1.5和2.1 mL,洗脱孵育时间从试剂盒默认的2 min延长至10 min。所有提取均使用同一管血液样本,确保结果可比。采用qPCR定量提取的DNA,扩增子、扩增体系和条件同“1.2.3”。
此外,评估不同存放时间对血浆cfDNA提取效率的影响。利用样本32和样本33,比较以下3种状态下cfDNA提取量:初次提取、在‒80 ℃下冻存1个月,以及在-80 ℃下冻存6个月。在整个实验过程中,样本均为同一批次血浆分管存放,未进行反复冻融处理。
1.2.8 cfRNA提取与定量
采用Kit E(柱法)和Kit F(磁珠法)对血浆样本进行cfRNA提取。每例血浆样本500 μL,均分为2份,分别采用2种方法进行提取。根据说明书微调操作,洗脱时采用两步法(分别使用30、20 μL洗脱液),以最大化回收并浓缩RNA。提取cfRNA后使用SuperScript Ⅳ VILO Master Mix即时反转录为第一链cDNA。以该混合物为模板合成第二链cDNA,加入RNase处理以去除残留RNA。纯化双链cDNA后,以0.1×TE溶液洗脱。通过qPCR检测甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase,GAPDH)的CT值。据CT=18设定cDNA浓度为100 ng/μL。采用qPCR对提取的DNA定量,并选用特定扩增子3对血浆DNA样本进行扩增。扩增子引物信息见表1。扩增体系与条件同“1.2.3”。
1.3 统计学方法
数据分析使用Origin和R软件(版本4.2.1)。对于符合正态分布且方差齐性的数据,以均值表示,采用t检验或单因素方差分析(ANOVA)进行比较;对于偏态分布的数据,以中位数表示,使用单边检验进行比较分析。P<0.05表示差异具有统计学意义。
2 结果
2.1 标准曲线的建立结果
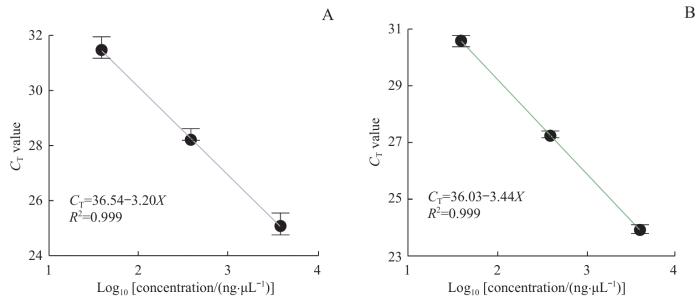
cfDNA提取物浓度通常较低,这使得使用紫外光谱或荧光光谱等方法进行定量分析面临挑战[20]。基于qPCR检测通常用于cfDNA的测定[21],在定量提取DNA时,利用qPCR检测获得的CT值,建立标准曲线并对DNA含量进行定量。使用已知浓度的样品可以为标准曲线的建立提供准确的参考,有助于校正仪器和试剂的误差,确保测量过程中DNA浓度与信号强度之间的关系保持线性。这不仅能验证样品浓度的有效性,还能确保结果的可比性和重复性。通过内部质控,可以监测实验质量,确保每次实验的可靠性和一致性。如图1所示,建立的标准曲线显示不同浓度与CT值之间具有良好的线性关系(R²≥0.999),因此可根据该标准曲线对样本DNA进行定量分析。
图1
图1
基于qPCR技术制作的2组标准曲线
Note: A. Standard curve plotted based on DNA concentration and CT value after qPCR reaction using amplicon 1 with an amplified DNA fragment length of 103 bp. B. Standard curve plotted based on DNA concentration and CT value after qPCR reaction using amplicon 2 with an amplified DNA fragment length of 71 bp. The correlation coefficients (R²) for the standard curves are close to 1 (R²≥0.999), indicating a strong linear relationship between logarithmic value of sample concentration (ng/μL) and CT signal intensity.
Fig 1
Two sets of standard curves based on qPCR technology
2.2 白细胞gDNA提取效率比较
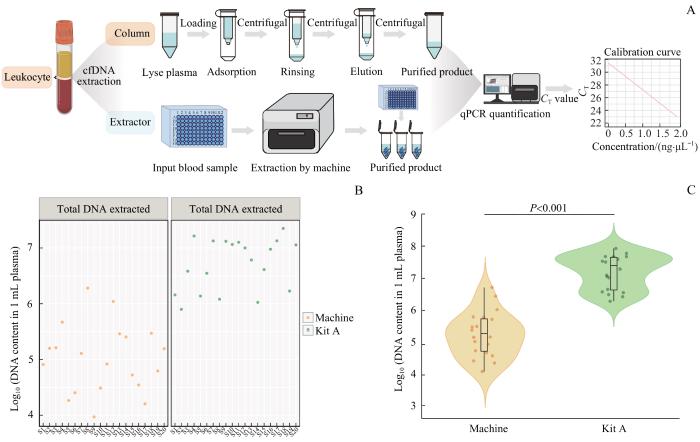
图2
图2
白细胞gDNA不同提取方法效率对比
Note: A. Schematic of leukocyte gDNA extraction methods, including extraction using Kit A and automated extraction with a nucleic acid extractor. B. Scatter plots of DNA content extracted from samples of 20 patients with Alzheimer's disease using a nucleic acid extractor and a nucleic acid extraction kit respectively. C. Comparison of gDNA extraction efficiency from samples of 20 patients with Alzheimer's disease using a nucleic acid extractor and a nucleic acid extraction kit.
Fig 2
Comparison of the efficiency of various methods for extracting leukocyte gDNA
表2 白细胞gDNA提取试剂盒及核酸提取仪的基本特征
Tab 2
| Name | Extraction method | Time-consumin/min | Sample type | Applicable volume/μL | Elution volume/μL | Indicative price for each reaction/yuan |
|---|---|---|---|---|---|---|
| Kit A | Adsorption column | 50 | Tissue and cell | 100‒200 | 50‒100 | 25 |
| EX3600 plus automated nucleic acid extractor | Magnetic bead | 40 | Whole blood, serum, and plasma | 300 | 50‒100 | 65 |
2.3 血浆cfDNA提取效率比较及条件优化
2.3.1 血浆中cfDNA提取效率比较
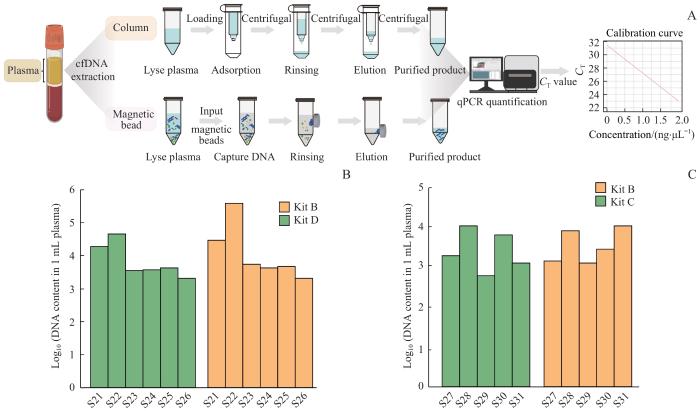
图3
图3
3种cfDNA提取方法比较
Note: A. Schematic of two plasma DNA extraction methods: the magnetic bead-based methods (Kit C and Kit D) and the column-based method (Kit B). B. Comparison of DNA content extracted from 1 mL of plasma using Kit B and Kit D respectively. Plasma samples were obtained from 6 patients with colorectal cancer. C. Comparison of DNA content extracted from 1 mL of plasma using Kit B and Kit C respectively. Plasma samples were obtained from 5 fibrosis patients.
Fig 3
Comparison of three cfDNA extraction methods
表3 血浆cfDNA提取试剂盒基本特征
Tab 3
| Name | Extraction method | Time-consumin/min | Sample type | Applicable volume/μL | Elution volume/μL | Indicative price for each reaction/yuan |
|---|---|---|---|---|---|---|
| Kit B | Adsorption column | 60 | Plasma, serum, and cell-free body fluid | 100‒200 | 50‒100 | 60 |
| Kit C | Magnetic bead | 70 | Serum, plasma, and cell-free body fluid | 200‒2 000 | 30‒50 | 30 |
| Kit D | Magnetic bead | 50 | Plasma, serum, urine, pleural fluid, cerebrospinal fluid, and saliva | 100‒5 000 | 50‒100 | 30 |
结果显示,3种试剂盒在提取cfDNA的总量上表现出相当的效能,表明它们均能有效地从血浆中富集cfDNA。但Kit B提取的cfDNA量明显高于Kit C和Kit D。具体而言,Kit B提取的DNA含量平均值分别是Kit D和Kit C的4.24倍(每毫升19 022拷贝数 vs 每毫升4 483拷贝数)和1.18倍(每毫升1 690拷贝数 vs 每毫升1 431拷贝数)(图3B、C)。尤其在低cfDNA含量样本(如样本S29、S31)中,柱法(Kit B)提取的cfDNA量高于磁珠法(Kit C),表明柱法在低浓度样本提取中具有更高的提取效率。
2.3.2 血浆中cfDNA提取优化
3种cfDNA提取试剂盒的性能比较结果表明,Kit B在cfDNA富集效率方面表现最佳。为进一步提高提取量,选择Kit B通过以下措施优化提取流程,包括增大血浆上样体积、减少洗脱液体积和延长洗脱孵育时间(图4A)。在优化后的流程中,选取3例样本,通过扩增子2进行qPCR定量,评估每微升和每毫升血浆中的cfDNA含量。
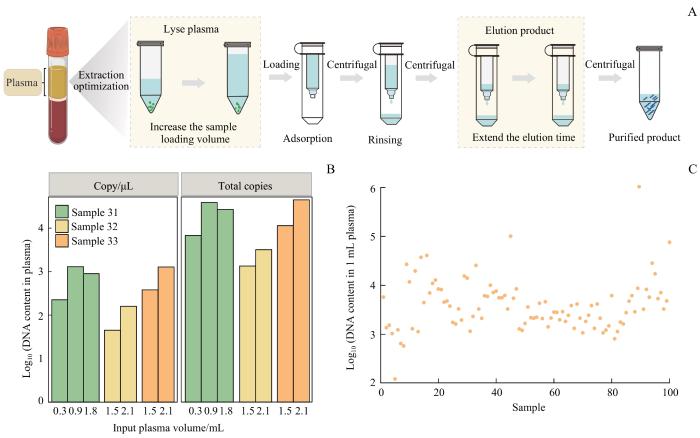
图4
图4
血浆cfDNA提取流程优化及100例结直肠癌样本血浆cfDNA提取
Note: A. Schematic of the optimized cfDNA extraction workflow from plasma by increasing the sample loading volume and extending the elution incubation time. B. Analysis of the results of cfDNA extraction using Kit B and optimized extraction process for sample 31 (initial input volumes: 0.3, 0.9, and 1.8 mL) from fibrosis patient and samples 32 and 33 (initial input volumes: 1.5 and 2.1 mL) from colorectal cancer patients. C. Plasma cfDNA content extracted from 100 colorectal cancer patient samples using Kit B.
Fig 4
Optimization of plasma cfDNA extraction process and plasma cfDNA extraction from 100 colorectal cancer samples
图4B展示了不同上样体积下提取的cfDNA量。对比3例样本在不同上样体积下的cfDNA定量结果,发现血浆大上样体积时的cfDNA提取量分别是小上样体积时的3.98倍(每毫升27 087拷贝数vs每毫升6 800个拷贝数)、2.38倍(每毫升3 209个拷贝数vs每毫升1 350个拷贝数)和3.82倍(每毫升44 942个拷贝数 vs 每毫升11 460个拷贝数)。结果表明,增大血浆初始上样体积及延长洗脱孵育时间能够提高血浆cfDNA的提取量。
在确定了提取样本的试剂盒及操作方法后,采用该方案提取100例结直肠癌患者血浆中的cfDNA。结果显示每毫升血浆的cfDNA拷贝数中位数为3 716个(图4C)。提示该提取方案在临床样本中的适用性良好,能够稳定提取到足够量的cfDNA。
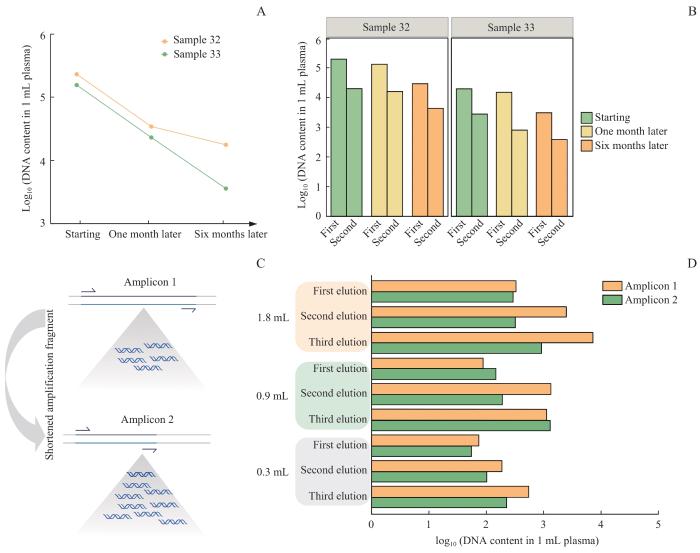
2.3.3 存放时间及扩增子长度对血浆cfDNA含量的影响
已有研究[22]表明,不同的存储条件对血浆中cfDNA含量会造成影响。为此我们探究了血液样本采集后存放时间对血浆cfDNA含量的影响。对2例结直肠癌样本不同存放时间的血浆进行cfDNA提取实验。所有样本均使用Kit B进行cfDNA提取。通过扩增子1进行qPCR定量,评估不同存放时间下每毫升血浆中cfDNA的产量。结果显示,冻存6个月后的样本cfDNA含量分别降低至新鲜样本的35.4%(每毫升69 150个拷贝数 vs 195 183个拷贝数)和15.6%(每毫升3 052个拷贝数vs 19 561个拷贝数),表明血浆cfDNA含量随着存放时间延长而降低(图5A)。并且,无论是第一次洗脱还是第二次洗脱,DNA的含量均呈现相似的下降趋势(图5B)。此外,cfDNA的典型大小分布表现为主峰在166 bp,且存在以167 bp(85.0%)为主峰、50~153 bp范围内呈10 bp周期振荡的峰值。对于低于100 bp的片段,常规长探针的捕获效率较低[23]。为了评估短扩增子与长扩增子在捕获cfDNA分子数量上的差异,设计2组扩增子(扩增子1和2)(图5C),并通过qPCR定量分析了样本31中血浆的cfDNA产量。图5D显示了2组扩增子对cfDNA分子捕获的差异:短扩增子捕获的平均分子数是长扩增子的2.39倍(每毫升541个拷贝数 vs 每毫升226个拷贝数)。结果表明,短扩增片段相比长扩增片段显著提高了cfDNA的捕获效率,尤其在处理片段化cfDNA时表现出明显优势。
图5
图5
血浆中cfDNA受存放时间影响及扩增子长度对DNA分子捕获差异的比较
Note:A. cfDNA content in the first elution of colorectal cancer patient samples 32 and 33 at three time points (30 μL elution). B. cfDNA content in both the first and second elutions of colorectal cancer patient samples 32 and 33 at three time points (20 μL elution for the second). C. Schematic of amplicon design: amplicon 1 (103 bp) and amplicon 2 (71 bp). D. qPCR quantification of cfDNA from fibrosis patient sample 31 using the two amplicons (30 μL for the first elution, 20 μL for the second and third).
Fig 5
Comparison of the impact of storage time on plasma cfDNA and the difference in DNA molecule capture based on primer length
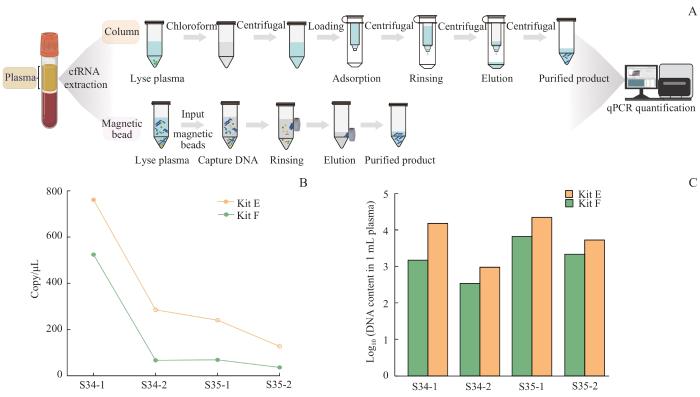
2.4 血浆cfRNA提取效率比较
血浆中cfRNA代表细胞外转录本的动态情况,在生殖系统疾病、肿瘤和神经退行性疾病的预测、诊断和监测中展现了广泛的应用前景[24]。因此我们也探究了血浆cfRNA的提取方法。对2种cfRNA提取试剂盒Kit E和Kit F的性能进行比较,分析采用吸附柱法与磁珠法的提取效果(图6A)。2种试剂盒基本特征信息见表4。Kit E相较于Kit F更经济且操作时间更短。我们选取2例健康个体血浆样本,使用2种试剂盒进行提取,并对其效率进行对比。通过对GAPDH基因的qPCR定量,测定血浆中提取出的cfRNA含量。结果(图6B)显示,柱法提取的cfRNA含量中位数是磁珠法提取的cfRNA含量的5.01倍(每毫升29 460个拷贝数 vs 每毫升5 880个拷贝数),且单边检验分析显示两者差异具有统计学意义(P=0.034)。图6C进一步比较了2种方法在每毫升血浆样本中DNA提取量,结果显示Kit E的提取效率高于Kit F。这一结果表明,柱法提取试剂盒(Kit E)在cfRNA提取效率上具有优势,能够获得更高的cfRNA产量,对于临床应用中cfRNA的检测和监测具有重要价值。
图6
图6
血浆中cfRNA提取效率对比
Note: A. Schematic comparison of cfRNA extraction methods using column-based and magnetic bead-based techniques. Samples 34-1 and 35-1 represent the first 30 μL elutions, while samples 34-2 and 35-2 represent the second 20 μL elutions. B. Comparison of cfDNA copy number (per microliter of plasma) extracted from the blood plasma of two normal individuals using Kit E and Kit F. The line chart shows that the column-based method is more efficient in extraction. C. Comparison of the log copy numbers of cfRNA extracted using Kit E and Kit F from 1 mL of blood plasma from two normal individuals, with two elutions of DNA content.
Fig 6
Comparison of cfRNA extraction efficiency from plasma
表4 血浆cfRNA提取试剂盒基本特征
Tab 4
| Name | Extraction method | Time-consuming/min | Sample type | Applicable volume/μL | Elution volume/μL | Indicative price for each time/ yuan |
|---|---|---|---|---|---|---|
| Kit E | Adsorption column | 60 | Blood, plasma, serum, and lymph | 250 | 30‒50 | 54 |
| Kit F | Magnetic bead | 70 | Plasma and serum | 200‒1 000 | 30‒50 | 70 |
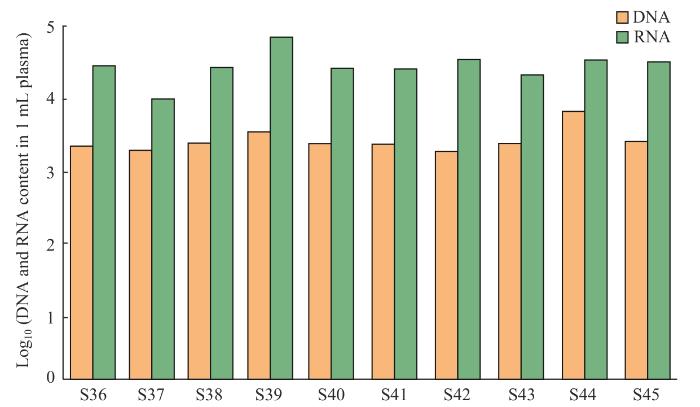
2.5 血浆中cfDNA与cfRNA含量对比
cfRNA相比cfDNA具有潜在优势,因其含量较高且能够提供更为丰富的肿瘤基因表达信息;尤其在cfDNA量不足时,cfRNA可弥补这一不足,有助于预测癌症患者的治疗反应[25]。为探究血浆样本中cfDNA与cfRNA的含量差异,对10例健康个体血浆样本进行系统的cfDNA与cfRNA提取实验。cfDNA和cfRNA分别使用试剂盒Kit B和Kit E提取,最终洗脱体积均为30 µL。比较同一血浆样本中cfDNA与cfRNA的含量,并通过qPCR定量以量化其差异。结果(图7)显示,在10例样本中,血浆cfRNA的平均含量是cfDNA的27.65倍(每毫升6 500个拷贝数 vs 每毫升179 757个拷贝数)。单因素方差分析(ANOVA)结果显示,cfDNA与cfRNA的含量差异具有统计学意义(P<0.001)。通过比较10例健康个体血浆样本中的cfDNA与cfRNA含量,我们发现cfRNA含量显著高于cfDNA,突出了cfRNA作为疾病生物标志物的潜力。
图7
图7
10例样本血浆中cfDNA与cfRNA含量对比
Fig 7
Comparison of cfDNA and cfRNA content in plasma from 10 samples
3 讨论
本研究对比试剂盒法和核酸提取仪法在白细胞gDNA提取中的表现,发现核酸提取仪的操作时间较短,但试剂盒法在效率上表现更优,尤其适用于高产量的研究需求。Kit A因其提取所需费用较低,表现出较好的经济效益。此外,该试剂盒不仅适用于细胞和组织DNA的提取,还可用于血浆cfDNA的提取,展现出广泛的应用潜力。因此,Kit A有望成为一种全能提取试剂盒,在临床诊断和研究中具有较好的应用前景。然而,其临床推广仍需克服一些挑战,例如提取效率的稳定性和大规模应用中人工提取操作时间过长等问题,这些都有待于在实际应用中进一步验证和优化。
对于cfDNA的提取,3种试剂盒对比结果表明,其表现相当。但试剂盒Kit B在处理低浓度cfDNA样本时展现出略高的提取效率,并且通过优化提取流程(如增加上样量、减少洗脱液体积)可显著提升其提取效率。然而,Kit C和D在操作简便性和成本方面则更有优势。在临床应用中,需要综合考虑提取效率、操作流程、成本等因素。并且,柱法的应用可能受到处理时间和试剂消耗的限制,因此在实际应用中需要平衡这些挑战并进行相应的优化。此外,研究还揭示了样本存储条件对cfDNA稳定性的影响,这一结果强调了样本存储条件对cfDNA分析结果影响较大,提示在实际操作中应尽量缩短样本处理和分析的时间,以减少cfDNA含量的损失。鉴于cfDNA的片段化特性[6,26],我们优化了cfDNA的捕获策略,发现相比于长扩增子,短扩增子能够更有效地捕获cfDNA分子。这一改进有助于提高后续分析的灵敏度和准确性,从而增强检测结果的可靠性。这一发现为cfDNA扩增子设计提供了新思路,有助于在高度片段化的样本中提升分析性能。
血浆循环cfRNA因其具有组织、癌症亚型以及来源细胞类型特异性,成为肿瘤诊断、监测及个性化医疗的潜在工具[11,18]。我们对比了柱法和磁珠法在cfRNA提取中的性能,发现柱法不仅提取效率高,且操作简洁、成本低廉,成为经济高效的提取方案。然而柱法提取过程中使用的有机溶剂三氯甲烷具有潜在的毒性,因此在长期或大规模应用时必须特别关注其安全性。为降低这一风险,可以通过选择更安全的三氯甲烷替代物、优化操作流程以降低溶剂的使用量和缩短操作时间来减少毒性暴露。此外,加强实验室的防护措施,采用自动化提取系统和封闭式操作流程,也能有效减少人工操作带来的风险。通过这些措施,能够有效降低有机溶剂的毒性风险,从而保障实验的安全性和准确性。相较于DNA,RNA分子在表达过程中可大量转录,尤其是肿瘤特异性转录本的过表达,显著增强血浆中肿瘤衍生的RNA信号[14,27]。本研究结果也表明,血浆中cfRNA的丰度远高于cfDNA,提取的cfRNA平均分子量是cfDNA的27.65倍,进一步凸显了cfRNA作为疾病生物标志物的巨大潜力[28]。
综上所述,本研究不仅系统性地对比了多种DNA提取方法的效率差异,还进一步对cfDNA的提取流程进行了优化;不仅为血液样本核酸提取提供了新视角,还突出了方法优化与选择的重要性。特别是cfRNA在血浆中的高丰度,预示着其作为疾病生物标志物的巨大潜力,为癌症等相关疾病的早期诊断、监测及治疗评估开辟了新途径。
AUTHOR's CONTRIBUTIONS
HAN Da and SONG Ping were responsible for the design of the project and experiments, and participated in the writing and revision of the paper. LU Ping, PENG Junjie and WANG Zimin contributed to sample collection. SU Xinglei was in charge of experimental operations, data analysis, figure preparation, and manuscript writing. All authors have read the final version of paper and consented to its submission.
作者贡献声明
韩达、宋萍负责课题和实验的设计并参与论文的写作与修改,路平、彭俊杰和汪滋民参与样本收集,宿星蕾负责实验操作、数据分析、作图及撰写文章。所有作者均阅读并同意了最终稿件的提交。
利益冲突声明
所有作者声明不存在利益冲突。
COMPETING INTERESTS
The authors declare no conflicts of interest.
参考文献