多细胞动物的进化和发育过程具有许多共同的特征。2个过程都起始于一个真核单细胞:单细胞原生动物或受精卵。在进化和发育过程中产生特化的细胞类型,并由它们开始形成肌肉、神经、血管、肠道和骨骼等多种组织[1]。在基因编码区,发育过程中表达的基因与进化之间有很强的相关性:在发育早期阶段,不同物种胚胎的转录谱较为相似;而在发育成熟后,器官更多地表达物种特异性基因[2]。一般来说,在生长发育中起关键作用的编码基因,例如同源异型盒(homeobox,HOX)、配对盒(paired box,PAX)、骨形态发生因子(bone morphogenetic protein,BMP)等编码重要的转录因子及外源性形态发生因子的基因家族[3],具有很强的保守性。它们往往参与调控细胞生长、周期以及早期胚胎体轴决定等重要生命活动,并在不同物种中具有非常高的同源性。而一些参与特殊生理过程或者调控蛋白活性的编码基因,例如参与神经活动、免疫调节、生殖以及蛋白质修饰的基因,其保守性相对较弱。另一方面,基因的表达模式由调控序列决定,因此这些非编码区的调控序列也存在相应的保守性差异。保守性调控元件起源于脊椎动物进化早期(哺乳类与鸟类、爬行类出现分离时),其所调控的基因多为参与胚胎结构形成的转录调控因子[4]。此后进化中出现的新调控区与细胞外信号传递的基因相关;最后出现的调控区与蛋白翻译后修饰的基因相关[4]。
非编码调控区决定了基因的表达模式,而增强子元件在该过程中发挥了关键调控作用。增强子是长度多为几百个碱基对的DNA片段,通常被多种转录因子结合并占据。H3组蛋白第27位赖氨酸乙酰化(histone H3 acetylated at lysine 27,H3K27ac)是基因组上活性增强子的标志,可以通过H3K27ac染色质免疫共沉淀测序(chromatin immunoprecipitation sequencing,ChIP-seq)数据鉴定的峰值定义增强子。这些增强子能控制细胞类型特异性的基因表达模式[5-7]。2013年,WHYTE等[8]提出了超级增强子(super enhancer,SE)的概念,其他研究者对其不断进行补充[9-10]。超级增强子是基因组中短间距的增强子簇,由基因启动子上游或下游附近的一大簇活性增强子组成,在基因组上占据超过12.5 kb的区域,具有开放的染色质并富集较多的转录共激活因子和核心转录因子。ROSE程序可以将相距不超过12.5 kb的增强子串联在一起得到所有增强子簇;H3K27ac信号富集程度最大的增强子簇即为超级增强子[8]。超级增强子存在于各种类型的细胞中,并在人类、小鼠等多个物种中被检测到[11]。此外,超级增强子上也富集了与结直肠癌、自身免疫性疾病、冠状动脉疾病和糖尿病等多种疾病相关的单核苷酸多态性(single nucleotide polymorphism,SNP)序列[12-15]。针对脊椎动物的研究[16]表明,普通增强子(typical enhancer,TE)和超级增强子的序列保守性比其附近的基因组区域更高。此外,研究[16]还发现,在斑马鱼大多数组织超级增强子序列的保守性明显高于普通增强子。但是,小鼠和人类中两者序列保守性的差异仍不明确,其可能取决于具体分析的组织类型[16]。
本研究拟在基因编码区建立一种量化基因进化程度的方法,并且在广泛的发育过程中验证其评估组织和细胞发育潜能的能力。而针对非编码调控区,从发育全局的角度出发研究不同发育阶段的组织中增强子和超级增强子的保守性,以此探究非编码调控区的进化与发育的关系。
1 资料与方法
1.1 数据获取
从高通量基因表达数据库(Gene Expression Omnibus,GEO)(
1.2 基因编码区保守率计算
从UCSC genome browser(
1.3 ChIP-seq数据分析和增强子保守性评分计算
对于增强子表观遗传标志物H3K27ac的ChIP-seq数据,用Trim_galore(version 3.4)程序过滤掉低质量的读数(reads),并通过FastQC程序查看质量控制结果。通过bowtie2(version 2.4.2)程序将reads比对到hg19参考基因组。使用samtools(version 1.9)程序对获得的二进制sam文件进行处理,包括排序和建立索引等步骤。利用MACS2(version 2.2.7.1)程序的“--broad”参数寻找峰值(peak),并将peak区间的序列定义为增强子,截断值(cutoff)设为0.1。
使用bedtools(version 2.30.0)程序的“intersect”功能进行增强子数据组间的差异增强子比较。根据重叠情况,将重叠超过2/3的增强子定义为重复的增强子,而其他增强子则为特异性增强子。使用PhastCons程序计算组织特异性增强子的DNA保守性评分(conservation score,CS)。
1.4 RNA-seq数据分析
对原始SRA格式进行格式转换、质量控制(去除低质量的reads和接头序列)、比对和计数等处理,具体步骤如下。使用Trim_galore(version 3.4)程序过滤掉低质量的reads,并使用FastQC程序查看质量控制结果。采用hisat2(version 2.2.1)程序将reads比对到hg19参考基因组。使用samtools(version 1.9)程序处理获得的二进制sam文件,包括排序和建立索引等步骤。为了获取总体RNA表达矩阵、mRNA表达矩阵和lncRNA表达矩阵,使用featureCounts(version 2.0.1)程序对比对后的片段进行计数和注释。采用DESeq2(1.20.0)R包进行基因转录组表达数据组间的差异基因比较,并选取差异倍数(fold change)大于2和调整后P值小于0.05作为差异基因的筛选标准。
1.5 超级增强子和关联基因鉴定
1.6 超级增强子相关基因的功能富集、信号通路分析
为探究超级增强子相关基因可能参与的信号通路,利用cytoscape软件中的clueGO拓展功能对相关基因进行基因本体数据库(gene ontology,GO)功能分析,包括生物学过程(biological process,BP)和京都基因与基因组百科全书(Kyoto encyclopedia of genes and genome,KEGG)通路分析。将P<0.05设定为富集通路的入选标准。
1.7 统计学分析
应用Graphpad Prism 8.0软件进行数据统计分析及作图。定量资料用x±s表示;2组间比较采用独立样本t检验,多组间比较采用单因素方差分析。P<0.05表示差异具有统计学意义。
2 结果
2.1 建立基因保守性的量化指标——保守率
以人类基因组hg19为参考,对UCSC基因组浏览器中的100个物种基因组进行比对;基于基因编码的氨基酸的变化,计算每个基因的保守率。可见,保守基因微管蛋白α 1a基因(tubulin alpha 1a,TUBA1A)编码氨基酸的保守率明显高于人类特异性新基因Rho三磷酸鸟苷酶激活蛋白11B(Rho GTPase activating protein 11B,ARHGAP11B)编码氨基酸的保守率(图1A)。根据保守率对基因排序后,按照每
图1
图1
基因保守率的计算及将基因分配到189个集合的流程
Note: A. Calculation of CR and bins of genes. B. GO analysis of gene bins on aging and embryonic stem term. Vertical axis—calculation of percentage of Bin′s genes in terms. CR—conservation rate.
Fig 1
Process of calculating genes′ CR and assigning genes to 189 bins
100个基因为一个区间,将18 883个基因分配到189个基因集合(bin)中。从1号到189号,bin的基因保守率逐渐上升。为了研究基因保守率与发育和衰老关系,选择以下2个通路进行分析:干细胞通路(Embryonic stem)包含胚胎干细胞相关基因,衰老通路(Aging)包含随着年龄增长而表达增加的基因。通路富集分析的结果(图1B)显示,保守率越高的bin,其包含的基因越富集在干细胞通路;而保守率与衰老通路无相关性。这表明保守率与发育潜能相关而与衰老无关。
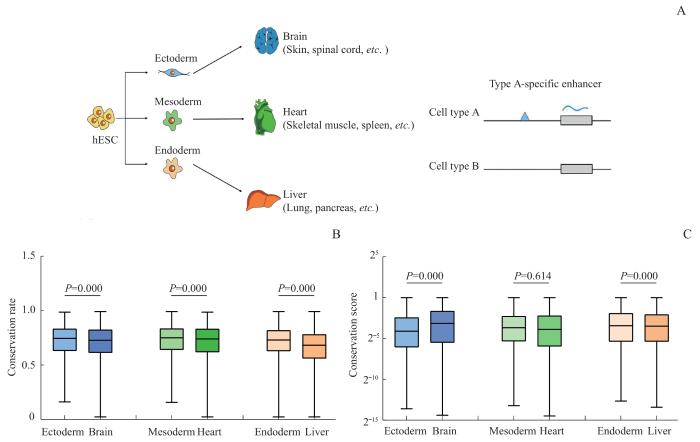
2.2 三胚层发育有关的基因和增强子区域保守性的分析
图2
图2
发育过程细胞类型特异性基因和增强子的保守性分析
Note: A. Schematic diagram of germ layer specification and organ development. hESC—human embryonic stem cell. B. CR of cell type-specific genes. C. CS of cell type-specific enhancers.
Fig 2
Conservation analysis of cell type-specific genes and enhancers in development
另外,使用GTEx数据库中3个胚层发育的其他器官组织(皮肤、脊髓、脾、骨骼肌、胰腺和肺)的基因表达数据,与GEO数据库下载分析的三胚层和器官(内胚层、中胚层、外胚层、肝脏、心脏和大脑)基因表达数据比较,选取表达差异2倍以上且P<0.05的基因进行保守率评分(表1)。所有早期胚层特异性表达的基因的保守率均高于对应的成熟组。该结果表明,本研究建立的保守率可以区分发育前后的细胞和组织:保守率越高的细胞和组织,其发育潜能越强。
表1 多种器官发育前后特异表达基因的保守率统计
Tab 1
| CR | Ectoderm development | Mesoderm development | Endoderm development | ||||||
|---|---|---|---|---|---|---|---|---|---|
| Brain | Skin | Spinal cord | Heart | Skeletal muscle | Spleen | Liver | Lung | Pancreas | |
| Progenitor-specific | 0.72 | 0.72 | 0.73 | 0.72 | 0.74 | 0.75 | 0.71 | 0.74 | 0.75 |
| Organ-specific | 0.70 | 0.69 | 0.66 | 0.71 | 0.72 | 0.65 | 0.66 | 0.65 | 0.68 |
| P value | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 |
| Fold change | 1.02 | 1.05 | 1.10 | 1.02 | 1.02 | 1.16 | 1.08 | 1.14 | 1.11 |
人类基因组DNA上非编码区占据了比编码区更多的区域。增强子元件在非编码区发挥了关键调控作用。在确定DNA编码区的保守性可以评估细胞和组织发育潜能后,接下来我们继续探究非编码区增强子的保守性与发育潜能的关系。组蛋白H3K27ac是基因组上活跃增强子的标志。我们下载并分析了H3K27ac的ChIP-seq数据,以其在基因组上的结合位置定义为增强子。选取器官和对应胚层不重叠的增强子部分定义为各自状态的差异增强子(图2A)。然而,针对差异性增强子的DNA序列,根据PhastCons程序计算的保守性评分与发育阶段并无一致性的相关趋势(图2C、表2)。例如,中胚层发育的器官(心脏)的增强子的保守性评分无显著差异;内胚层发育的器官(肝脏)的保守性评分随着器官成熟下降;外胚层发育的器官(大脑)的保守性评分随着器官成熟反而上升。这一结果在其他器官的分析中也有所体现(表2)。针对该情况,我们对增强子类别中的重要组分进行深入分析,重点探索超级增强子的保守性与发育的相关性。
表2 多种器官发育前后特异增强子的保守性评分统计
Tab 2
| CS | Ectoderm development | Mesoderm development | Endoderm development | ||||||
|---|---|---|---|---|---|---|---|---|---|
| Brain | Skin | Spinal cord | Heart | Skeletal muscle | Spleen | Liver | Lung | Pancreas | |
| Progenitor-specific | 0.15 | 0.15 | 0.14 | 0.15 | 0.15 | 0.15 | 0.18 | 0.18 | 0.18 |
| Organ-specific | 0.20 | 0.11 | 0.16 | 0.15 | 0.12 | 0.11 | 0.17 | 0.11 | 0.12 |
| P value | 0.000 | 0.000 | 0.000 | 0.614 | 0.001 | 0.000 | 0.000 | 0.000 | 0.000 |
| Fold change | 0.72 | 1.33 | 0.87 | 1.00 | 1.27 | 1.37 | 1.10 | 1.66 | 1.54 |
2.3 三胚层及对应的组织器官的超级增强子的鉴定
增强子在基因组上的序列数量远远多于基因的数量,其中发挥关键调节作用的部分尚不确定。超级增强子是增强子信号尤其富集的增强子簇,其调控与细胞身份最为相关的基因,能够促进这些基因的大量表达。因此,我们主要关注超级增强子特有的DNA序列进化与发育的关系。利用ROSE程序对H3K27ac的信号排序来鉴定超级增强子(图3A)。共鉴定了9个器官和3个胚层的超级增强子。组成超级增强子的增强子称为超级增强子元件,除了超级增强子元件的其他增强子称为普通增强子。具体超级增强子、超级增强子元件、普通增强子的数量分布如表3所示。3个胚层及其代表性器官(大脑、心脏、肝脏)的超级增强子和普通增强子都在其对应的细胞类型中富集H3K27ac信号(图3B、C)。此外,通过对成熟器官超级增强子附近100 kb内的基因进行功能分析,发现基因富集在与之相应的生物学功能:例如大脑、心脏和肝脏的超级增强子调控的基因分别富集在神经形成、心脏发育和营养代谢等通路(图3D)。这表明本研究鉴定出的超级增强子图谱与细胞类型具有高度相关性。
图3
图3
早期三胚层向相应器官发育过程中细胞特异性超级增强子的鉴定
Note: A. The procedure of SE identifications. B. ChIP-seq signal of H3K27ac in TE regions. C. ChIP-seq signal of H3K27ac in SE regions. D. Biological process of SE-related gene. SE—super enhancer; TE—typical enhancer.
Fig 3
Identification of SEs during the development of three early germ layers to corresponding organs
表3 三胚层和相应多种器官中超级增强子、超级增强子元件和普通增强子的数量
Tab 3
| Tissue type | SE/n | SE element/n | TE/n |
|---|---|---|---|
| Ectoderm | 839 | 5 078 | 74 570 |
| Brain | 825 | 5 654 | 50 322 |
| Spinal cord | 942 | 6 362 | 67 360 |
| Skin | 1 390 | 12 679 | 73 065 |
| Mesoderm | 508 | 4 054 | 79 411 |
| Heart | 1151 | 11 050 | 72 112 |
| Spleen | 1 712 | 15 581 | 78 865 |
| Skeletal Muscle | 1 496 | 12 534 | 80 847 |
| Endoderm | 671 | 3 770 | 57 547 |
| Liver | 777 | 3 424 | 43 749 |
| Lung | 538 | 4 304 | 57 040 |
| Pancreas | 1 657 | 16 475 | 94 829 |
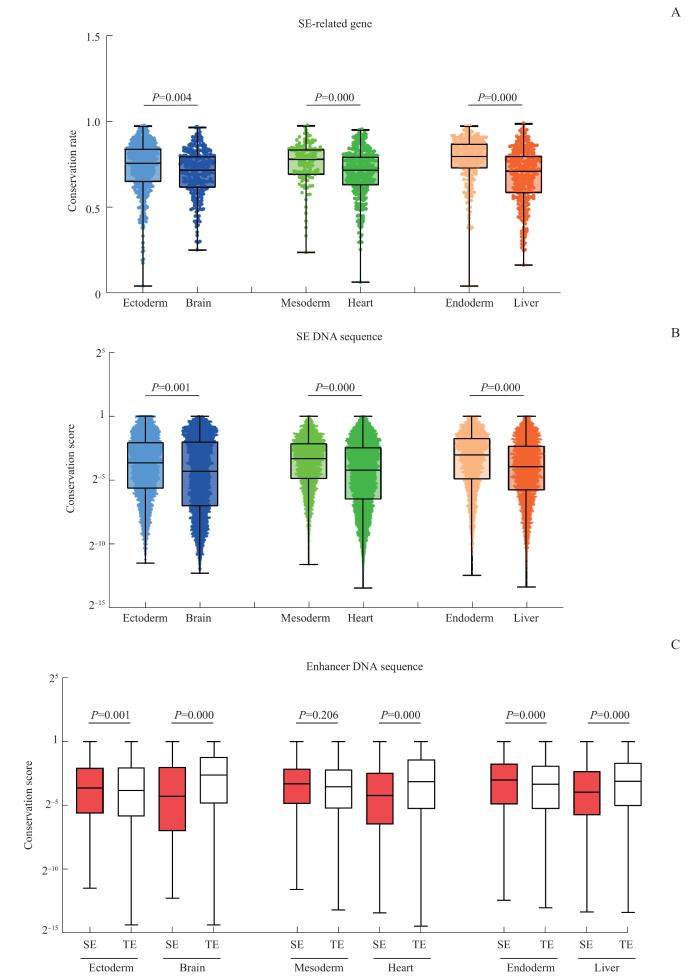
2.4 超级增强子保守性与发育潜能的关系
选取转录起始位点(transcription start site,TSS)在超级增强子附近100 kb内的基因作为其可能调控的靶基因。通过分析3个不同胚层来源器官超级增强子调控的基因保守率,发现胚层超级增强子调控的基因保守性显著高于其对应的成熟器官(图4A),这表明超级增强子调控的基因保守率也与发育具有相关性。此外,通过分析超级增强子元件的保守性评分,发现胚层超级增强子元件本身DNA序列的保守性也显著高于其对应的成熟器官(图4B、表4),这说明非编码调控区的保守性评分也可以区分发育前后的细胞或组织。最后比较了三胚层发育中超级增强子元件和普通增强子的保守性,发现在早期胚层中的超级增强子元件保守性显著高于普通增强子,而在成熟器官中则相反(图4C、表5)。推测这是因为相较于普通增强子,超级增强子与细胞谱系更相关:超级增强子在早期胚层中与高发育潜能的细胞身份相关,调控高保守性的基因,序列也在进化上更保守;在成熟器官中的超级增强子与高度特异化的细胞身份相关,调控高度特异性基因,从而体现在进化和突变水平上更加活跃。
图4
图4
超级增强子的保守性与发育潜能的相关性
Note: A. CR of SE-related genes. B. CS of SE elements. C. CS of SEs and TEs.
Fig 4
Correlation between conversation of SEs and development potential
表4 多种器官发育前后超级增强子元件的保守性评分
Tab 4
| CS | Ectoderm development | Mesoderm development | Endoderm development | ||||||
|---|---|---|---|---|---|---|---|---|---|
| Brain | Skin | Spinal cord | Heart | Skeletal muscle | Spleen | Liver | Lung | Pancreas | |
| Progenitor-specific | 0.17 | 0.17 | 0.17 | 0.16 | 0.16 | 0.16 | 0.20 | 0.20 | 0.20 |
| Organ-specific | 0.16 | 0.11 | 0.15 | 0.13 | 0.11 | 0.10 | 0.15 | 0.10 | 0.11 |
| P value | 0.001 | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 |
| Fold change | 1.09 | 1.60 | 1.14 | 1.22 | 1.42 | 1.59 | 1.37 | 1.93 | 1.75 |
表5 三胚层及相关器官超级增强子元件与普通增强子的保守性评分统计
Tab 5
| CS | Ectoderm development | Mesoderm development | Endoderm development | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Ectoderm | Brain | Skin | Spinal cord | Mesoderm | Heart | Skeletal muscle | Spleen | Endoderm | Liver | Lung | Pancreas | |
| TE | 0.06 | 0.16 | 0.06 | 0.08 | 0.08 | 0.11 | 0.07 | 0.06 | 0.10 | 0.12 | 0.05 | 0.06 |
| SE | 0.08 | 0.05 | 0.11 | 0.15 | 0.10 | 0.05 | 0.06 | 0.04 | 0.12 | 0.06 | 0.04 | 0.05 |
| P value | 0.001 | 0.000 | 0.000 | 0.000 | 0.206 | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 |
| Fold change | 1.31 | 0.31 | 0.84 | 0.84 | 1.18 | 0.47 | 0.83 | 0.72 | 1.25 | 0.55 | 0.82 | 0.83 |
Note: As the data deviated from the normal distribution, the median value was used to reflect the data and calculate the foldchange.
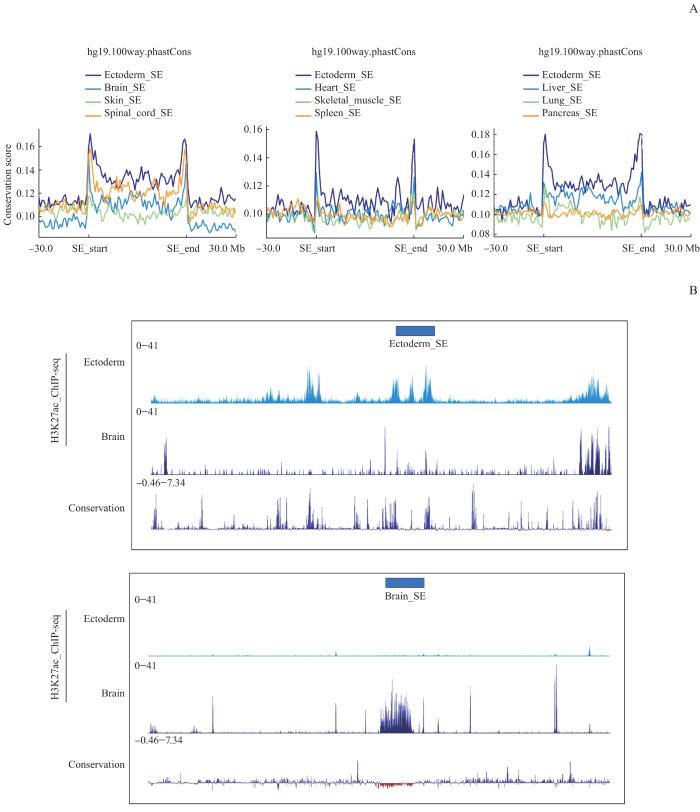
2.5 超级增强子与附近基因组区域DNA序列在进化中的保守性分析
图5
图5
超级增强子和附近区域的保守性
Note:A. CS signal of SEs and nearby regions. B. The SEs of ectoderm and brain showed on the UCSC genome browser. Vertical axis—signal of ChIP-seq or degree of conservation (UCSC).
Fig 5
Conservation of SEs and nearby regions
接下来我们比较了超级增强子元件与附近30 kb的普通增强子中所包含的高保守性增强子(保守性评分大于0.8)比例,发现胚层超级增强子元件内的保守性增强子比例显著高于其在附近普通增强子中的比例。而在对应的成熟器官中,发现部分器官(大脑、皮肤、脊髓、脾、骨骼肌和肺等)的超级增强子元件所包含的保守性增强子比例低于附近的普通增强子,其他器官(心脏、肝脏和胰腺)则呈现相反趋势(表6)。由此我们认为:发育早期的超级增强子元件倾向于比附近区域普通增强子保守,但发育后期的保守性情况取决于具体的器官类型。
表6 高保守增强子在超级增强子和附近普通增强子中的百分比
Tab 6
| Tissue type | SE/% | Nearby SE/% | Fold change |
|---|---|---|---|
| Ectoderm | 3.18 | 2.83 | 1.12 |
| Mesoderm | 1.04 | 0.93 | 1.12 |
| Endoderm | 2.32 | 2.04 | 1.14 |
| Brain | 1.67 | 1.70 | 0.98 |
| Skin | 0.61 | 0.66 | 0.93 |
| Spinal cord | 1.60 | 1.77 | 0.91 |
| Heart | 1.10 | 0.81 | 1.36 |
| Muscle | 0.42 | 0.56 | 0.75 |
| Spleen | 0.78 | 0.92 | 0.85 |
| Liver | 1.35 | 1.07 | 1.26 |
| Lung | 0.78 | 1.28 | 0.61 |
| Pancreas | 0.96 | 0.77 | 1.25 |
为了更加形象化地展示超级增强子的保守性,分别选取外胚层和大脑组织的2个超级增强子进行展示(图5B)。其分别位于人类基因组(hg19版本)的chr2:44809603~45118530和chr19:37492810~37933580区域。首先,可以明显观察到外胚层超级增强子的保守性高于发育成熟的大脑中的超级增强子。其次,外胚层超级增强子内增强子信号(H3K27ac ChIP-seq)较高的DNA序列比附近区域的序列更加保守,而对应的成熟大脑组织中并无此趋势。
2.6 DNA序列进化与发育的关系
如图6所示,本研究在3个不同胚层来源的器官发育中研究DNA序列的进化与发育的关系。发现随着发育进程的推进,编码区表达的基因的保守率下降,非编码区增强子的保守性评分无此趋势,但是超级增强子的保守性评分则显示出和基因的保守率一致的下降趋势。这表明其进化保守性和基因一样能用于评估细胞发育潜能。
图6
图6
发育潜能与DNA序列的保守性
Fig 6
Developmental potential and conservation of DNA sequence
3 讨论
科学家很早就尝试将进化过程与胚胎发育联系起来,并从形态学到分子生物学层面进行了许多相关研究;但仍然需要进一步在全局的角度量化进化的保守性并验证其与发育关系。本研究着眼于在DNA序列层面评估进化保守性与发育的关系。
在编码区,我们利用基因所对应的氨基酸序列建立保守率这一指标。结果表明:保守率高的基因与胚胎干细胞通路更相关,在胚胎发育到成熟器官中表达的特异性基因的保守率明显下降。这体现了保守率具有评估发育潜能的作用,未来有希望将其运用于对发育轨迹和单细胞数据分析的研究中。在非编码调控区,我们关注增强子区域的DNA序列的保守性。发现发育早期和晚期细胞和组织中的差异性增强子的保守性评分与发育阶段无显著的相关趋势,这可能是由于全基因组染色质区域包括太多背景信息而无法有效去除所致。超级增强子是富集增强子信号的增强子簇。超级增强子虽然仅是少部分增强子的集合,但富集了大量细胞命运相关转录因子的调控序列,从而调控与细胞身份密切相关的基因。我们分析发现,在胚胎发育早期的超级增强子的DNA序列保守性评分明显高于成熟器官的超级增强子;这提示调控区的进化与发育的相关性。
过去在基因非编码调控区的研究[18-19]表明,增强子或顺式调节元件与蛋白质编码基因不同,通常其DNA序列保守性较弱,并在哺乳动物中经历快速进化,具有很强的细胞异质性。相关研究[20]表明,胎生哺乳动物的基因组中存在3个进化保守的超级增强子,其激活与多能性相关,这提示保守调控区与细胞干性相关。我们的研究在多个胚层发育中证明了胚胎发育早期特异表达的基因和超级增强子都相对保守。以往的研究[16]发现人类普通增强子和超级增强子序列保守的差异取决于所要分析的组织类型。而我们的研究提示该差异有一定的规律:发育早期组织的超级增强子序列的保守性高于普通增强子,这与高发育潜能的组织特点密切相关;发育晚期组织的超级增强子序列的保守性低于普通增强子,其与高度特异化的组织功能相关。研究[16]同时也提示:哺乳动物保守直系同源基因的超级增强子比其他超级增强子具有更高的序列保守性。结合本研究的结果,我们认为发育早期细胞的超级增强子和其调控的基因在进化过程中保守,两者共同维持了细胞的发育潜能。晚期成熟的各种复杂器官执行特异化的功能。这些复杂功能的基因和对应调控区是由活跃变异的DNA区域进化而来,因此这些组织特异表达基因是非保守的;同时与这种细胞身份密切相关并且调控这些非保守基因的超级增强子也在进化中活跃地变异,体现序列的不保守性。研究增强子的保守性及灵活性的进化规律,一方面能够揭示早期胚胎发育以及成熟器官不同的调控模式,发现核心调控元件以及基因表达多样性调控元件的功能和机制;另一方面可通过增强子来调控干细胞发育潜能、体细胞可塑性,以及针对增强子异常调控引起的疾病开展新的治疗策略。
需要指出的是,本研究仅在发育的早期和成熟2个阶段研究了DNA序列的保守性与发育的关系。未来还需要进一步收集更多的发育阶段和组织类型数据进行深入的拓展研究。也可以进一步通过评估DNA序列保守性,来探索其在单细胞和谱系追踪研究中的应用潜力。
作者贡献声明
李令杰和张丰负责实验设计;王志明收集整理数据并进行分析;张丰、童冉、杨晨、焦慧媛、王一好、李林颖和王烨欣协助参与了数据分析;所有作者参与了论文的写作和修改。所有作者均阅读并同意了最终稿件的提交。
AUTHOR's CONTRIBUTIONS
The study was designed by LI Lingjie and ZHANG Feng. The data were collected and analyzed by WANG Zhiming. ZHANG Feng, TONG Ran, YANG Chen, JIAO Huiyuan, WANG Yihao, LI Linying and WANG Yexin contributed to the data analysis. The manuscript was drafted and revised by all the authors. All the authors have read the last version of paper and consented for submission.
利益冲突声明
所有作者声明不存在利益冲突。
COMPETING INTERESTS
All authors disclose no relevant conflict of interests.
参考文献