人类巨噬细胞是免疫系统的重要参与者[1-2],不但与多种感染性疾病相关(如登革热、结核病、获得性免疫缺陷综合征等),还与自身免疫病关系密切[3-5]。此外,巨噬细胞疗法在临床治疗中的潜力也受到瞩目,利用巨噬细胞作为肿瘤等疾病治疗的工具与靶点的临床策略正逐步发挥重要影响[6-7]。然而,尽管巨噬细胞在病理生理学研究及临床治疗方面具有重要作用,但是巨噬细胞的来源却十分有限,用于科学研究的巨噬细胞往往来源于肿瘤细胞系,大量研究表明来源于肿瘤的细胞系与健康巨噬细胞有着迥异的功能与特征。而临床实践中,巨噬细胞自体移植主要来源于自身外周血,需要高度特异性的高成本临床诊疗方案支持。因而构建稳定的、可持续的健康巨噬细胞来源是目前临床与科研中的重要需求,也是构建巨噬细胞标准化产品的重要基础。
但针对人类iPSC来源的巨噬细胞(iPSC-derived macrophage,IPSDM)和目前常用的外周血来源的巨噬细胞(peripheral blood-derived macrophage,PBDM)之间异质性的研究目前较少。ALASOO等[16]曾使用RNA测序方法对比IPSDM与PBDM的转录组差异。为了进一步探究IPSDM与PBDM的细胞组成与转录组差异,本研究利用单细胞测序方法建立了这2种巨噬细胞的高分辨率单细胞转录图景,通过差异分析与富集分析探究它们的异质性,寻找IPSDM与PBDM潜在的功能差异,为IPSDM的临床应用提供理论支持。
1 材料与方法
1.1 细胞
人类iPSC(DYR0100)、人组织细胞淋巴瘤细胞U-937(TCHu159)来源于中国科学院细胞库。
1.2 主要试剂及仪器
RPMI 1640培养基(Gibco,11875093,美国),Dulbecco磷酸盐缓冲溶液(Dulbecco's phosphate buffered saline,DPBS;Gibco,14190144,美国),胎牛血清(fetal bovine serum,FBS;Gibco,10091148,美国),Matrigel基质胶(Corning BioCoat,354277,美国),β-巯基乙醇(Merck,21985023,德国),无酶消化液ReLeSR(Stemcell,100-0484,美国),mTeSR Plus干细胞完全培养基(Stemcell,85850,美国),人类骨形态发生蛋白(bone morphogenetic protein 4,BMP-4;PeproTech,120-05ET,美国),人类血管内皮生长因子(vascular endothelial growth factor,VEGF;PeproTech,100-20,美国),人类干细胞因子(stem cell factor,SCF;PeproTech,300-07,美国),X-VIVO 15无血清培养基(Lonza,02-060Q,瑞士),人类白细胞介素-3(interleukin-3,IL-3;PeproTech,200-03,美国),人类巨噬细胞集落刺激因子(macrophage colony stimulating factor,M-CSF;PeproTech,300-25,美国),Y-27632(Stemcell,72305,美国),抗CD14 PE-Cy7流式抗体(BD,557742,美国),细胞活力染料Fixable Viability Dye eFluorTM 780(Thermo Scientific,65-0865-14,美国),0.1%吉利丁溶液(Stemcell,07903,美国),青霉素-链霉素双抗溶液(Gibco,15140122,美国),反转录试剂盒PrimeScriptTM RT reagent Kit(TaKaRa,RR014A,日本),实时荧光定量PCR预混液Taq Pro Universal SYBR qPCR Master Mix(南京诺唯赞生物科技股份有限公司,Q712),TRIzol(Thermo Scientific,15596018,美国)。
C2+共聚焦显微镜(Nikon,日本),FACSAriaTM Fusion流式分选仪(BD,美国),VeritiTM Dx热循环仪(Thermo Scientific,美国),ViiATM 7实时荧光定量PCR系统(Thermo Scientific,美国),NanoDrop2000微量分光光度计(Thermo Scientific,美国)。
1.3 实验方法
1.3.1 U-937细胞培养方案
细胞培养于含5%CO2的37 ℃无菌细胞培养箱中。
将冻存的U-937细胞于37 ℃水浴快速复苏后,使用含有10%FBS及1%青霉素-链霉素双抗溶液的RPMI 1640完全培养基培养细胞,细胞每3 d按照1∶4的比例传代。
1.3.2 将人类iPSC分化为巨噬细胞
细胞培养及分化过程均在含5%CO2的37 ℃无菌细胞培养箱中进行。
将冻存的iPSC于37 ℃水浴快速复苏后,使用添加Y-27632的mTeSR Plus干细胞完全培养基重悬复苏的细胞以提高其活力。随后将其置于Matrigel基质胶预先包被好的细胞培养皿内,之后使用无添加的mTeSR Plus干细胞完全培养基每日换液,待细胞生长汇合度达到80%左右进行传代。
选择生长状态良好的细胞,待汇合度达到80%后进行类单核细胞分化实验。从培养皿中吸出所有培养液,用DPBS轻柔冲洗,小心吸尽DPBS后,用无酶消化液ReLeSR消化贴壁的iPSC;随后用干细胞完全培养基重悬细胞,肉眼可见未分化的iPSC团块漂浮在表面,尽量轻柔地收集这些细胞团块,利用重力自然沉降细胞团块,吸尽上清液后备用。
第一阶段胚体(embryonic body,EB)分化(1~7 d):配制EB分化培养基,即在干细胞完全培养基的基础上添加细胞因子BMP-4、VEGF、SCF,终浓度分别为50、50、20 ng/mL。用EB分化培养基重悬细胞团块,并将其均匀种植在超低黏附性细胞培养皿内,开始第一阶段由iPSC向EB分化。此阶段持续约7 d,在第4日添加等体积的新鲜EB分化培养基。
第二阶段单核-巨噬细胞分化(8~29 d):配制类单核细胞分化培养基,即在X-VIVO 15无血清培养基中按照体积比1∶100添加青霉素-链霉素双抗溶液,另外添加β-巯基乙醇、M-CSF和IL-3,终浓度分别为0.055 mmol/L、50 ng/mL、25 ng/mL。用10 mL的移液器枪头小心收集EB,尽量不要破坏EB的完整性;利用重力自然沉降EB后,使用类单核细胞分化培养基重悬,并将其均匀种植于预先用0.1%吉利丁包被的六孔细胞培养皿内,每孔约40个EB。在之后的3周内,每4 d更换1次新鲜的类单核细胞分化培养基。3周后,收集上清液中所有的悬浮细胞,即为IPSDM。收集到的IPSDM一部分用于流式细胞分析与RNA提取以便对细胞类型初步鉴定,另一部分进行单细胞测序。
1.3.3 反转录实时荧光定量PCR
分别取约1×106个IPSDM及U-937细胞,室温下280×g离心5 min去除上清液后得到细胞沉淀,使用TRIzol试剂按其说明书提取RNA。得到的RNA用微量分光光度计检测浓度,取500 ng RNA进行后续反转录实时荧光定量PCR(real-time reverse transcription quantitative polymerase chain reaction,RT-qPCR)反应。
使用PrimeScriptTM RT reagent Kit将RNA反转录得到cDNA,取1 μL cDNA进行qPCR反应,qPCR的引物序列如表1所示。反应程序:95 ℃ 15 s;95 ℃ 5 s,60 ℃ 30 s,共40个循环;95 ℃ 15 s,60 ℃ 1 min,95 ℃ 15 s。使用专用软件QuantStudio Real-Time PCR Software(v1.7.1)对RT-qPCR数据进行处理,使用核糖体蛋白L13a(ribosomal protein L13a,RPL13A)基因作为内参基因,并且用△△CT法计算以下目标基因在IPSDM和U-937细胞中的表达水平:主要组织相容性复合体Ⅱ类,DR alpha (major histocompatibility complex,class Ⅱ,DR alpha,HLA-DRA)基因、白细胞分化抗原86(cluster of differentiation antigen 86,CD86)基因、甘露糖受体C型1(mannose receptor C-type 1,MRC1)基因和CD68基因。
表1 RT-qPCR引物序列
Tab 1
| Gene | Forward primer (5´→3´) | Reverse primer (5´→3´) |
|---|---|---|
| HLA-DRA | AGCTGTGGACAAAGCCAACCTG | CTCTCAGTTCCACAGGGCTGTT |
| CD68 | CGAGCATCATTCTTTCACCAGCT | ATGAGAGGCAGCAAGATGGACC |
| CD86 | CCATCAGCTTGTCTGTTTCATTCC | GCTGTAATCCAAGGAATGTGGTC |
| MRC1 | AGCCAACACCAGCTCCTCAAGA | CAAAACGCTCGCGCATTGTCCA |
| RPL13A | CTCAAGGTGTTTGACGGCATCC | TACTTCCAGCCAACCTCGTGAG |
1.3.4 流式细胞术
取约1×106个抗IPSDM,将其重悬于200 μL DPBS中。使用抗CD14 PE-Cy7流式抗体与Fixable Viability Dye eFluorTM 780细胞活力染料对细胞进行染色,具体操作按照说明书。染色后用DPBS洗涤细胞2次,再用200 μL含2%胎牛血清的DPBS重悬细胞,置于4 ℃备用。使用FACSAriaTM Fusion流式分选仪检测细胞活力及CD14的表达水平,流式数据使用FlowJo软件(v10.8.1)分析。
1.3.5 IPSDM和PBDM的单细胞测序数据来源
制备IPSDM的单细胞测序样本,使用冷却的含2%胎牛血清的DPBS重悬IPSDM(由约1×107个iPSC分化后得到),4 ℃保存。单细胞测序上机及原始数据生成稀疏矩阵由上海生物芯片有限公司完成。
PBDM的单细胞测序数据来源于基因表达综合数据库(Gene Expression Omnibus,GEO;
1.3.6 IPSDM和PBMD的单细胞转录图景生成
为了获得PBMD与IPSDM的单细胞转录图景,我们使用R语言seurat包对单细胞测序得到的稀疏矩阵进行下游分析。对数据质控、过滤与标准化之后,寻找表达量差异最大的高变异基因,随后利用高变异基因进行主成分分析方法(principal component analysis,PCA)线性降维。
使用clustree包选择合适的聚类分辨率。根据聚类树图显示,随着分辨率增高,聚类得到的亚群数目会增加,新的亚群会从原有的亚群中产生,而不稳定的亚群也会消失。当大量透明箭头组成交织网络,意味着过度聚类。因此可以根据聚类树推断获得真实稳定亚群聚类时的分辨率进行后续分析。
细胞聚类后,继续使用seurat软件包进行t分布随机邻域嵌入(t-distributed stochastic neighbor embedding,tSNE)和均匀曲面逼近与投影(uniform manifold approximation and projection,UMAP)非线性降维。tSNE与UMAP非线性降维后获取PBMD和IPSDM的各细胞聚类的差异表达标志物,并基于这些标志物进行细胞类型注释。
对PBDM的细胞类型注释参考SingleR软件包自动注释结果。对IPSDM的细胞类型注释基于scmap软件包的分析结果,并参考细胞类型注释(annotation of cell types,ACT)工具[18]及相关研究。scmap是一种用于单细胞测序数据分析的R软件包,它的主要功能是进行细胞类型注释和基于参考的细胞聚类(reference-based clustering,RBC)。使用scmap包可以将已注释好的PBDM数据集的细胞高变异基因作为参考数据集,将需要查询的IPSDM数据集中的细胞高变异基因映射到参考数据集,由此推断IPSDM数据集中的细胞身份,同时比较2个数据集中的细胞高变异基因之间的相似性。scmapCluster的结果可以可视化为Sankey图,以显示细胞聚类的匹配方式。
1.3.7 IPSDM与PBDM的差异基因分析及富集分析
为了进一步探究IPSDM与PBDM的差异,我们对这2个样本中被注释为巨噬细胞的2个细胞亚群进行转录组差异表达分析,并依据差异表达基因(differentially expressed gene,DEG)进行基因本体(Gene Ontology,GO)分析(
2 结果
2.1 将iPSC分化为IPSDM
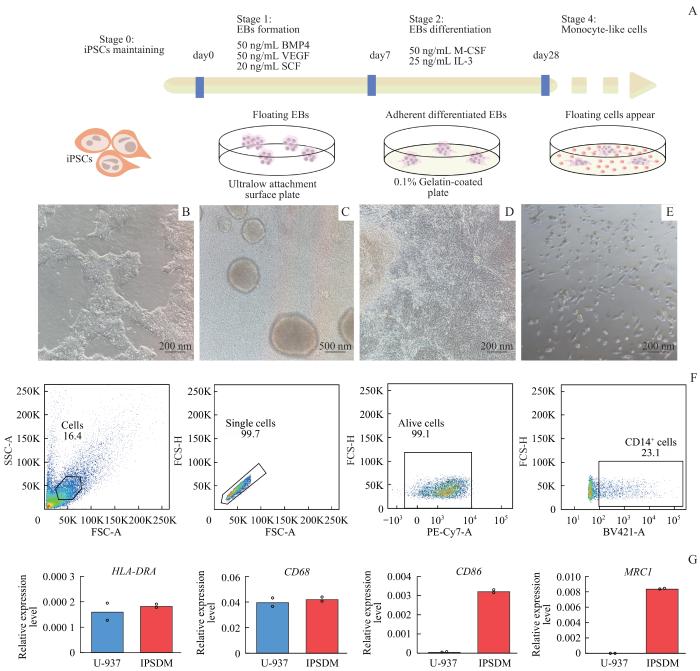
图1
图1
iPSC分化为IPSDM的过程及鉴定
Note: A. Flowchart of iPSCs differentiation into IPSDMs. B. Morphology of iPSCs used for differentiation (×100). C. Morphology of iPSCs on the 3rd day of differentiation into EBs (×40). D. Morphology of EBs on the 7th day of differentiation into monocytes (×100). E. Morphology of cells in the medium on the 21st day of EB differentiation into monocytes (×100). F. Flow cytometry analysis results of CD14 marker expression in IPSDMs. G. RT-qPCR results of HLA-DRA, CD68, CD86 and MRC1 gene expression levels in IPSDMs and U-937 cells.
Fig 1
Process and identification of iPSCs differentiation into IPSDMs
通过流式细胞术检测IPSDM表面CD14的表达。CD14是髓系细胞特异性的表面抗原,在单核细胞及巨噬细胞表面均有表达。结果(图1F)显示约23.1%的IPSDM表达CD14抗原,说明IPSDM细胞具有髓系细胞的特征。
为了进一步明确IPSDM的种类,通过RT-qPCR技术检测更多的单核-巨噬细胞标志基因的表达水平。U-937细胞系是一种具有单核细胞样特征的细胞系,使用U-937细胞的cDNA作为对照,评估IPSDM的单核细胞及巨噬细胞的表面标志基因的表达水平。结果(图1G)显示,单核-巨噬细胞表面标志基因HLA-DRA及CD68在IPSDM及U-937细胞中有相当的表达,而巨噬细胞特异性的表面标志基因CD86及MRC1在IPSDM中高表达,而U-937细胞中表达量极低。说明IPSDM更具有巨噬细胞表面标志的特征。
2.2 PBDM的单细胞基因组学分析
为了更好地了解IPSDM的单细胞转录组特征,我们采用了单细胞测序手段来评估IPSDM的细胞组分和单细胞转录组特征,并使用PBDM的单细胞转录组数据作为参考数据集。
作为参考的PBDM数据集是来源于GEO公共数据库的数据集GSE126085[17],该数据集为外周血来源的CD14+细胞经M-CSF体外诱导分化6 d后得到细胞的单细胞测序数据。
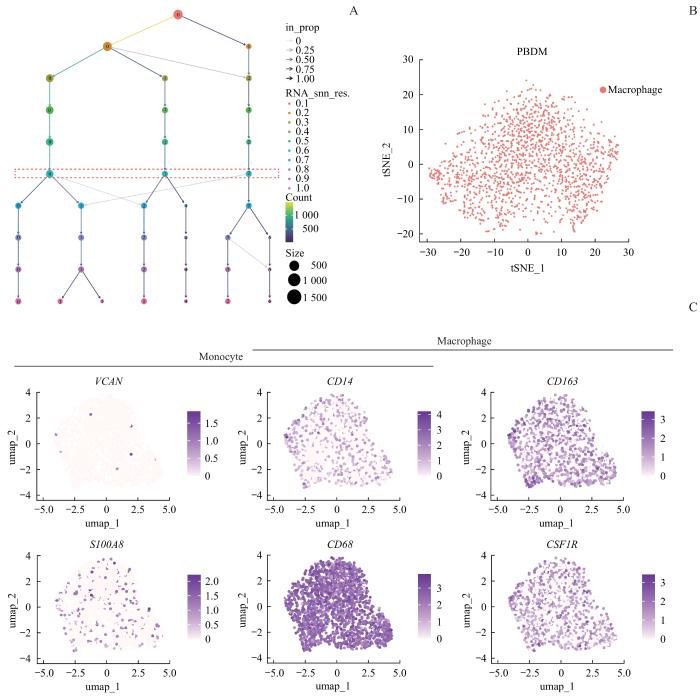
下载数据后,首先对PBDM的单细胞测序数据进行质控、降维。为了确定最合适的聚类分辨率,生成聚类树(图2A),结果可见聚类树从分辨率0.6开始出现较多半透明的分支,代表着不稳定的分群,可能造成过度聚类,因此选择聚类分辨率0.6进行后续分析,此时细胞可以形成3个聚类。参考SingleR软件包注释结果及相关研究[19]对细胞类型进行注释,发现PBDM中的3个聚类的细胞类型均为巨噬细胞,如图2B所示。检查PBDM中与单核-巨噬细胞相关的表面标志物的基因表达状况,基因表达分布特征图如图2C所示,单核细胞高表达的标志物多能蛋白聚糖(versican,VCAN)基因与S100钙结合蛋白A8(S100 calcium binding protein A,S100A8)基因在PBDM中的表达分布已经十分零星;作为单核-巨噬细胞共享的标志基因CD14与CD68在PBDM中的表达模式具有典型巨噬细胞的特征,即CD14的表达较低而CD68的表达水平较高;另外,巨噬细胞特异性的CD163基因及集落刺激因子1受体(colony stimulating factor 1 receptor,CSF1R)基因在大多数PBDM中均有较高的表达。这些标志基因的表达模式证实了GSE126085数据集中的细胞为巨噬细胞。
图2
图2
GSE126085中PBDM样本细胞聚类过程及注释后的细胞分布特征图
Note: A. The cluster tree of the PBDM sample. B. Distribution of the PBDM sample after annotation. C. Feature distribution plots of marker gene expression in the PBDM sample.
Fig 2
Cell clustering process and distribution feature plot of the PBDM samples after annotation in GSE126085
2.3 IPSDM单细胞基因组学图景
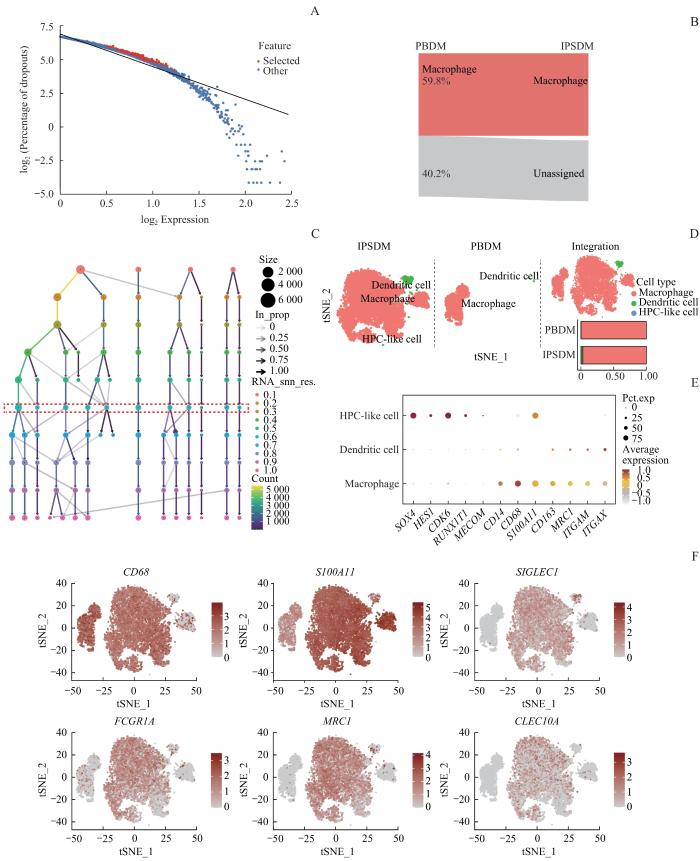
为了描绘IPSDM单细胞基因组学图景,我们先整合IPSDM数据与PBDM数据。首先以注释后的PBDM单细胞的高变基因数据构建参考数据集,使用scmap算法将IPSDM样本的高变基因映射于PBDM参考数据集,以寻找IPSDM样本中与PBDM潜在的相似成分。选择PBDM中信息量最大的一批基因(图3A)作为后续投影的参考特征,基于这些参考基因构建scmap的聚类索引。随后使用scmapCluster算法通过映射对比IPSDM与PBDM样本,投影结果(图3B)显示,59.8%的IPSDM依据PBDM的索引可以被注释为“巨噬细胞”,而余下的40.2%IPSDM无法从PBDM得到映射。说明IPSDM中的部分细胞高变基因表达谱与PBDM中的高变基因表达谱存在相似性,但也存在一定的差异。因此IPSDM中的细胞亚群需要进一步注释分析。
图3
图3
IPSDM样本的单细胞基因组学图景
Note:A. Distribution of PBDM index genes. B. The cell flow for projecting the IPSDM dataset onto the PBDM dataset. C. The cluster tree of IPSDM and PBDM samples. D. Distribution of IPSDM and PBDM samples after annotation. E. Bubble plots showing the expression levels of cell marker genes in different cell types. F. Expression distribution of tissue-resident macrophage-specific marker genes of IPSDM and PBDM samples.
Fig 3
Single-cell RNA genomics landscape of IPSDM samples
我们参考ACT工具及相关研究[18-20],对IPSDM的单细胞测序数据进行人工分析与注释。首先通过生成聚类树确定合适的聚类分辨率为0.6(图3C)。根据每个聚类的细胞特异性标志基因的表达对聚类进行注释。注释后的细胞类型、对应的标志基因和细胞占比如表2所示。IPSDM最主要的组分为巨噬细胞(图3D),占97.15%。这群细胞高表达巨噬细胞重要的标志基因CD68,巨噬细胞其他相关标志基因CD14、CD163与S100钙结合蛋白A11(S100 calcium binding protein A11,S100A11)在巨噬细胞中亦有广泛表达(图3E)。其次是造血前体细胞样细胞(hematopoietic progenitor cell-like cell,HPC-like cell),占2.71%。这类细胞具有多能性,具有分化为单核-巨噬细胞的潜力,其标志基因包括SRY-Box转录因子4(SRY-box transcription factor 4,SOX4)、Hes家族BHLH转录因子1(Hes family BHLH transcription factor 1,HES1)、细胞周期蛋白依赖性激酶6(cyclin dependent kinase 6,CDK6)、RUNX1伴侣转录辅阻遏物1(RUNX1 partner transcriptional co-repressor 1,RUNX1T1)、MDS1和EVI1复合位点(MDS1 and EVI1 complex locus,MECOM)。第三类细胞被注释为树突状细胞,此类细胞的标志基因为整合素亚基αM(integrin subunit alpha M,ITGAM)和整合素亚基αX(integrin subunit alpha X,ITGAX);该类细胞具有ITGAM和ITGAX高表达但CD68低表达的特点,这群细胞仅占约0.14%。值得一提的是,在整合IPSDM与PBDM数据后同时注释2组细胞数据集,PBDM除了巨噬细胞外,还出现了极少量的造血前体细胞样细胞,仅占约0.11%。
表2 IPSDM与PBDM中细胞类型、对应的标志基因和细胞数
Tab 2
| Cell type | Marker gene | IPSDM/n(%) | PBDM/n(%) |
|---|---|---|---|
| Total | ‒ | 9 745 | 1 796 |
| Macrophage | CD14, CD68, S100A11, and CD163 | 9 467 (97.15) | 1 794 (99.89) |
| HPC-like cell | HES1, SOX4, CDK6, RUNX1T1, and MECOM | 264 (2.71) | 2 (0.11) |
| Dendritic cell | ITGAM and ITGAX | 14 (0.14) | 0 (0) |
注释后的细胞亚群分布图(图3D)显示,IPSDM与PBDM的巨噬细胞分布存在明显差异,因此我们查询了更多组织驻留巨噬细胞的标志基因[21]在IPSDM与PBDM的表达分布状态,如图3F所示。其中包括中枢神经系统驻留巨噬细胞标志基因——唾液酸结合免疫球蛋白样凝集素1(sialic acid binding Ig like lectin 1,SIGLEC1)、消化系统驻留巨噬细胞标志基因——Fcγ受体Ⅰa(Fc gamma receptor Ⅰa,FCGR1A)、皮肤驻留巨噬细胞标志基因——MRC1和含10A的C型凝集素结构域(C-type lectin domain containing 10A,CLEC10A)。不同于巨噬细胞通用的标志基因CD68或S100A11在IPSDM与PBDM中均有较高且广泛的表达,这些组织驻留巨噬细胞特异性的标志基因仅仅在IPSDM中存在表达分布,而在PBDM中几乎没有表达,提示通过无血清无饲养层细胞方案分化iPSC得到的巨噬细胞具有一定的组织驻留巨噬细胞的相关特征。
2.4 IPSDM和PBDM中的巨噬细胞转录组差异分析及基因富集分析
对IPSDM和PBDM的单细胞转录数据进行分析后,我们发现2种来源的巨噬细胞的基因表达图景具有较大差异。为了探究基因表达的差异性及背后潜在的生物学功能差异。我们对IPSDM及PBDM的单细胞转录组数据进行差异分析及基因富集分析。
首先我们通过差异表达分析,寻找IPSDM与PBDM中巨噬细胞之间的DEG。上调与下调变化倍数(fold change,FC)排名前10的DEG如表3所示,丝裂原活化蛋白激酶激酶激酶激酶3发散转录本(mitogen-activated protein kinase kinase kinase kinase 3 divergent transcript,MAP4K3-DT)、富含脯氨酸的核受体辅激活因子2(proline rich nuclear receptor coactivator 2,PNRC2)、末端核苷酸转移酶2(terminal nucleotidyltransferase 2,TENT2)等基因在IPSDM中表达高于PBDM,而丝裂原活化蛋白激酶14(mitogen-activated protein kinase 14,MAPK14)、4号染色体的结构维持蛋白(structural maintenance of chromosomes 4,SMC4)基因等在PBDM中表达更高。
表3 PBDM与IPSDM中的巨噬细胞之间上调与下调变化倍数排名前10的DEG
Tab 3
| Gene | Average log2FC | pct.1 | pct.2 | P value | Adjusted P value |
|---|---|---|---|---|---|
| MAP4K3-DT | 10.399 | 0.497 | 0 | 8.45×10-305 | 1.66×10-301 |
| PNRC2 | 10.246 | 0.498 | 0 | 2.28×10-305 | 4.50×10-302 |
| TENT2 | 10.154 | 0.488 | 0 | 8.07×10-297 | 1.59×10-293 |
| SELENON | 9.884 | 0.402 | 0 | 2.94×10-226 | 5.79×10-223 |
| RAB7B | 9.847 | 0.341 | 0 | 2.27×10-181 | 4.47×10-178 |
| FAAP20 | 9.780 | 0.396 | 0 | 2.85×10-221 | 5.61×10-218 |
| RAB29 | 9.757 | 0.405 | 0 | 1.58×10-228 | 3.11×10-225 |
| UTP11 | 9.677 | 0.370 | 0 | 6.19×10-202 | 1.22×10-198 |
| TMEM35B | 9.662 | 0.377 | 0 | 2.08×10-207 | 4.11×10-204 |
| ELOA | 9.568 | 0.376 | 0 | 7.27×10-207 | 1.43×10-203 |
| EMILIN1 | -3.939 | 0.049 | 0.278 | 7.48×10-244 | 1.47×10-240 |
| CCR5 | -2.685 | 0.147 | 0.319 | 3.16×10-102 | 6.22×10-99 |
| PHLDA3 | -2.645 | 0.160 | 0.430 | 7.26×10-208 | 1.43×10-204 |
| PCM1 | -2.429 | 0.578 | 0.719 | 5.15×10-262 | 1.02×10-258 |
| VAMP3 | -2.383 | 0.424 | 0.679 | 7.93×10-295 | 1.56×10-291 |
| RRM2 | -2.362 | 0.086 | 0.307 | 9.29×10-164 | 1.83×10-160 |
| AGO1 | -2.284 | 0.279 | 0.432 | 7.54×10-97 | 1.49×10-93 |
| ITPKB | -2.238 | 0.166 | 0.279 | 1.49×10-51 | 2.94×10-48 |
| SMC4 | -2.161 | 0.235 | 0.337 | 6.41×10-46 | 1.26×10-42 |
| MAPK14 | -2.077 | 0.372 | 0.499 | 1.82×10-102 | 3.58×10-99 |
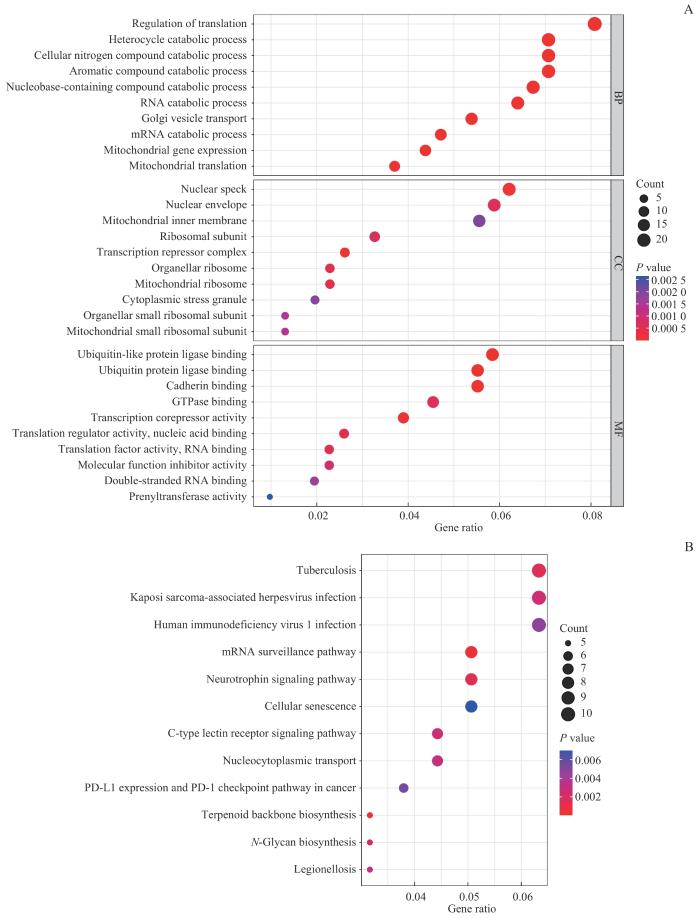
为了进一步探究IPSDM与PBDM之间的差异,我们针对∣log2FC∣>0.25且校正P<0.01的DEG进行GO分析及KEGG通路富集分析。GO分析结果(图4A)显示:分子功能主要富集于泛素样蛋白连接酶结合,可能与蛋白质泛素化降解相关;细胞组分主要定位于核斑点与核膜,可能涉及RNA的加工、转录修饰;生物学过程则主要富集于翻译调控。GO结果提示,DEG可能会影响包括蛋白质合成、降解在内的多种核心生物过程。为了进一步了解DEG背后潜在的生物学作用,我们又进行了KEGG通路富集分析,结果(图4B)显示排名前三的3个通路分别为结核病、卡波西肉瘤相关疱疹病毒感染、人类免疫缺陷病毒1型感染。这3类病原体均为胞内感染病原体,而巨噬细胞在胞内感染免疫中扮演重要角色。此外,KEGG通路富集分析还发现DEG富集于C型凝集素受体信号通路,该通路也与巨噬细胞的病原体识别及免疫反应触发有关。因此IPSDM与PBDM之间的DEG可能对巨噬细胞的免疫功能有潜在影响。
图4
图4
IPSDM和PBDM中的巨噬细胞DEG的功能分析
Note: A. GO enrichment analysis results of DEGs in the macrophages from the IPSDM sample vs the PBDM sample. B. KEGG pathway enrichment analysis results of DEGs in the macrophages from the IPSDM sample vs the PBDM sample.
Fig 4
Functional analysis of DEGs between the macrophages from PBDM and IPSDM
3 讨论
本研究首先在体外使用无饲养层无血清方案诱导iPSC分化为IPSDM,这一过程持续约1个月,最终收集的细胞样本中25%以上细胞表达CD14。通过scmap算法对不同来源的巨噬细胞的单细胞数据集的高变基因进行映射对比,结果发现iPSC来源的近60%细胞与PBDM具有一定相似性,另一部分则不具备,因此对IPSDM重新手动详细注释。我们发现IPSDM中的主要细胞亚群为巨噬细胞,约占98%,另外IPSDM中还有很少的造血前体细胞样细胞及树突状细胞。尽管这2种来源的细胞均高表达巨噬细胞特异性的表面标志物CD68,但是IPSDM还高表达组织驻留巨噬细胞相关标志物如SIGLEC1、FCGR1A、MRC1和CLEC10A。这意味着IPSDM可能与组织驻留巨噬细胞更相似。然而由于原代组织驻留巨噬细胞的单细胞测序数据目前较少,仍需要未来进行进一步的研究。
此外,为了进一步探索IPSDM与PBDM的异质性,我们通过对DEG的GO分析与KEGG通路富集分析发现,这些基因主要富集的生物学功能可能与翻译调控、蛋白质的合成和泛素化降解相关。特别是KEGG通路富集分析发现IPSDM与PBDM可能在面对胞内病原感染时表现出差异化的免疫功能。因此我们认为这两类巨噬细胞在转录组层面具有一定的差异,这些差异可能会影响到巨噬细胞针对胞内感染的免疫功能,但是相关的功能研究需要更多实验来探索。
本研究亦存在一定的局限性。首先本研究对于iPSC衍生细胞与原代细胞之间异质性的比较维度较为单一,而两者的形态学与功能学的异质性仍需要更多探索,如细胞因子分泌功能、抗原呈递功能、巨噬细胞的吞噬功能等等,这些研究对于后续iPSC衍生细胞的应用具有重大意义。另外,原代的组织驻留巨噬细胞与IPSDM的转录组学及功能差异也需要深入探究。
综上所述,本研究证明了iPSC向巨噬细胞分化的潜能,并且以PBDM为参考绘制了IPSDM的单细胞转录图景,并比较了2种不同来源的巨噬细胞之间的差异。
作者贡献声明
沈南、侯国俊参与实验设计;张羽桐完成实验操作;沈南、侯国俊、张羽桐参与论文的写作和修改;张羽桐负责数据处理。所有作者均阅读并同意了最终稿件的提交。
AUTHOR's CONTRIBUTIONS
The study was designed by SHEN Nan and HOU Guojun. The experiments were operated by ZHANG Yutong. The manuscript was drafted and revised by SHEN Nan, HOU Guojun and ZHANG Yutong. The data were analyzed by ZHANG Yutong. All the authors have read the last version of paper and consented for submission.
利益冲突声明
所有作者声明不存在利益冲突。
COMPETING INTERESTS
All authors disclose no relevant conflict of interests.
参考文献