葡萄球菌蛋白A(staphylococcal protein A,spa)基因是金黄色葡萄球菌特有的高度保守基因,并且该基因包含的短重复序列区域可以作为spa分型的靶点,因此将其作为检测样品中是否含有金黄色葡萄球菌并进行spa分型的目标区域(region of interest,ROI)[3-6]。MRSA对β-内酰胺类抗生素具有耐药性,且具有高传播性、多药耐药和高死亡率的特点[7-9]。葡萄球菌盒式染色体mec(staphylococcal cassette chromosome mec,SCCmec)基因元件中含有mecA耐药基因,当基因元件被盒式染色体重组酶(cassette chromosome recombinase,CCR)整合到金黄色葡萄球菌基因组中后,能够使其获得耐药性成为MRSA,因此以SCCmec基因元件作为第2个ROI能够检测样本中是否存在MRSA,并能够测出其SCCmec分型[10-11]。
长读长测序技术的出现为病原体检测领域带来了新的检测思路,许多新型检测技术应运而生。利用皂角苷去除宿主背景后进行纳米孔宏基因组测序能够快速准确地检测下呼吸道感染的病原菌[12]。在靶向测序方面,纳米孔靶向测序(nanopore targeted sequencing,NTS)技术能够对新型冠状病毒的毒力相关基因和特异性基因进行扩增,从而实现对病毒的快速、特异性检测[13];此外,TELSeq(target-enriched long-read sequencing)将另一长读长测序技术PacBio与靶向探针捕获技术结合,实现了对低丰度耐药基因的检测[14]。FLASH技术将成簇规律间隔短回文重复序列/相关蛋白9(clustered regularly interspaced short palindromic repeats/CRISPR-associated protein 9,CRISPR/Cas9)系统引入靶向测序,利用Cas9对ROI进行靶向切割,再经扩增实现富集,处理好的FLASH文库可以进行第二代测序(next-generation sequencing,NGS),实现对耐药基因的多重检测;而将文库投入纳米孔测序则能够缩短检测时间,实现耐药基因的快速多重检测[15-16]。纳米孔Cas9靶向测序(nanopore Cas9-targeted sequencing,nCATS)技术结合了CRISPR/Cas9系统的特异性识别和纳米孔测序的单分子长读长实时测序,并且无需PCR扩增即可对基因组中的ROI进行靶向测序。将该技术运用到临床诊断中能有效提高检测速度、检测灵敏度和特异性[17-19]。nCATS的靶向富集减少了分析所需的核酸投入量。例如,对乳腺癌组织的单核苷酸变异进行靶向测序,仅需投入3 μg基因组DNA即可获得足够测序深度[20]。本研究利用nCATS技术实现金黄色葡萄球菌的快速检测,并能够同时获得spa分型和SCCmec分型,帮助患者早期精准用药,且有助于高效开展分子流行病学调查。
1 材料和方法
1.1 材料
1.1.1 菌株
MRSA菌株由上海交通大学医学院附属仁济医院检验科提供,为血平板分离的纯培养物,经测定多位点序列分型(multilocus sequence typing,MLST)为ST5。
1.1.2 主要试剂和仪器
CRISPR RNA(crRNA)、反式激活crRNA(trans-activating crRNA,tracrRNA)[百力格生物科技(上海)股份有限公司],模拟靶标DNA、PCR引物[生工生物工程(上海)股份有限公司],Alt-R® Cas9蛋白(Integrated DNA Technologies,美国),细菌基因组DNA提取试剂盒(TIANamp Bacteria DNA Kit)、溶菌酶[天根生化科技(北京)有限公司],建库试剂盒(Native Barcoding Kit 24 V14;Oxford Nanopore Technologies,英国),平末端/TA连接酶预混液、FFPE DNA修复混合液、末端修复/加dA尾模块、快速连接模块、快速小牛肠碱性磷酸酶、NEBuffer r2.1(New England Biolabs,美国),ZymoBIOMICSTM微生物群对照Ⅱ(对数分布)[Microbial Community DNA Standard Ⅱ (Log Distribution);ZYMO RESEARCH,美国]。
Flow cell R10.4.1测序芯片、MinION Mk1C纳米孔测序仪(Oxford Nanopore Technologies,英国),NanoDrop One微量分光光度计(Thermo Fisher,美国)。
1.2 方法
1.2.1 crRNA的设计
以金黄色葡萄球菌的spa基因和SCCmec基因元件作为nCATS靶向测序的2个ROI,从美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)的GenBank获取其碱基序列(GenBank登录号分别为JQ066313.1和D86934.2)作为设计crRNA的模板。
使用CRISPOR工具(
使用生物大分子序列对比搜索工具BLAST(Basic Local Alignment Search Tool)检查序列具有保守性后,将导向序列中的胸腺嘧啶(T)替换为尿嘧啶(U),去除3'端3 nt的原间隔区邻近基序(protospacer adjacent motif,PAM),并在3'端添加用于与tracrRNA互补结合的16 nt序列,即构成36 nt的crRNA序列。
1.2.2 组装Cas9核糖核蛋白
以终浓度10 μmol/L,1∶1混合tracrRNA和crRNA,95 ℃退火5 min完成向导RNA(guide RNA,gRNA)组装。取2.8 μL 10×NEBuffer、3 μL gRNA、4.8 μL 1∶5稀释的Cas9蛋白和19.4 μL DEPC水,轻弹混匀,20 ℃或25 ℃孵育20 min,完成Cas9核糖核蛋白(ribonucleoprotein,RNP)组装。
1.2.3 模拟靶标DNA
针对每种crRNA设计42 bp的模拟靶标DNA用于验证与优化Cas9 RNP的切割能力,切割后将形成2段21 bp的切割产物。模拟靶标DNA为双链,将正链与负链1∶1混合,使终浓度为20 μmol/L,95 ℃退火5 min形成双链。模拟靶标DNA序列见表1。
表1 模拟靶标双链DNA序列
Tab 1
| crRNA | Strand | Corresponding simulated target DNA (5'→3') |
|---|---|---|
| spa-fw1-crRNA | + | ACCAAATACAGTTGTACCGATGAATGGATTTTCTTCACCAGT |
| - | ACTGGTGAAGAAAATCCATTCATCGGTACAACTGTATTTGGT | |
| spa-fw2-crRNA | + | TGCAATGTCATTTACTGTATCACCAGGTTTAACGACATGTAC |
| - | GTACATGTCGTTAAACCTGGTGATACAGTAAATGACATTGCA | |
| spa-rev1-crRNA | + | GTTACAGATGCAATACCTACACCTAGTTTACGAATTGAATAA |
| - | TTATTCAATTCGTAAACTAGGTGTAGGTATTGCATCTGTAAC | |
| spa-rev2-crRNA | + | GCAGCAGGTGTTACGCCACCAGATATAAGTAATGTACCTAAA |
| - | TTTAGGTACATTACTTATATCTGGTGGCGTAACACCTGCTGC | |
| SCC2-fw1-crRNA | + | ATTAATCGTATTAATAGAATACAAGGGCAACTAAATGGAATT |
| - | AATTCCATTTAGTTGCCCTTGTATTCTATTAATACGATTAAT | |
| SCC2-fw2-crRNA | + | TAATAGAATGTGTAAAAGTAGCTGCGGATGATGAAGAAAGCT |
| - | AGCTTTCTTCATCATCCGCAGCTACTTTTACACATTCTATTA | |
| SCC2-rev1-crRNA | + | AATTCTTGGGCCAATCCTTCGGAAGATAGCATCTTTCCTTGT |
| - | ACAAGGAAAGATGCTATCTTCCGAAGGATTGGCCCAAGAATT | |
| SCC2-rev2-crRNA | + | TACAAAGTCGCTTTGCCCTTGGGTCATGCGTTGGTTCAATTC |
| - | GAATTGAACCAACGCATGACCCAAGGGCAAAGCGACTTTGTA |
1.2.4 Cas9 RNP切割效率检测
取20 μL Cas9 RNP、1 μL模拟靶标DNA、4 μL 10×NEBuffer和15 μL DEPC水,轻弹混匀,37 ℃孵育。切割产物用10%尿素聚丙烯酰胺凝胶电泳分离,150 V电泳50 min,SYBR Gold染色20 min后成像。用ImageJ对目标条带进行灰度值(integrated density)分析,以切割产物灰度值占投入靶标DNA灰度值的百分比作为切割效率(cleavage efficiency)。
1.2.5 菌落计数
1 000 mL水中加40 g LB固体培养基粉末,高压灭菌后倒板得到LB平板。挑取1个菌落溶解于1 mL PBS中制成单菌落菌液,用微量分光光度计测定菌液在600 nm波长处吸光度。作10倍倍比稀释,每个稀释度涂布3块平板,每块滴加100 μL菌液。37 ℃培养24 h后选取菌落数介于30~300个的平板计数菌落数量。取3块平板,计算菌落数的平均值,从而推算原始菌液中的菌体浓度。
1.2.6 金黄色葡萄球菌基因组DNA抽提
在单菌落制成的110 μL 1×1010 CFU/mL菌液中加入90 μL溶菌酶溶液,37 ℃、600 r/min处理1 h,然后根据细菌基因组DNA提取试剂盒说明书进行抽提操作,25 μL抽提物稀释100倍后取25 μL投入nCATS体系(图1A)。
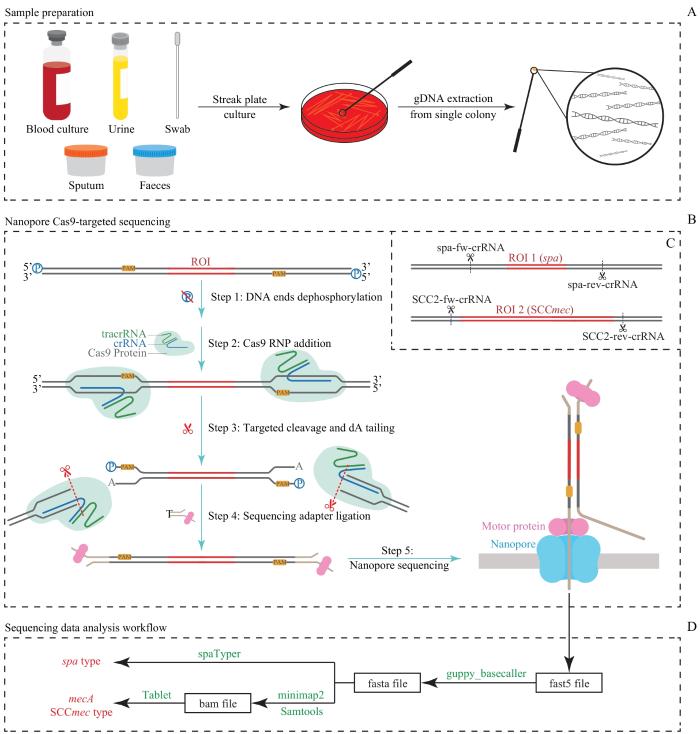
图1
图1
nCATS技术流程图
Note: A. Schematic of the sample preparation process. B. Schematic of the nCATS mechanism. C. Schematic of the two ROIs and four crRNA cleavage sites. D. Sequencing data analysis workflow.
Fig 1
Schematic diagram of nCATS workflow
1.2.7 nCATS
对样品中的总DNA做去磷酸化处理,后加入Cas9 RNP,经过设计的crRNA使Cas9 RNP能从患者及其他微生物的基因序列背景中精准识别并结合到ROI上下游序列,切割DNA双链。由于crRNA特异性识别的特点,非ROI序列不会被Cas9 RNP切割,双侧末端保持去磷酸化状态,无法被T4连接酶连接上测序接头,而ROI序列被Cas9 RNP识别并切割,在ROI双侧重新暴露出磷酸基团,从而能够连接上带有马达蛋白(motor protein)的测序接头。纳米孔测序时,马达蛋白将双链DNA解旋成单链并匀速牵引进纳米孔。crRNA的序列方向经过设计使得Cas9 RNP切割后会留在不含ROI的那段DNA上,阻挡了测序接头的连接反应,因此这段DNA虽然也重新产生了磷酸基团,依然难以连接上测序接头。nCATS原理见图1B,4种crRNA的切割位点见图1C。
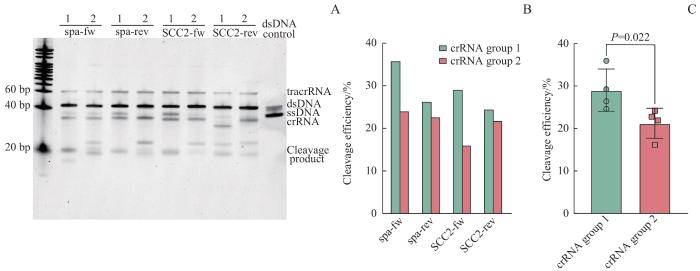
图2
图2
根据切割效率筛选crRNA
Note: A. Cleavage products of eight crRNAs from two groups. B. Cleavage efficiencies of eight crRNAs from two groups. C. Comparison of cleavage efficiencies between the two groups of crRNAs. dsDNA—double-stranded DNA; ssDNA—single-stranded DNA.
Fig 2
Screening of crRNAs based on cleavage efficiency
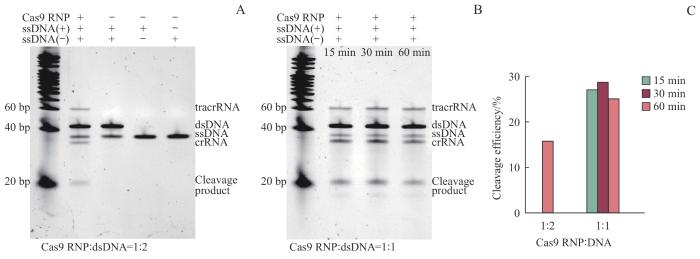
图3
图3
Cas9 RNP切割体系优化
Note: A. Verification of cleavage products using the Cas9 RNP cleavage system (Cas9 RNP:dsDNA=1:2) at 20 ℃ for 60 min. B. Cleavage products verification of Cas9 RNP cleavage system (Cas9 RNP:dsDNA=1:1) with different cleavage time at 20 ℃. C. Cleavage efficiencies of Cas9 RNP under different ratios and different cleavage time.
Fig 3
Optimization of Cas9 RNP cleavage system
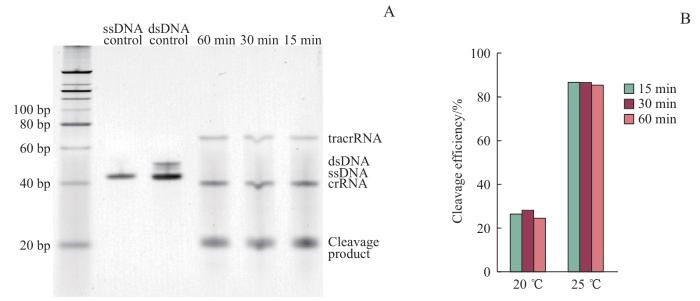
图4
图4
Cas9 RNP合成温度筛选
Note: A. Cleavage products of Cas9 RNP synthesized at 25 ℃, with different cleavage incubation times. B. Cleavage efficiencies of Cas9 RNP synthesized at 20 ℃ or 25 ℃, under different cleavage incubation times.
Fig 4
Screening of Cas9 RNP synthesis temperatures
(1) 基因组DNA末端去磷酸化。取3 μL 10×NEBuffer、25 μL基因组DNA、1 μL ZymoBIOMICSTM Microbial Community DNA Standard Ⅱ(8种细菌与2种真菌的基因组DNA混合物)和1 μL快速小牛肠碱性磷酸酶,轻弹混匀,37 ℃孵育10 min去磷酸化,80 ℃孵育2 min使酶失活。
(2) 靶向切割基因组DNA与加dA尾。取30 μL去磷酸化的产物、10 μL Cas9 RNP、1 μL 5 mmol/L dATP和1 μL 1∶5稀释的Taq DNA聚合酶,轻弹混匀,37 ℃孵育20 min使Cas9 RNP切割基因组DNA双链,72 ℃孵育5 min使Cas9 RNP失活,并在重新暴露了磷酸基团的DNA上加dA尾。
(3) 纳米孔测序文库构建与测序。依据SQK-CS9109建库试剂盒的说明书进行磁珠纯化、测序接头连接和文库上样,使用“fast”模式测序。
1.2.8 测序数据分析方法
使用guppy_basecaller工具(version 6.5.7)处理fast5格式的电流信号文件,选择“dna_r10.4.1_e8.2_400bps_sup.cfg”配置文件进行超高准确度碱基识别生成fasta碱基序列文件。使用NanoPlot工具(version 1.42.0)对测序结果进行质控,得到测序的数据量、碱基数、读长(read length)分布和质量值(read quality)等信息。将fasta文件上传至spa分型工具网站spaTyper(
表2 SCCmec 分型比对数据库所含序列GenBank登录号
Tab 2
| SCCmec type | GenBank No. |
|---|---|
| SCCmec Ⅱ | AB127982.1 |
| AB261975.1 | |
| AB433542.1 | |
| AB435013.1 | |
| AB435014.1 | |
| AB665981.1 | |
| AJ810120.1 | |
| D86934.2 | |
| SCCmec Ⅲ | AB038513.1 |
| AB037671.1 | |
| AB047088.2 | |
| AB047089.2 | |
| AB425427.1 |
1.2.9 PCR法鉴定spa分型
配制引物终浓度为500 nmol/L的PCR反应体系,反应条件为:94 ℃预变性5 min;94 ℃变性45 s,60 ℃退火45 s,72 ℃延伸90 s,共35个循环;最终72 ℃延伸10 min。用1.5%琼脂糖凝胶电泳成像验证PCR产物纯度后进行一代测序。将测序结果fasta文件上传至spaTyper获得spa分型结果。PCR法鉴定spa分型使用的引物序列见表3。
表3 PCR法鉴定 spa 分型使用的引物序列
Tab 3
| Primer | Sequence (5'→3') |
|---|---|
| spa-1113f | TAAAGACGATCCTTCGGTGAGC |
| spa-1514r | CAGTAGTGCCGTTTGCTT |
1.2.10 多重PCR法鉴定SCCmec分型
表4 多重PCR法鉴定SCCmec 分型使用的引物序列
Tab 4
| mPCR | Primer | Sequence (5'→3') |
|---|---|---|
| mPCR1 | mecA1 | TGCTATCCACCCTCAAACAGG |
| mecA2 | AACGTTGTAACCACCCCAAGA | |
| ccrA1 | AACCTATATCATCAATCAGTACGT | |
| ccrB123 | ATTGCCTTGATAATAGCCTTCT | |
| ccrA2 | TAAAGGCATCAATGCACAAACACT | |
| ccrA3 | AGCTCAAAAGCAAGCAATAGAAT | |
| ccrA4 | GTATCAATGCACCAGAACTT | |
| ccrB4 | TTGCGACTCTCTTGGCGTTT | |
| mPCR2 | mecI | CATAACTTCCCATTCTGCAGATG |
| mecA | ATATACCAAACCCGACAACTACA | |
| IS1272 | ATGCTTAATGATAGCATCCGAATG | |
| IS431 | TGAGGTTATTCAGATATTTCGATGT |
2 结果
2.1 crRNA设计结果
spa基因的设计模板序列(GenBank登录号 JQ066313.1)中有93处潜在导向序列,SCCmec Ⅱ基因的设计模板序列(GenBank 登录号D86934.2)的上游和下游1 000 bp中分别有72处和63处潜在导向序列。筛选出2组理论切割效果最佳的crRNA(表5),每组含4种crRNA,分别对应4个切割位点。
表5 Cas9 RNP中的RNA序列
Tab 5
| Name | Sequence (5'→3') |
|---|---|
| tracrRNA | AGCAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCUUU |
| spa-fw1-crRNA | AAUACAGUUGUACCGAUGAAGUUUUAGAGCUAUGCU |
| spa-rev1-crRNA | UCAAUUCGUAAACUAGGUGUGUUUUAGAGCUAUGCU |
| SCC2-fw1-crRNA | AUCGUAUUAAUAGAAUACAAGUUUUAGAGCUAUGCU |
| SCC2-rev1-crRNA | GGAAAGAUGCUAUCUUCCGAGUUUUAGAGCUAUGCU |
| spa-fw2-crRNA | AUGUCAUUUACUGUAUCACCGUUUUAGAGCUAUGCU |
| spa-rev2-crRNA | GGUACAUUACUUAUAUCUGGGUUUUAGAGCUAUGCU |
| SCC2-fw2-crRNA | AGAAUGUGUAAAAGUAGCUGGUUUUAGAGCUAUGCU |
| SCC2-rev2-crRNA | UGAACCAACGCAUGACCCAAGUUUUAGAGCUAUGCU |
2.2 crRNA切割效率筛选
分别使用2组8种crRNA构建Cas9 RNP切割体系,对比每个切割位点所对应的2个候选crRNA的切割效率。结果(图2)显示第1组的平均切割效率显著高于第2组(P=0.022),因此后续选择第1组crRNA进行实验。
2.3 Cas9 RNP切割体系优化
使用模拟靶标DNA验证Cas9 RNP的切割能力,并对切割体系中Cas9 RNP的比例与切割的时间进行优化。以spa-fw1-crRNA构建的Cas9 RNP为例,实验结果表明:增加Cas9 RNP在切割体系中的比例有助于提高Cas9 RNP的切割效率。在孵育60 min的条件下,Cas9 RNP与靶标dsDNA比例为1∶2时的切割效率为16.01%,提升Cas9 RNP的比例至1∶1后切割效率提高到25.31%。固定Cas9 RNP与靶标DNA的比例为1∶1,进行切割时间梯度实验,Cas9 RNP对靶标DNA分别进行15、30和60 min的切割反应,结果显示切割15 min时Cas9 RNP的切割效率达到27.28%,与切割30 min时28.94%和切割60 min时25.31%无明显差异,提示Cas9 RNP切割体系在15 min时切割能力即达到饱和(图3)。
2.4 Cas9 RNP合成温度筛选
经过Cas9 RNP切割体系优化和crRNA切割效率筛选,本研究Cas9 RNP能够达到的切割效率约为30%,效率较低。因此我们以spa-fw1-crRNA构建的Cas9 RNP为例,对构建条件进行优化。当Cas9 RNP的合成温度为20 ℃时,切割效率约为30%(图3B、C);当把Cas9 RNP的合成温度提升至25 ℃后,电泳图中靶标DNA的条带几乎不可见,切割产物的条带颜色加深(图4A)。灰度值分析结果显示,合成温度提升至25 ℃后,切割15、30、60 min时的切割效率均提升至2倍以上,分别为87.41%、87.37%和86.16%(图4B)。3个时间点之间切割效率无明显变化,因此切割时间仍定为15 min。
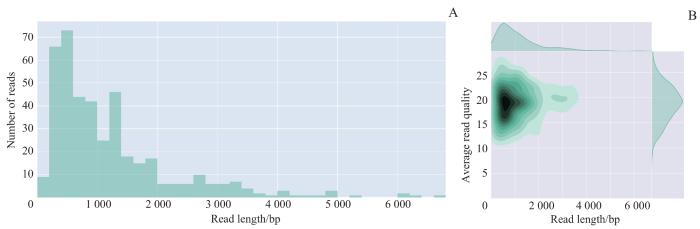
2.5 nCATS数据质控
图5
图5
nCATS测序数据质控结果
Note: A. Histogram of sequencing read length. B. Plot of sequencing read length and average read quality.
Fig 5
Quality control results of nCATS sequencing data
2.6 nCATS分型结果
将fastq文件转换为fasta文件,上传至spaTyper得到样本中金黄色葡萄球菌的spa分型为t2。用Tablet打开与金黄色葡萄球菌基因组比对后的bam文件和mecA基因比对数据库,按照mismatch%不超过5%的标准过滤结果,得到样本中含有mecA基因,即含有MRSA。与SCCmec分型比对数据库比对后,共有61条read比对到SCCmec Ⅱ型,其中33条read均比对到SCCmec Ⅱ型的D86934.2基因上,按照mismatch%不超过5%的标准过滤结果,得到唯一分型结果,即SCCmec Ⅱ型。
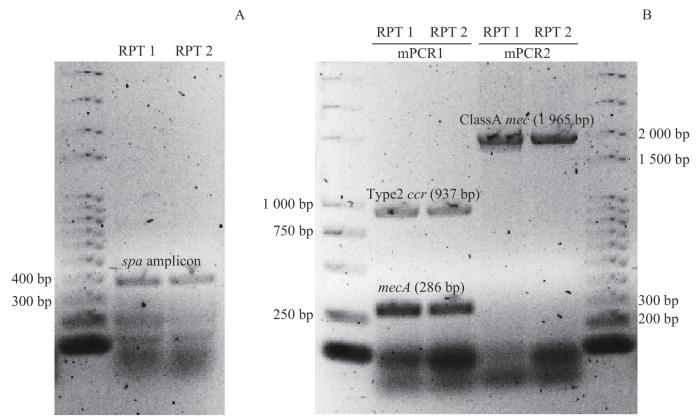
2.7 PCR法分型结果
图6
图6
PCR和多重PCR法分型结果
Note: A. Verification of spa amplicons. B. SCCmec typing results from multiplex PCR. RPT—repeat.
Fig 6
Typing results obtained using PCR and multiplex PCR methods
3 讨论
病原体的快速、精准检测一直是临床治疗感染性疾病和预防大范围传染性疾病暴发的关键步骤。目前临床诊断的金标准是微生物培养法,该方法特异性高,操作简便,但灵敏性不高且耗时较长,一般需要18~24 h才能获得有识别特征的菌落,使得临床无法在早期对患者进行精准治疗。除此之外,耐药性检测也是辅助临床治疗决策中不可或缺的步骤,但目前的金标准纸片扩散法同样存在耗时长的局限性,需在获得纯培养物后再进行18~24 h的纸片扩散法培养,无法在早期辅助临床制定治疗策略。针对上述限制,研究人员开发出分子生物学检验方法,在保持检测灵敏度和特异性的同时将检测时间由数天缩短到数小时,检测靶标一般是病原体基因组中的特定基因,例如检测MRSA常用的mecA基因[23-25]。但分子生物学检验方法同样存在局限性,PCR中难以避免的扩增错误有可能导致假阳性结果,并且对于金黄色葡萄球菌,在以SCCmec基因元件为靶标的PCR检测中,由于mecA丢失(mecA dropout)现象,有可能将甲氧西林敏感菌株误判为MRSA,造成抗MRSA药物的不必要使用[1,25]。
此外,对病原体进行分子生物学分型能够帮助社区和医院及时了解不同菌株的时空分布特征、耐药性变化及系统发育趋势,有助于高效协助分子流行病学调查,更好地预防和控制该类病原菌的感染与传播。目前多数病原体的分子生物学分型判定都无法与病原体检测同时进行,需要花费额外的时间和费用。
本研究使用nCATS技术,实现对金黄色葡萄球菌同时进行快速检测和分型。该方法建库流程仅需3 h,与传统微生物培养法相比缩短了检测时间,且检测过程不涉及PCR扩增,规避了扩增假阳性的风险。由于nCATS能同时对检测到的菌株进行spa分型和SCCmec分型,在临床使用中能够实现高效的分子流行病学调查,使金黄色葡萄球菌感染性疾病得到更好的控制和预防。该方法成功在8种细菌和2种真菌存在的复杂背景中对107 CFU的MRSA-ST5菌株进行快速检测,并测得该菌株为SCCmec Ⅱ型MRSA,spa分型为t2,与PCR方法所得结果均一致。
该方法设计性强,对于可能存在突变的ROI双侧序列,只需设计多组crRNA,使其覆盖的识别区域增长,即至少有1组crRNA不受序列突变的影响,从而保证该方法在突变存在时依然能够维持敏感性。同样,针对不同病原体设计相应的crRNA即可实现病原微生物的多重检测。此外,该方法使用的建库方法可以同时检测多例患者样本,能够大幅降低单样本的检测成本。
本方法尚存在2个可进一步优化的方向。其一,细菌基因组DNA提取效率是制约本方法灵敏度的重要因素。金黄色葡萄球菌作为厚壁菌门成员,其细胞壁结构致密,常规提取方法难以使其完全裂解。后续研究可尝试结合物理破碎(如超声处理、珠磨法)或生物酶解(如溶葡萄球菌素处理)等技术,实现细胞壁的高效裂解,进而提升基因组DNA的提取效率,降低检测所需的样本起始量。其二,在nCATS的建库流程中,多次磁珠清洗步骤会不可避免地导致样本损失,因此开发更为温和且高效的清洗策略,减少样本在处理过程中的损耗,是提高建库产率、优化该技术体系的关键所在。
作者贡献声明
王鹏飞负责研究设计和项目管理,潘抒凡负责实验操作,杨栋磊和潘抒凡负责数据分析,王鹏飞、杨栋磊和潘抒凡参与论文撰写。所有作者均阅读并同意最终稿件的提交。
AUTHOR's CONTRIBUTIONS
WANG Pengfei designed and supervised the project. PAN Shufan conducted the experiments. YANG Donglei and PAN Shufan analyzed the data. WANG Pengfei, YANG Donglei and PAN Shufan completed the manuscript. All authors have read the final version of paper and consented to submission.
利益冲突声明
所有作者声明不存在利益冲突。
COMPETING INTERESTS
All authors disclose no relevant conflict of interests.
参考文献