小泛素样修饰蛋白(small ubiquitin-like modifier,SUMO)是存在于真核生物细胞内的一种蛋白质,相对分子质量约12 000[1]。在SUMO化修饰过程中,SUMO被传递至底物蛋白并与其共价结合。SUMO化作为一种蛋白质翻译后修饰,其通过影响蛋白质的定位、激活、转运等过程,参与细胞信号转导、周期调控、凋亡和增殖等多种重要生理活动[2]。SUMO化已被证实在白血病、乳腺癌、前列腺癌、肺癌、黑色素瘤等多种癌症的发生发展中发挥重要作用。异常上调的SUMO化水平被视为肿瘤发生的“推动者”或“保护者”,使癌细胞在应对基因毒性、蛋白质毒性、炎症等内外压力下保持活性[3]。靶向SUMO信号通路的抑制手段则具有抗癌治疗的潜力:一方面,SUMO化对细胞周期具有调控作用,阻断SUMO化可抑制细胞周期进程,尤其是在有丝分裂阶段;另一方面,SUMO化还是触发免疫激活基因表达的主要抑制因素,阻断SUMO化或可激活免疫系统、引发抗肿瘤反应[4]。
SUMO活化酶(SUMO activation enzyme,SAE)是SUMO化修饰过程中必不可少的唯一的激活酶,由2个亚基SAE1和SAE2组成;SAE1/SAE2复合体E1通过消耗ATP的2步水解反应激活SUMO蛋白的C端[5]。大量研究结果表明SAE1、SAE2的表达上调与包括高组织等级、高进展阶段、转移和不良预后等在内的肿瘤疾病的表型密切相关,证实了SAE介导SUMO化修饰,进而影响肿瘤的发生发展[6],使得SAE成为颇具潜力的肿瘤药物研发靶点。近年来陆续报道了SAE的多种抑制剂,包括TAK-981[7]、COH000[8]等小分子物质,以及银杏酸[9]、davidiin[10]等天然产物。然而,这些抑制剂均存在不同问题,限制了其药物应用价值。例如,银杏酸可诱导神经元细胞死亡;davidiin可显著抑制α淀粉酶活性并可能影响磷脂代谢通路;TAK-981在联合帕博利珠单克隆抗体治疗非小细胞肺癌和微卫星稳定(microsatellite stable,MSS)型结直肠癌的临床Ⅰb期研究中,有88%患者出现的不良事件[11]。
针对当前SAE抑制剂存在的问题或潜在的安全风险,开发一种通过靶向抑制SAE1与SAE2相互作用来破坏SAE的正常功能,进而抑制SUMO化通路的肽抑制剂是可行的策略。相较于化学小分子,肽类抑制剂同蛋白靶点之间具有更好的靶标特异性、强亲和力、不易在体内蓄积造成毒性等显著优势。此外,由于SAE1与SAE2的相互作用界面相较于SAE的活性位点具有较低的保守性,针对这一界面设计的多肽抑制剂作为一种SAE的非正构抑制剂,更易实现选择性抑制,从而减少不良反应和耐药性的产生[12-13]。因此,本研究拟建立一种可靠的应用于筛选SAE1与SAE2相互作用的多肽抑制剂的体外方法,获得多肽抑制活性的关键数据以支持后续的优化改造,也为类似蛋白-蛋白相互作用(protein-protein interaction,PPI)抑制剂筛选体系的建立提供有效参考。
1 材料与方法
1.1 细菌和细胞
大肠埃希菌DH5α、BL21(DE3)和Rosetta(DE3)感受态细胞(唯地生物,中国),人胰腺hTERT-HPNE细胞由本实验室保存。
1.2 主要试剂与仪器
QuickCut限制性内切酶(Takara,日本),2×Phanta Max Master Mix(Vazyme,中国),ClonExpress® Ⅱ One Step克隆试剂盒(Vazyme,中国),DNA标准品(DNA Ladder;Vazyme,中国),DNA纯化试剂盒(TIANGEN,中国),快速质粒小提试剂盒(TIANGEN,中国),卡那霉素(Sigma,美国),异丙基-β-D-硫代半乳糖苷(isopropyl β-D-thiogalactopyranoside,IPTG;Sigma,美国),二硫苏糖醇(dithiothreitol,DTT;Sigma,美国),三羟甲基氨基甲烷(trihydroxymethyl aminomethane,Tris;沪试,中国),咪唑(Sigma,美国),聚丙烯酰胺凝胶(polyacrylamide gel electrophoyresis,PAGE)快速制备试剂盒(雅酶,中国),One-step-blue考马斯亮蓝染液(Biotium,美国),三色预染蛋白标记物(雅酶,中国),二甲基亚砜(dimethyl sulfoxide,DMSO;Sigma,美国),Kinase-Glo试剂盒(Promega,美国),吐温-20(Tween-20;Sigma,美国),COH000(MCE,美国),多肽(肽谷生物)。
C1000 Touch PCR仪(Bio-Rad,美国),AH-MINI 1高压细胞破碎仪(ATS,中国),ÄKTA pure蛋白纯化仪(GE Healthcare,美国),Synergy Neo酶标仪(BioTek,美国),NanoDrop 2000分光光度计(Thermo Fisher Scientific,美国),88880020微孔板振荡仪(Thermo Fisher Scientific,美国),真MicroCal ThermoVac空脱气泵(GE Healthcare Life Science,美国),Microcal ITC200等温滴定量热仪(GE Healthcare Life Science,美国),SPR Biacore 8K及配套CM5芯片(Cytiva,美国)。
1.3 方法
1.3.1 质粒构建
从人胰腺hTERT-HPNE细胞cDNA中通过PCR技术分别扩增全长SAE1(参考序列NM 005500.3)、全长SAE2(参考序列NM 005499.2)和SUMO1的基因片段(第1~97位氨基酸),全部引物序列见表1。载体pET-28a分别经快速限制性内切酶BamH
表1 PCR引物序列
Tab 1
| Primer | Forward sequence (5′→3′) | Reverse sequence (5′→3′) |
|---|---|---|
| SAE1 | GCAAATGGGTCGCGGATCCGAATTCATGGTGGAGAAGGAGGAG | GTGGTGCTCGAGTGCGGCCGCATCACTTGGGGCCAAGG |
| SAE2 | GCAAATGGGTCGCGGATCCGAATTCATGGCACTGTCGCG | GTGGTGCTCGAGTGCGGCCGCATCAATCTAATGCTATGAC |
| SUMO1 | GCGCGGCAGCCATATGGCTAGCATGTCTGACCAGGAGGC | GGTGGTGGTGGTGGTGCTCGAGTTAACCCCCCGTTTGTTCC |
1.3.2 蛋白表达与纯化
将SAE1及SAE2的表达质粒转化至Rosetta(DE3)感受态细胞中,SUMO1的表达质粒转化至BL21(DE3)感受态细胞中。随后,涂板并挑选单克隆加入50 mL含卡那霉素的LB液体培养基中培养。以37 ℃,220 r/min于摇床上培养12~16 h。接着,再将培养物转入1 L含卡那霉素的LB液体培养基中培养,以37 ℃、220 r/min于摇床上培养至D(600 nm)0.6~0.8时加入IPTG进行诱导。不同蛋白的诱导条件见表2。诱导结束后离心收取菌体,并使用相应裂解缓冲液(不同蛋白纯化过程涉及的各缓冲液成分见表3)重悬菌体。经高压破碎仪破碎细胞后离心并收集上清液,先后经镍柱亲和层析纯化(SAE1、SAE2、SUMO1)和离子交换柱纯化(仅SAE2)后得到纯化蛋白。经SDS-PAGE验证蛋白的纯度及条带位置。如需冻存,将蛋白浓缩至5 mg/mL以上浓度,并添加30%甘油,经液氮速冻后保存至-80 ℃冰箱。
表2 不同蛋白的诱导表达条件
Tab 2
| Protein | IPTG concentration/(mmol·L-1) | Temperature/℃ | Time/h |
|---|---|---|---|
| SAE1 | 0.3 | 10 | 36 |
| SAE2 | 0.3 | 10 | 36 |
| SUMO1 | 1.0 | 16 | 24 |
表3 不同蛋白使用的缓冲液
Tab 3
| Buffer | Component | |||
|---|---|---|---|---|
| Tris-HCl (pH 8.0)/(mmol·L-1) | NaCl/(mmol·L-1) | Imidazole/(mmol·L-1) | DTT/(mmol·L-1) | |
| SAE1 lysis buffer | 50 | 300 | 10 | 1 |
| SAE1 Ni-NTA equilibration/wash buffer | 50 | 50 | 20 | 1 |
| SAE1 Ni-NTA elution buffer | 50 | 50 | 40 | 1 |
| SAE2 lysis buffer | 50 | 300 | / | 1 |
| SAE2 Ni-NTA equilibration/wash buffer | 50 | 50 | / | 1 |
| SAE2 Ni-NTA elution buffer | 50 | 50 | 25 | 1 |
| SAE2 anion exchange buffer A | 50 | 50 | / | 1 |
| SAE2 anion exchange buffer B | 50 | 1 | / | 1 |
| SUMO1 lysis buffer | 50 | 500 | 20 | / |
| SUMO1 Ni-NTA equilibration/wash buffer | 50 | 500 | 20 | / |
| SUMO1 Ni-NTA elution buffer | 50 | 50 | 400 | / |
1.3.3 等温滴定量热检测实验
在等温滴定量热检测(isothermal titration calorimetry,ITC)实验中,先将蛋白置于50 mmol/L Tris-HCl(pH 8.0)、300 mmol/L NaCl和1 mmol/L DTT组成的反应缓冲液中,在4 ℃透析过夜。随后,浓缩蛋白至适宜浓度,并使用相同缓冲液将待测多肽样品稀释至适宜浓度。确保所有样品均需充分脱气。滴定前检查仪器状态,充分清洗上方滴定针及下方样品池。使用脱气后的反应缓冲液润洗样品池后,将200 μL蛋白样品用注射器加入样品池中。控制程序向滴定针中加载多肽样品40 μL,注意避免产生气泡。在仪器的程序设定中输入温度(25 ℃)、样品浓度、滴定体积(2 μL/滴,共20滴)、采样间隔(120 s)、参考功率(5.0 µcal/s)等参数后启动滴定程序。反应结束后清洗滴定针及样品池,于配套Origin软件中进行数据分析处理。
1.3.4 荧光偏振实验
在荧光偏振(fluorescence polarization,FP)实验中,先使用50 mmol/L Tris-HCl(pH 8.0)、300 mmol/L NaCl和1 mmol/L DTT组成的反应缓冲液配制所需不同浓度的蛋白和示踪剂(tracer)多肽样品,每孔体积为100 μL,每个实验组做3个复孔。在室温下,摇床250 r/min孵育1 h。而后使用Synergy Neo酶标仪检测FP值,检测相关参数为发射光528 nm、激发光485 nm,校正因子G设置为1.75。
1.3.5 表面等离子共振实验
使用Biacore 8K仪器进行表面等离子共振(surface plasmon resonance,SPR)实验,操作遵循仪器使用标准操作程序。① 配体偶联:使用含0.05%吐温-20的PBS(经0.22 μmol/L滤膜过滤并充分脱气)作为缓冲液,偶联SAE1蛋白样品使用pH 5.0的NaAc缓冲液稀释至20~50 μg/mL,经活化、配体注入和封闭后固定于CM5芯片上。② 蛋白-多肽互作样品检测:配体偶联后,更换缓冲液为含有5%DMSO和0.05%吐温-20的PBS(经0.22 μmol/L滤膜过滤并充分脱气),分别配制含有4%、4.67%、5%、5.33%、6%DMSO的缓冲液用作溶剂矫正;根据实验需要设置进样程序,配制梯度稀释的多肽样品并在96孔板指定位置加样,程序运行完毕后于配套软件中分析实验结果。
1.3.6 基于SAE酶活的荧光实验
在不透明白色384孔板中进行,每个反应的总体积为20 μL。反应缓冲液成分为:50 mmol/L Tris-HCl(pH 8.0)、50 mmol/L NaCl、5 mmol/L MgCl2和0.01%吐温-20。反应组分包括:1.25 μmol/L SAE1、1.25 μmol/L SAE2、200 μmol/L SUMO1和200 μmol/L ATP。所有多肽溶解于DMSO(终浓度5%)。首先将SAE1、SAE2和多肽混合后于4 ℃摇床孵育2 h。随后,加入ATP和SUMO1蛋白,于37 ℃、摇床500 r/min反应45 min,平衡至室温。接着,加入Kinase-Glo试剂盒中的检测试剂于室温、摇床200 r/min反应10 min。使用酶标仪检测荧光值,仪器设置参见试剂盒说明书。
2 结果
2.1 蛋白表达与纯化
分别纯化了全长的SAE1蛋白、全长的SAE2蛋白及成熟的SUMO1蛋白(体内环境下经SUMO特异性蛋白酶剪切后暴露C端双甘氨酸残基的形式,包含1~97位氨基酸),用于后续各筛选体系的建立。
2.2 ITC实验
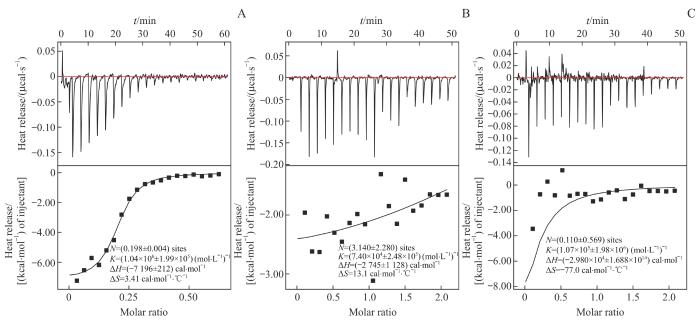
由于首批设计合成的11条多肽截取自SAE1和SAE2共晶揭示的SAE1上同SAE2结合的热点片段(对蛋白相互作用结合能贡献最大的区域),因此使用ITC检测多肽同SAE2蛋白之间的相互作用。首先检测了SAE1与SAE2之间的相互作用以验证纯化蛋白的体外活性及该方法的可靠性。在尝试不同滴定浓度组合后,于300 μmol/L SAE1滴定100 μmol/L SAE2的实验中获得了较理想的“S”型滴定曲线(图1A),结果表明反应为放热反应,测得解离常数(Kd)为0.96 μmol/L。随后,为进一步检测多肽与SAE2蛋白的结合能力,经滴定浓度组合优化,选定300 μmol/L多肽、30 μmol/L SAE2蛋白进行ITC筛选。然而,由于这一批多肽的亲和力较低,ITC无法获得标准的滴定曲线用于结合能的测算。因此,只能根据放热进行粗略筛选,确定表现最优的2条多肽为PEPT7(图1B)和PEPT2(图1C)。
图1
图1
ITC筛选体系的建立
Note: A. ITC experiments for the binding of SAE1 to SAE2. B. ITC experiments for the binding of PEPT7 to SAE2. C. ITC experiments for the binding of PEPT2 to SAE2. 1 cal=4.184 J. N—the number of sites per SAE2; K—the binding constant; ΔH—heat change; ΔS—entropy change.
Fig 1
Establishment of ITC screening system
2.3 FP实验
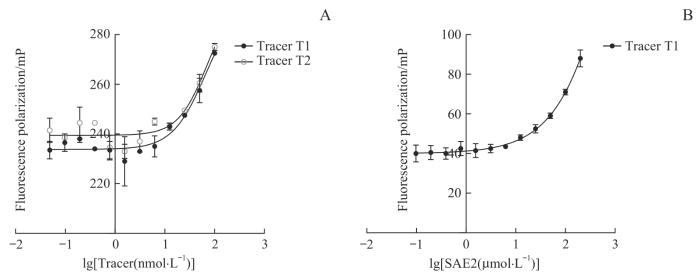
通过ITC确定了多肽PEPT7与SAE2亲和力最强。在PEPT7的N端和C端分别连接荧光素异硫氰酸酯(fluorescein isothiocyanate,FITC)基团,得到示踪剂T1和T2。为了确定适宜的示踪剂浓度,从100 nmol/L开始,对示踪剂进行了2倍梯度稀释,随后测定FP值。根据检测结果(图2A),T1的检测窗口较T2更宽,因此选择50 nmol/L的T1用于后续筛选。
图2
图2
FP筛选体系的建立
Note: A. FP curve of tracer T1 and T2 at different concentrations. B. FP curve of tracer T1 binding with SAE2 at different concentrations. lg [Tracer (nmol·L-1)] means the logarithm of the concentration of Tracer, where the unit of concentration of Tracer is nmol·L-1. The concentration of T1 is 50 nmol/L.
Fig 2
Establishment of FP screening system
为确定最适宜的SAE2蛋白浓度,固定示踪剂浓度为50 nmol/L,测定其与不同浓度SAE2产生的FP值。然而,根据检测结果(图2B),即使SAE2浓度达到200 μmol/L,FP值仍未达平台期(示踪剂与SAE2蛋白100%结合)。因此,无法从当前数据绘制完整的曲线来确定SAE2蛋白的适宜浓度(通常是示踪剂与蛋白结合率达80%时对应的蛋白浓度)。
2.4 SPR实验
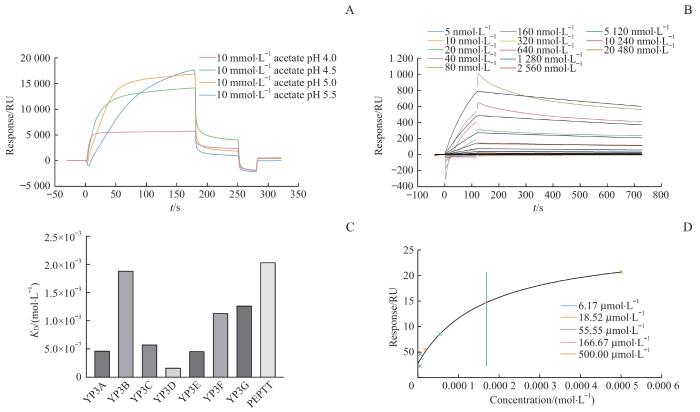
首先确定SAE2蛋白偶联的pH值(图3A)。根据一般要求,在引起较大共振单位(resonance unit,RU;满足实验需求即可)的前提下选择最温和的pH值。因此选择pH 5.0的10 mmol/L NaAc溶液稀释SAE2用于偶联。
图3
图3
SPR筛选体系的建立
Note: A. SPR pH scouting results for SAE2 immobilization. B. SPR sensorgrams of SAE1 binding to immobilized SAE2. C. Affinities of YP3 peptides for SAE2 determined by SPR. D. Affinity of YP3D for SAE2 determined by SPR equilibrium analysis.
Fig 3
Establishment of SPR screening system
接着,通过SPR检测SAE1与SAE2蛋白之间的亲和力,结果见图3B。动力学分析显示Kd值为1.13 μmol/L,与ITC实验中测得的SAE1与SAE2相互作用的Kd值0.96 μmol/L接近,表明SPR检测方法的可靠性。
2.5 基于SAE酶活的荧光实验
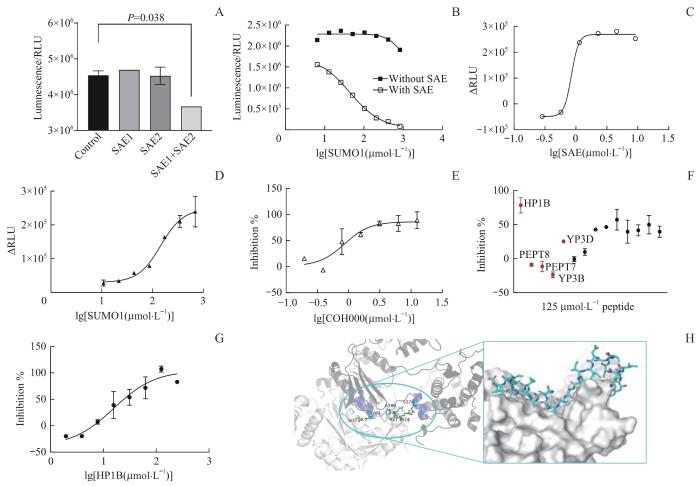
根据SAE的催化原理,拟开发一种通过ATP的消耗量检测完整的SAE1和SAE2复合物的腺苷化催化活性,从而间接反映SAE1与SAE2相互作用的强度。实验中,在酶活反应体系中加入SAE1和SAE2单体蛋白、SUMO1及ATP。反应完成后使用Kinase-Glo试剂盒检测体系中剩余的ATP量,相对发光单位(relative luminescence unit,RLU)读值与ATP浓度成正比。
图4
图4
基于SAE酶活的筛选体系的建立
Note: A. Effect of the absence of SAE1 or SAE2 on the adenylation efficiency of SAE. B. The adenylation efficiencies of SAE under different concentrations of SUMO1. C. Optimal concentration of SAE; ΔRLU=RLUcontrol-RLUsample. D. Optimal concentration of SUMO1. E. Inhibition curve of COH000. F. Inhibition rates of peptides at 125 μmol·L-1. G. Inhibition curve of HP1B. H. Binding interaction between HP1B (blue) and SAE1 (gray). Key interacting residues of interaction are shown as sticks and labeled. Yellow dashed lines represent hydrogen bonds.
Fig 4
Establishment of a SAE enzyme activity-based screening system
应用验证完毕的酶活体系,在125 μmol/L浓度下分别检测SPR实验确定的亲和力最强的多肽YP3D,以及亲和力最差的YP3B与PEPT7。结果显示,YP3D的抑制效果显著优于几乎没有活性的PEPT7和YP3B;此外,在ITC实验中表现放热微弱的PEPT8也未在这一酶活体系中表现出活性。上述结果与此前通过ITC及SPR得出的结论一致,再次证明该酶活体系的可靠性(图4F)。
3 讨论
PPI构成了蛋白质生理功能的重要一环,是治疗包括肿瘤在内的多种疾病的药物靶点。在靶向PPI的抑制剂开发上,对于复合物的共晶结构已经得到解析的靶点,通过截取相互作用界面的热点以设计肽抑制剂是一种广泛被采用的理性设计策略。然而,并非所有PPI相互作用界面的热点都是集中的,有相当一部分PPI的界面大(>2 000 Å2)而平坦、热点分散,且蛋白之间的亲和力较弱(KD>200 nmol/L)。针对这类PPI靶点进行理性设计的难度较高,目前鲜有报道[15]。例如,本研究中探讨的靶点SAE1与SAE2,其相互作用界面热点分散,且蛋白间的亲和力约为1 μmol/L。由于理性设计策略获得的候选多肽普遍具有较低亲和力,因此在筛选体系的建立上困难重重。
本研究首先尝试建立了多种基于亲和力的筛选体系。ITC是一种经典的通过精确测量结合反应产生或吸收的热量来检测相互作用的生物物理学方法,可提供相互作用的熵、焓、结合亲和力、化学计量等重要信息[16]。ITC可以在溶液条件下直接测得反应组分之间的结合能。但在检测低亲和力(中微摩尔范围)的相互作用时,该方法面临多重挑战,包括结合热微弱,难以准确测定,以及蛋白用量高等。此外,ITC实验对样品制备和操作具有极高的要求。滴定针和样品池中缓冲液成分稍有不一致就可能导致显著的溶解热干扰。且仪器清洗不当、样品脱气不充分或进样时不慎引入气泡都可能导致滴定曲线的较大波动,影响数据分析的准确性。在检测通量上,由于单个样品的检测时长可达数小时,因此,ITC也并不适用于较大规模的药物筛选。
FP是一种常用的检测溶液中分子相互作用的方法。其一般原理为:示踪剂是一种带荧光基团的可同靶标蛋白结合的配体。当在溶液中引入与示踪剂竞争性结合靶标蛋白的小分子时,小分子同靶标蛋白的亲和力越强,示踪剂同蛋白结合越少,进而对激发光的去偏振作用越弱,产生的发射光的FP值越高,从而实现对靶标的亲和配体的筛选[17]。FP是PPI抑制剂筛选中常见的高通量检测方法,具备方便、快速、灵敏和稳定等多种优势。然而,一个同靶标蛋白具有高亲和力的小分子配体对于构建FP筛选体系而言是十分必要的前提条件。这使得FP体系难以实现基于低亲和力候选分子的PPI抑制剂的筛选体系构建。
SPR可以实时检测生物分子之间的相互作用。检测时将发生相互作用的一方(配体)偶联固定在特制的芯片上,另一方(分析物)随缓冲液流经芯片表面;配体和分析物间的结合将引起传感器共振单位的数值变化,从而反映两者结合或解离的即时状态。通过对不同浓度的分析物加以动力学或稳态分析,可以获得相互作用的结合化学计量、可逆性、亲和力等关键数据。不同于FP需要一个已知高亲和力配体作为示踪剂,SPR并不存在这一限制,并且较ITC具有更高的分辨率和灵敏度,但其实验时间长、仪器和耗材十分昂贵,同样不能满足药物研发初期的筛选通量需求,更适用于对初筛得到的活性较好的分子进行精确的亲和力测定。
在靶向酶类蛋白抑制剂的开发上,除了采用ITC、FP、SPR等基于亲和力的筛选方法之外,构建基于酶本身活性的筛选方法也有许多成功的应用范例。值得一提的是,基于亲和力的筛选方法只能筛选出同靶标具有强亲和力的化合物,而强亲和力并不完全等价于良好的抑制活性。相比之下,基于酶活的筛选方法可以直接获得具有抑制活性的化合物,极大地提高药物筛选的效率。因此,本研究构建了一种基于SAE酶活的高通量筛选方法以促进多肽抑制剂的发现。SAE作为SUMO化通路唯一的激活酶,其催化过程主要分为2步:第一步,在由SAE1和SAE2共同构成的腺苷化活性位点上,消耗ATP完成对SUMO1蛋白C端的腺苷化,随后释放出PPi;第二步,位于SAE2上的催化半胱氨酸活性位点攻击腺苷酸化的SUMO,在173位半胱氨酸和SUMO之间形成硫酯键并释放出AMP。研究[18]表明,只有其中的第一步需要SAE1和SAE2的共同参与。目前已有的基于酶活的SUMO化通路关键酶抑制剂的筛选方法有ATP-PPi交换实验[19]、均相时间分辨荧光(homogeneous time-resolved fluorescence,HTRF)[8]等。经过一系列条件优化和验证,本研究最终确立了一种兼具高灵敏度、稳定性和可接受的检测成本的高通量筛选方法,且相较于基于亲和力的筛选方法,基于酶活的筛选可以直接获得具有抑制活性的化合物,因而更加高效。应用该方法,我们对一批截取自SAE1或SAE2上的相互作用热点片段的多肽进行了初筛,初筛结果与SPR的结论基本一致。测得其中活性最好的多肽HP1B的IC50值为15.72 μmol/L,并基于SAE1和SAE2共晶结构分析了HP1B同SAE1结合的可能模式,为后续多肽的改造提供了良好的设计起点。此外,由于该多肽分子量大且疏水性氨基酸比例高,溶解性较差,后续研究将聚焦于如何进一步优化以改良其成药性。
综上所述,本研究综合比较了多种筛选PPI抑制剂的体外方法,并最终建立了一种基于酶活的SAE1与SAE2相互作用的肽抑制剂的高通量筛选体系,为获得多肽抑制活性的重要数据、指导多肽的优化改造奠定了坚实的基础。作为一次针对亲和力弱、热点分散的PPI靶点开发抑制剂筛选体系的成功实践,本研究为类似靶点的筛选体系建立提供了有效参考。
作者贡献声明
杨秀岩负责课题设计,胡晨阳负责实验设计、实验操作、数据分析和论文撰写,杨秀岩和陆绍永参与论文的审阅与修改。所有作者均阅读并同意了最终稿件的提交。
AUTHOR's CONTRIBUTIONS
YANG Xiuyan was responsible for project design. HU Chenyang was responsible for experimental design and operation, data analysis and thesis writing. YANG Xiuyan and LU Shaoyong participated in the reviewing and revising. All the authors have read the last version of paper and consented for submission.
利益冲突声明
所有作者声明不存在利益冲突。
COMPETING INTERESTS
All authors disclose no relevant conflict of interests.
参考文献